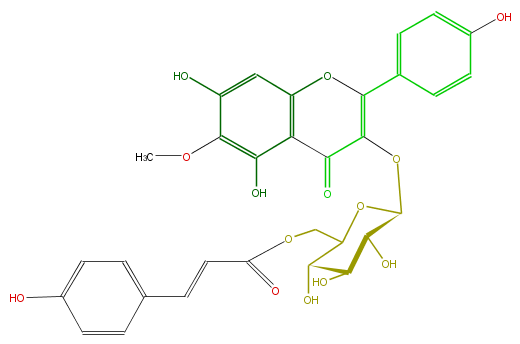
Mol:FL5FEAGS0022
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.7656 2.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7656 2.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7656 1.9596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7656 1.9596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4801 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4801 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1946 1.9596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1946 1.9596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1946 2.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1946 2.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4801 3.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4801 3.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0512 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0512 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3367 1.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3367 1.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6222 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6222 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6222 0.7221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6222 0.7221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3367 0.3096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3367 0.3096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0512 0.7221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0512 0.7221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0923 1.9596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0923 1.9596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8067 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8067 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8067 0.7221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8067 0.7221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0923 0.3096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0923 0.3096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3367 -0.4087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3367 -0.4087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5212 1.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5212 1.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7288 0.2873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7288 0.2873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7953 3.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7953 3.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0923 -0.3899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0923 -0.3899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4729 0.3375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4729 0.3375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2094 0.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2094 0.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9501 -1.8446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9501 -1.8446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7608 -1.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7608 -1.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1236 -1.2574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1236 -1.2574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8135 -0.8045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8135 -0.8045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0027 -0.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0027 -0.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6398 -1.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6398 -1.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0936 -1.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0936 -1.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5705 -1.3107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5705 -1.3107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9501 -2.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9501 -2.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2717 -2.1694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2717 -2.1694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4964 -1.8051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4964 -1.8051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2621 -1.7674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2621 -1.7674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3114 -2.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3114 -2.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0859 -1.7678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0859 -1.7678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4975 -2.4816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4975 -2.4816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3214 -2.4820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3214 -2.4820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7343 -1.7677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7343 -1.7677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5593 -1.7682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5593 -1.7682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9714 -2.4828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9714 -2.4828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5585 -3.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5585 -3.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7335 -3.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7335 -3.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7953 -2.4833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7953 -2.4833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 15 22 1 0 0 0 0 | + | 15 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | 42 45 1 0 0 0 0 | + | 42 45 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FEAGS0022 | + | ID FL5FEAGS0022 |
| − | KNApSAcK_ID C00013825 | + | KNApSAcK_ID C00013825 |
| − | NAME 6-Hydroxykaempferol 6-methyl ether 3-(6''-p-coumaroylglucoside);5,7-Dihydroxy-2-(4-hydroxyphenyl)-3-[[6-O-[(2E)-3-(4-hydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-6-methoxy-4H-1-benzopyran-4-one | + | NAME 6-Hydroxykaempferol 6-methyl ether 3-(6''-p-coumaroylglucoside);5,7-Dihydroxy-2-(4-hydroxyphenyl)-3-[[6-O-[(2E)-3-(4-hydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-6-methoxy-4H-1-benzopyran-4-one |
| − | CAS_RN 235741-38-5 | + | CAS_RN 235741-38-5 |
| − | FORMULA C31H28O14 | + | FORMULA C31H28O14 |
| − | EXACTMASS 624.147905604 | + | EXACTMASS 624.147905604 |
| − | AVERAGEMASS 624.5456200000001 | + | AVERAGEMASS 624.5456200000001 |
| − | SMILES C(c(c5)ccc(O)c5)=CC(OCC(C(O)4)OC(C(C4O)O)OC(C3=O)=C(Oc(c32)cc(O)c(c2O)OC)c(c1)ccc(O)c1)=O | + | SMILES C(c(c5)ccc(O)c5)=CC(OCC(C(O)4)OC(C(C4O)O)OC(C3=O)=C(Oc(c32)cc(O)c(c2O)OC)c(c1)ccc(O)c1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
2.7656 2.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7656 1.9596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4801 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1946 1.9596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1946 2.7846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4801 3.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0512 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3367 1.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6222 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6222 0.7221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3367 0.3096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0512 0.7221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0923 1.9596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8067 1.5471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8067 0.7221 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0923 0.3096 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3367 -0.4087 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5212 1.9596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7288 0.2873 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7953 3.1314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0923 -0.3899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4729 0.3375 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2094 0.3375 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9501 -1.8446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7608 -1.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1236 -1.2574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8135 -0.8045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0027 -0.6503 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6398 -1.3916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0936 -1.1281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5705 -1.3107 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9501 -2.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2717 -2.1694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4964 -1.8051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2621 -1.7674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3114 -2.3409 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0859 -1.7678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4975 -2.4816 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3214 -2.4820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7343 -1.7677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5593 -1.7682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9714 -2.4828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5585 -3.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7335 -3.1967 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7953 -2.4833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
15 22 1 0 0 0 0
22 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
35 36 2 0 0 0 0
35 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 43 1 0 0 0 0
43 44 2 0 0 0 0
44 39 1 0 0 0 0
42 45 1 0 0 0 0
31 35 1 0 0 0 0
27 19 1 0 0 0 0
S SKP 8
ID FL5FEAGS0022
KNApSAcK_ID C00013825
NAME 6-Hydroxykaempferol 6-methyl ether 3-(6''-p-coumaroylglucoside);5,7-Dihydroxy-2-(4-hydroxyphenyl)-3-[[6-O-[(2E)-3-(4-hydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-6-methoxy-4H-1-benzopyran-4-one
CAS_RN 235741-38-5
FORMULA C31H28O14
EXACTMASS 624.147905604
AVERAGEMASS 624.5456200000001
SMILES C(c(c5)ccc(O)c5)=CC(OCC(C(O)4)OC(C(C4O)O)OC(C3=O)=C(Oc(c32)cc(O)c(c2O)OC)c(c1)ccc(O)c1)=O
M END