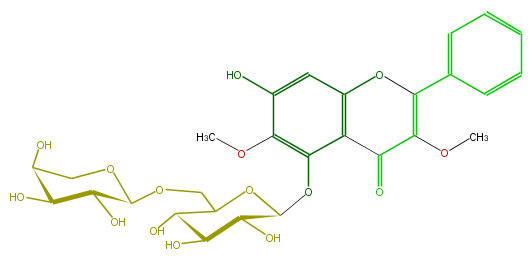
Mol:FL5FE9GS0001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 3.6253 1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6253 1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6253 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6253 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3397 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3397 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0542 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0542 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0542 1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0542 1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3397 2.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3397 2.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9108 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9108 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1963 1.1037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1963 1.1037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4818 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4818 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4818 -0.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4818 -0.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1963 -0.5463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1963 -0.5463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9108 -0.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9108 -0.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7673 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7673 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0528 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0528 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0528 -0.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0528 -0.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7673 -0.5463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7673 -0.5463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1963 -1.2646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1963 -1.2646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6616 1.1037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6616 1.1037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5171 -0.4839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5171 -0.4839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7673 -1.2663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7673 -1.2663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0955 -0.1499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0955 -0.1499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5769 -0.4974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5769 -0.4974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1892 -0.1438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1892 -0.1438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9390 -1.7101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9390 -1.7101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3181 -2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3181 -2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6301 -1.7982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6301 -1.7982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1939 -1.7572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1939 -1.7572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4269 -1.2138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4269 -1.2138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1150 -1.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1150 -1.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4024 -1.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4024 -1.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2405 -1.2253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2405 -1.2253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3615 -2.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3615 -2.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9060 -2.3413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9060 -2.3413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0278 -2.1924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0278 -2.1924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8273 -0.7736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8273 -0.7736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4148 -1.4880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4148 -1.4880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6166 -1.2789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6166 -1.2789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8234 -1.5057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8234 -1.5057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2358 -0.7912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2358 -0.7912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0340 -1.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0340 -1.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6593 -0.3125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6593 -0.3125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0542 -1.3583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0542 -1.3583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1474 -1.8662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1474 -1.8662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 19 21 1 0 0 0 0 | + | 19 21 1 0 0 0 0 |
| − | 15 22 1 0 0 0 0 | + | 15 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 20 1 0 0 0 0 | + | 27 20 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 31 1 0 0 0 0 | + | 38 31 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FE9GS0001 | + | ID FL5FE9GS0001 |
| − | FORMULA C28H32O15 | + | FORMULA C28H32O15 |
| − | EXACTMASS 608.174120354 | + | EXACTMASS 608.174120354 |
| − | AVERAGEMASS 608.54468 | + | AVERAGEMASS 608.54468 |
| − | SMILES c(c34)(C(=O)C(OC)=C(c(c5)cccc5)O4)c(c(OC)c(c3)O)OC(C(O)2)OC(C(C(O)2)O)COC(O1)C(O)C(O)C(C1)O | + | SMILES c(c34)(C(=O)C(OC)=C(c(c5)cccc5)O4)c(c(OC)c(c3)O)OC(C(O)2)OC(C(C(O)2)O)COC(O1)C(O)C(O)C(C1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
3.6253 1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6253 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3397 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0542 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0542 1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3397 2.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9108 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1963 1.1037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4818 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4818 -0.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1963 -0.5463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9108 -0.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7673 1.1037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0528 0.6912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0528 -0.1338 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7673 -0.5463 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1963 -1.2646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6616 1.1037 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5171 -0.4839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7673 -1.2663 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0955 -0.1499 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5769 -0.4974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1892 -0.1438 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9390 -1.7101 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3181 -2.2534 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6301 -1.7982 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1939 -1.7572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4269 -1.2138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1150 -1.6689 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4024 -1.2933 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2405 -1.2253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3615 -2.0577 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9060 -2.3413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0278 -2.1924 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8273 -0.7736 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4148 -1.4880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6166 -1.2789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8234 -1.5057 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2358 -0.7912 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0340 -1.0002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6593 -0.3125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0542 -1.3583 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1474 -1.8662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
19 21 1 0 0 0 0
15 22 1 0 0 0 0
22 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
27 20 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
35 41 1 0 0 0 0
36 42 1 0 0 0 0
37 43 1 0 0 0 0
38 31 1 0 0 0 0
S SKP 5
ID FL5FE9GS0001
FORMULA C28H32O15
EXACTMASS 608.174120354
AVERAGEMASS 608.54468
SMILES c(c34)(C(=O)C(OC)=C(c(c5)cccc5)O4)c(c(OC)c(c3)O)OC(C(O)2)OC(C(C(O)2)O)COC(O1)C(O)C(O)C(C1)O
M END