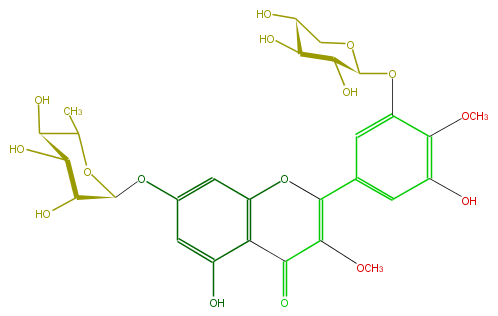
Mol:FL5FDJGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.7701 -0.8275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7701 -0.8275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7701 -1.6529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7701 -1.6529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0552 -2.0656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0552 -2.0656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3405 -1.6529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3405 -1.6529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3405 -0.8275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3405 -0.8275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0552 -0.4149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0552 -0.4149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3743 -2.0656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3743 -2.0656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0890 -1.6529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0890 -1.6529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0890 -0.8275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0890 -0.8275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3743 -0.4149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3743 -0.4149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3743 -2.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3743 -2.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8036 -0.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8036 -0.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5321 -0.8356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5321 -0.8356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2606 -0.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2606 -0.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2606 0.4262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2606 0.4262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5321 0.8469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5321 0.8469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8036 0.4262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8036 0.4262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4846 -0.4150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4846 -0.4150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5321 1.6878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5321 1.6878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0552 -2.8906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0552 -2.8906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5915 2.7832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5915 2.7832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6801 2.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6801 2.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3371 1.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3371 1.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8582 1.6817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8582 1.6817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6993 2.2753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6993 2.2753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0867 2.2985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0867 2.2985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0234 2.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0234 2.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0856 2.3857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0856 2.3857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6163 1.3418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6163 1.3418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7646 0.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7646 0.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5333 0.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5333 0.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9808 -0.0459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9808 -0.0459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7919 -0.7955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7919 -0.7955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0232 -0.7955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0232 -0.7955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5756 -0.2610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5756 -0.2610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9069 0.2022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9069 0.2022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5568 1.1679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5568 1.1679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9837 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9837 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3832 -1.1015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3832 -1.1015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9889 -0.8355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9889 -0.8355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9889 0.8468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9889 0.8468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9069 0.3167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9069 0.3167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8952 -2.1817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8952 -2.1817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9379 -1.9910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9379 -1.9910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 30 38 1 0 0 0 0 | + | 30 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 18 34 1 0 0 0 0 | + | 18 34 1 0 0 0 0 |
| − | 14 40 1 0 0 0 0 | + | 14 40 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 15 41 1 0 0 0 0 | + | 15 41 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 8 43 1 0 0 0 0 | + | 8 43 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 46 -0.7283 -0.4206 | + | M SBV 1 46 -0.7283 -0.4206 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 43 44 | + | M SAL 2 2 43 44 |
| − | M SBL 2 1 48 | + | M SBL 2 1 48 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 48 -0.8062 0.5289 | + | M SBV 2 48 -0.8062 0.5289 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FDJGS0002 | + | ID FL5FDJGS0002 |
| − | FORMULA C28H32O16 | + | FORMULA C28H32O16 |
| − | EXACTMASS 624.1690349759999 | + | EXACTMASS 624.1690349759999 |
| − | AVERAGEMASS 624.54408 | + | AVERAGEMASS 624.54408 |
| − | SMILES OC(C5O)C(OC(C5O)C)Oc(c4)cc(c1c4O)OC(c(c2)cc(OC(O3)C(O)C(O)C(C3)O)c(OC)c2O)=C(C1=O)OC | + | SMILES OC(C5O)C(OC(C5O)C)Oc(c4)cc(c1c4O)OC(c(c2)cc(OC(O3)C(O)C(O)C(C3)O)c(OC)c2O)=C(C1=O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-1.7701 -0.8275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7701 -1.6529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0552 -2.0656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3405 -1.6529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3405 -0.8275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0552 -0.4149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3743 -2.0656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0890 -1.6529 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0890 -0.8275 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3743 -0.4149 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3743 -2.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8036 -0.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5321 -0.8356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2606 -0.4150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2606 0.4262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5321 0.8469 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8036 0.4262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4846 -0.4150 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5321 1.6878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0552 -2.8906 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5915 2.7832 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6801 2.0761 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3371 1.9750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8582 1.6817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6993 2.2753 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0867 2.2985 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0234 2.8945 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0856 2.3857 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6163 1.3418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7646 0.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5333 0.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9808 -0.0459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7919 -0.7955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0232 -0.7955 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5756 -0.2610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9069 0.2022 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5568 1.1679 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9837 0.9501 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3832 -1.1015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9889 -0.8355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9889 0.8468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.9069 0.3167 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8952 -2.1817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9379 -1.9910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
16 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
19 24 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
30 38 1 0 0 0 0
33 39 1 0 0 0 0
18 34 1 0 0 0 0
14 40 1 0 0 0 0
41 42 1 0 0 0 0
15 41 1 0 0 0 0
43 44 1 0 0 0 0
8 43 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 OCH3
M SBV 1 46 -0.7283 -0.4206
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 43 44
M SBL 2 1 48
M SMT 2 OCH3
M SBV 2 48 -0.8062 0.5289
S SKP 5
ID FL5FDJGS0002
FORMULA C28H32O16
EXACTMASS 624.1690349759999
AVERAGEMASS 624.54408
SMILES OC(C5O)C(OC(C5O)C)Oc(c4)cc(c1c4O)OC(c(c2)cc(OC(O3)C(O)C(O)C(C3)O)c(OC)c2O)=C(C1=O)OC
M END