Mol:FL5FCCGSS001
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 47 50 0 0 0 0 0 0 0 0999 V2000 | + | 47 50 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1279 -1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1279 -1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1279 -1.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1279 -1.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3895 -2.4099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3895 -2.4099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6510 -1.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6510 -1.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6510 -1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6510 -1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3895 -0.7045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3895 -0.7045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9126 -2.4099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9126 -2.4099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1741 -1.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1741 -1.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1741 -1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1741 -1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9126 -0.7045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9126 -0.7045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9126 -3.1033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9126 -3.1033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6114 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6114 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3639 -1.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3639 -1.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1166 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1166 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1166 0.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1166 0.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3639 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3639 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6114 0.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6114 0.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2696 -2.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2696 -2.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4743 -2.4669 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4743 -2.4669 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4743 -1.7611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4743 -1.7611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6811 -2.4630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6811 -2.4630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4743 -3.1849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4743 -3.1849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0295 3.3398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0295 3.3398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3464 2.5880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3464 2.5880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2472 2.1114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2472 2.1114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5960 1.5222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5960 1.5222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7782 2.2018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7782 2.2018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1841 2.5756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1841 2.5756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6263 3.7960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6263 3.7960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6954 3.2193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6954 3.2193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1510 1.2710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1510 1.2710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3617 1.3733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3617 1.3733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5198 0.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5198 0.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7268 0.5901 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7268 0.5901 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7268 1.2300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7268 1.2300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9392 0.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9392 0.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7268 -0.1401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7268 -0.1401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3919 -4.6313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3919 -4.6313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3919 -3.9105 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3919 -3.9105 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6494 -3.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6494 -3.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3921 -3.1230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3921 -3.1230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1140 -3.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1140 -3.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8562 -0.7104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8562 -0.7104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5198 0.4393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5198 0.4393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4404 3.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4404 3.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1041 2.3322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1041 2.3322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1041 4.6313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1041 4.6313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 18 19 1 0 0 0 0 | + | 18 19 1 0 0 0 0 |
| − | 19 20 2 0 0 0 0 | + | 19 20 2 0 0 0 0 |
| − | 19 21 1 0 0 0 0 | + | 19 21 1 0 0 0 0 |
| − | 19 22 2 0 0 0 0 | + | 19 22 2 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 16 32 1 0 0 0 0 | + | 16 32 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 34 37 2 0 0 0 0 | + | 34 37 2 0 0 0 0 |
| − | 15 36 1 0 0 0 0 | + | 15 36 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 39 41 1 0 0 0 0 | + | 39 41 1 0 0 0 0 |
| − | 39 42 2 0 0 0 0 | + | 39 42 2 0 0 0 0 |
| − | 41 3 1 0 0 0 0 | + | 41 3 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 1 43 1 0 0 0 0 | + | 1 43 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 28 45 1 0 0 0 0 | + | 28 45 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 43 44 | + | M SAL 1 2 43 44 |
| − | M SBL 1 1 47 | + | M SBL 1 1 47 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 47 0.7283 -0.4205 | + | M SBV 1 47 0.7283 -0.4205 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 3 45 46 47 | + | M SAL 2 3 45 46 47 |
| − | M SBL 2 1 50 | + | M SBL 2 1 50 |
| − | M SMT 2 COOH | + | M SMT 2 COOH |
| − | M SBV 2 50 -0.2563 -0.9061 | + | M SBV 2 50 -0.2563 -0.9061 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FCCGSS001 | + | ID FL5FCCGSS001 |
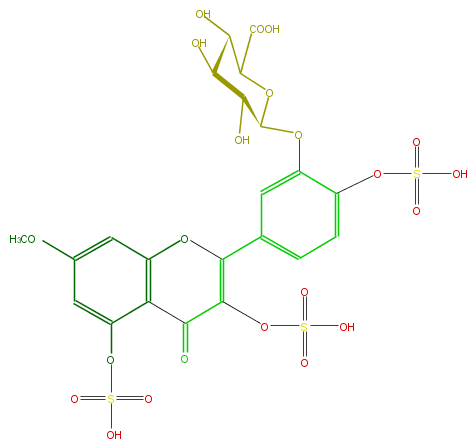
| − | FORMULA C22H20O22S3 | + | FORMULA C22H20O22S3 |
| − | EXACTMASS 731.960834394 | + | EXACTMASS 731.960834394 |
| − | AVERAGEMASS 732.5790000000001 | + | AVERAGEMASS 732.5790000000001 |
| − | SMILES C(c43)(C(OS(O)(=O)=O)=C(Oc3cc(OC)cc4OS(O)(=O)=O)c(c1)ccc(c(OC(O2)C(O)C(C(O)C2C(O)=O)O)1)OS(O)(=O)=O)=O | + | SMILES C(c43)(C(OS(O)(=O)=O)=C(Oc3cc(OC)cc4OS(O)(=O)=O)c(c1)ccc(c(OC(O2)C(O)C(C(O)C2C(O)=O)O)1)OS(O)(=O)=O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
47 50 0 0 0 0 0 0 0 0999 V2000
-3.1279 -1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1279 -1.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3895 -2.4099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6510 -1.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6510 -1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3895 -0.7045 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9126 -2.4099 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1741 -1.9835 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1741 -1.1309 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9126 -0.7045 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9126 -3.1033 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6114 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3639 -1.1193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1166 -0.6848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1166 0.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3639 0.6188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6114 0.1842 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2696 -2.4669 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4743 -2.4669 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
1.4743 -1.7611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6811 -2.4630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4743 -3.1849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0295 3.3398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3464 2.5880 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2472 2.1114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5960 1.5222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7782 2.2018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1841 2.5756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6263 3.7960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6954 3.2193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1510 1.2710 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3617 1.3733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5198 0.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7268 0.5901 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
3.7268 1.2300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9392 0.5901 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7268 -0.1401 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3919 -4.6313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3919 -3.9105 0.0000 S 0 0 0 0 0 0 0 0 0 0 0 0
-1.6494 -3.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3921 -3.1230 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1140 -3.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8562 -0.7104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5198 0.4393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4404 3.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1041 2.3322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1041 4.6313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
4 3 1 0 0 0 0
18 19 1 0 0 0 0
19 20 2 0 0 0 0
19 21 1 0 0 0 0
19 22 2 0 0 0 0
21 8 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 32 1 0 0 0 0
16 32 1 0 0 0 0
33 34 1 0 0 0 0
34 35 2 0 0 0 0
34 36 1 0 0 0 0
34 37 2 0 0 0 0
15 36 1 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
39 41 1 0 0 0 0
39 42 2 0 0 0 0
41 3 1 0 0 0 0
43 44 1 0 0 0 0
1 43 1 0 0 0 0
45 46 2 0 0 0 0
45 47 1 0 0 0 0
28 45 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 43 44
M SBL 1 1 47
M SMT 1 ^ OCH3
M SBV 1 47 0.7283 -0.4205
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 3 45 46 47
M SBL 2 1 50
M SMT 2 COOH
M SBV 2 50 -0.2563 -0.9061
S SKP 5
ID FL5FCCGSS001
FORMULA C22H20O22S3
EXACTMASS 731.960834394
AVERAGEMASS 732.5790000000001
SMILES C(c43)(C(OS(O)(=O)=O)=C(Oc3cc(OC)cc4OS(O)(=O)=O)c(c1)ccc(c(OC(O2)C(O)C(C(O)C2C(O)=O)O)1)OS(O)(=O)=O)=O
M END