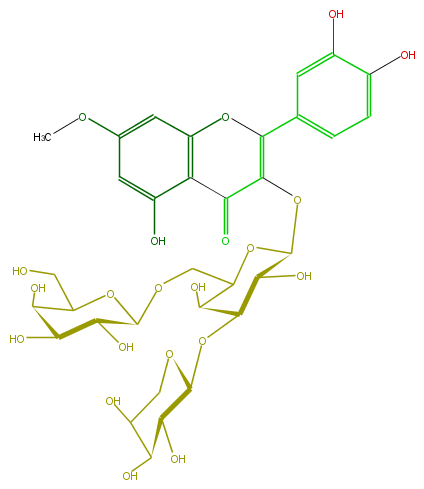
Mol:FL5FCCGS0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.4099 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4099 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6954 2.5591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6954 2.5591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6954 3.3841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6954 3.3841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4099 3.7965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4099 3.7965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1244 3.3841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1244 3.3841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1244 2.5591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1244 2.5591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9811 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9811 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2666 2.5591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2666 2.5591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4479 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4479 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4479 1.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4479 1.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2666 0.9091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2666 0.9091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9811 1.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9811 1.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1623 2.5591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1623 2.5591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8768 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8768 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8768 1.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8768 1.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1623 0.9091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1623 0.9091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2666 0.0842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2666 0.0842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5912 2.5591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5912 2.5591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6954 0.9091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6954 0.9091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1623 0.0842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1623 0.0842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8389 3.7965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8389 3.7965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4099 4.6215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4099 4.6215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2873 2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2873 2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2759 -1.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2759 -1.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5370 -1.4073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5370 -1.4073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8885 -0.6605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8885 -0.6605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5713 -0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5713 -0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7582 -0.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7582 -0.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4068 -0.8021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4068 -0.8021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3997 -0.4829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3997 -0.4829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0772 -0.8493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0772 -0.8493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3601 -0.8456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3601 -0.8456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1957 -1.9226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1957 -1.9226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7632 -0.6162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7632 -0.6162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6182 -1.2397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6182 -1.2397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0877 -1.8719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0877 -1.8719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3379 -1.5272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3379 -1.5272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5171 -1.6129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5171 -1.6129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0474 -0.9806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0474 -0.9806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7974 -1.3251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7974 -1.3251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1678 -0.6001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1678 -0.6001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7851 -0.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7851 -0.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5542 -0.8572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5542 -0.8572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8389 -1.8749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8389 -1.8749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8023 -2.0353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8023 -2.0353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6467 -3.5611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6467 -3.5611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2384 -4.2784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2384 -4.2784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0331 -3.4790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0331 -3.4790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4492 -2.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4492 -2.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8573 -2.1787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8573 -2.1787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0627 -2.9780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0627 -2.9780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0535 -3.1283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0535 -3.1283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7287 -4.6215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7287 -4.6215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7652 -4.2807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7652 -4.2807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 18 23 1 0 0 0 0 | + | 18 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 45 1 0 0 0 0 | + | 37 45 1 0 0 0 0 |
| − | 38 31 1 0 0 0 0 | + | 38 31 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 1 0 0 0 | + | 47 48 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 50 49 1 1 0 0 0 | + | 50 49 1 1 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 46 1 0 0 0 0 | + | 51 46 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 47 53 1 0 0 0 0 | + | 47 53 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 49 33 1 0 0 0 0 | + | 49 33 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FCCGS0011 | + | ID FL5FCCGS0011 |
| − | FORMULA C33H40O21 | + | FORMULA C33H40O21 |
| − | EXACTMASS 772.206208342 | + | EXACTMASS 772.206208342 |
| − | AVERAGEMASS 772.6581 | + | AVERAGEMASS 772.6581 |
| − | SMILES C(O)(C1OC(C3O)C(O)C(OC3OC(=C5c(c6)ccc(O)c6O)C(c(c4O)c(O5)cc(OC)c4)=O)COC(O2)C(O)C(C(C2CO)O)O)C(C(CO1)O)O | + | SMILES C(O)(C1OC(C3O)C(O)C(OC3OC(=C5c(c6)ccc(O)c6O)C(c(c4O)c(O5)cc(OC)c4)=O)COC(O2)C(O)C(C(C2CO)O)O)C(C(CO1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
2.4099 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6954 2.5591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6954 3.3841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4099 3.7965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1244 3.3841 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1244 2.5591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9811 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2666 2.5591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4479 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4479 1.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2666 0.9091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9811 1.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1623 2.5591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8768 2.1466 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8768 1.3216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1623 0.9091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2666 0.0842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5912 2.5591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6954 0.9091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1623 0.0842 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8389 3.7965 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4099 4.6215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2873 2.1572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2759 -1.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5370 -1.4073 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8885 -0.6605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5713 -0.1971 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7582 -0.0553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4068 -0.8021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3997 -0.4829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0772 -0.8493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3601 -0.8456 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1957 -1.9226 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7632 -0.6162 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6182 -1.2397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0877 -1.8719 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3379 -1.5272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5171 -1.6129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0474 -0.9806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7974 -1.3251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1678 -0.6001 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7851 -0.5185 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5542 -0.8572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8389 -1.8749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8023 -2.0353 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6467 -3.5611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2384 -4.2784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0331 -3.4790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4492 -2.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8573 -2.1787 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0627 -2.9780 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0535 -3.1283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7287 -4.6215 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7652 -4.2807 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
18 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
27 19 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
37 45 1 0 0 0 0
38 31 1 0 0 0 0
46 47 1 0 0 0 0
47 48 1 1 0 0 0
49 48 1 1 0 0 0
50 49 1 1 0 0 0
50 51 1 0 0 0 0
51 46 1 0 0 0 0
46 52 1 0 0 0 0
47 53 1 0 0 0 0
48 54 1 0 0 0 0
49 33 1 0 0 0 0
S SKP 5
ID FL5FCCGS0011
FORMULA C33H40O21
EXACTMASS 772.206208342
AVERAGEMASS 772.6581
SMILES C(O)(C1OC(C3O)C(O)C(OC3OC(=C5c(c6)ccc(O)c6O)C(c(c4O)c(O5)cc(OC)c4)=O)COC(O2)C(O)C(C(C2CO)O)O)C(C(CO1)O)O
M END