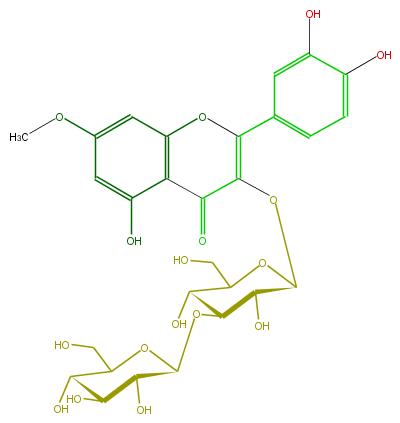
Mol:FL5FCCGS0009
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.1342 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1342 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4198 1.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4198 1.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4198 2.7260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4198 2.7260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1342 3.1385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1342 3.1385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8487 2.7260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8487 2.7260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8487 1.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8487 1.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7053 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7053 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0092 1.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0092 1.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7237 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7237 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7237 0.6635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7237 0.6635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0092 0.2510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0092 0.2510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7053 0.6635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7053 0.6635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4381 1.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4381 1.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1526 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1526 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1526 0.6635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1526 0.6635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4381 0.2510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4381 0.2510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0092 -0.5740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0092 -0.5740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8671 1.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8671 1.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4198 0.2510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4198 0.2510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4381 -0.5740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4381 -0.5740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5632 3.1385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5632 3.1385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1342 3.9635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1342 3.9635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5632 1.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5632 1.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2726 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2726 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3805 -2.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3805 -2.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0396 -1.6180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0396 -1.6180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8598 -1.5265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8598 -1.5265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2067 -1.0220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2067 -1.0220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5475 -1.5186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5475 -1.5186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1798 -1.0363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1798 -1.0363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3693 -0.9622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3693 -0.9622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5361 -2.2407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5361 -2.2407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1329 -2.0454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1329 -2.0454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1240 -2.2751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1240 -2.2751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6473 -3.2986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6473 -3.2986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9942 -3.8031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9942 -3.8031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3351 -3.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3351 -3.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5149 -3.2149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5149 -3.2149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1680 -2.7104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1680 -2.7104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8272 -3.2070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8272 -3.2070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1949 -2.7247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1949 -2.7247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7440 -2.6506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7440 -2.6506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9108 -3.9291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9108 -3.9291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5076 -3.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5076 -3.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2507 -3.9635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2507 -3.9635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 18 23 1 0 0 0 0 | + | 18 23 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 45 1 0 0 0 0 | + | 37 45 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FCCGS0009 | + | ID FL5FCCGS0009 |
| − | KNApSAcK_ID C00013905 | + | KNApSAcK_ID C00013905 |
| − | NAME Quercetin 7-methyl ether 3-laminaribioside;Rhamnetin 3-laminaribioside;2-(3,4-Dihydroxyphenyl)-3-[(3-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-7-methoxy-4H-1-benzopyran-4-one | + | NAME Quercetin 7-methyl ether 3-laminaribioside;Rhamnetin 3-laminaribioside;2-(3,4-Dihydroxyphenyl)-3-[(3-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-7-methoxy-4H-1-benzopyran-4-one |
| − | CAS_RN 194934-83-3 | + | CAS_RN 194934-83-3 |
| − | FORMULA C28H32O17 | + | FORMULA C28H32O17 |
| − | EXACTMASS 640.163949598 | + | EXACTMASS 640.163949598 |
| − | AVERAGEMASS 640.54348 | + | AVERAGEMASS 640.54348 |
| − | SMILES C(O)C(O1)C(O)C(OC(C5O)OC(C(C(O)5)O)CO)C(O)C1OC(=C3c(c4)cc(c(c4)O)O)C(c(c(O3)2)c(cc(OC)c2)O)=O | + | SMILES C(O)C(O1)C(O)C(OC(C5O)OC(C(C(O)5)O)CO)C(O)C1OC(=C3c(c4)cc(c(c4)O)O)C(c(c(O3)2)c(cc(OC)c2)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
2.1342 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4198 1.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4198 2.7260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1342 3.1385 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8487 2.7260 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8487 1.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7053 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0092 1.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7237 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7237 0.6635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0092 0.2510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7053 0.6635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4381 1.9010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1526 1.4885 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1526 0.6635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4381 0.2510 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0092 -0.5740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8671 1.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4198 0.2510 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4381 -0.5740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5632 3.1385 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1342 3.9635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5632 1.4991 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2726 -1.6102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3805 -2.1147 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0396 -1.6180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8598 -1.5265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2067 -1.0220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5475 -1.5186 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1798 -1.0363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3693 -0.9622 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5361 -2.2407 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1329 -2.0454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1240 -2.2751 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6473 -3.2986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9942 -3.8031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3351 -3.3064 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5149 -3.2149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1680 -2.7104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8272 -3.2070 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1949 -2.7247 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7440 -2.6506 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9108 -3.9291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5076 -3.7338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2507 -3.9635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
18 23 1 0 0 0 0
24 25 1 1 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 34 1 0 0 0 0
35 36 1 1 0 0 0
36 37 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
40 35 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
37 45 1 0 0 0 0
38 33 1 0 0 0 0
27 19 1 0 0 0 0
S SKP 8
ID FL5FCCGS0009
KNApSAcK_ID C00013905
NAME Quercetin 7-methyl ether 3-laminaribioside;Rhamnetin 3-laminaribioside;2-(3,4-Dihydroxyphenyl)-3-[(3-O-beta-D-glucopyranosyl-beta-D-glucopyranosyl)oxy]-5-hydroxy-7-methoxy-4H-1-benzopyran-4-one
CAS_RN 194934-83-3
FORMULA C28H32O17
EXACTMASS 640.163949598
AVERAGEMASS 640.54348
SMILES C(O)C(O1)C(O)C(OC(C5O)OC(C(C(O)5)O)CO)C(O)C1OC(=C3c(c4)cc(c(c4)O)O)C(c(c(O3)2)c(cc(OC)c2)O)=O
M END