Mol:FL5FCAGS0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 61 67 0 0 0 0 0 0 0 0999 V2000 | + | 61 67 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.4949 4.3422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4949 4.3422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4949 3.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4949 3.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2093 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2093 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9237 3.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9237 3.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9237 4.3422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9237 4.3422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2093 4.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2093 4.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7804 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7804 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0660 3.5172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0660 3.5172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6485 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6485 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6485 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6485 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0660 1.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0660 1.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7804 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7804 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3630 3.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3630 3.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0774 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0774 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0774 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0774 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3630 1.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3630 1.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0660 1.1246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0660 1.1246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7069 1.7641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7069 1.7641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5245 4.6891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5245 4.6891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3630 1.1678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3630 1.1678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1990 -0.6248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1990 -0.6248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0192 -0.5336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0192 -0.5336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1479 0.2813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1479 0.2813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6740 0.9171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6740 0.9171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8538 0.8260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8538 0.8260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7249 0.0109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7249 0.0109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0982 -0.0097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0982 -0.0097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4587 -0.5666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4587 -0.5666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1990 -1.3737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1990 -1.3737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7198 -1.0523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7198 -1.0523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0575 0.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0575 0.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8458 -1.6598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8458 -1.6598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1021 -2.4442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1021 -2.4442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3724 -2.0586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3724 -2.0586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5481 -2.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5481 -2.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2112 -1.2678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2112 -1.2678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0216 -1.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0216 -1.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3813 -2.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3813 -2.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8029 -2.8041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8029 -2.8041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6335 -2.3018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6335 -2.3018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5543 -1.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5543 -1.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9202 -2.2459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9202 -2.2459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2264 -2.7762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2264 -2.7762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3084 -2.2916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3084 -2.2916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5007 -1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5007 -1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1744 -1.2639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1744 -1.2639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4370 -2.0740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4370 -2.0740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9931 -1.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9931 -1.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1969 -1.4343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1969 -1.4343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6963 -1.7834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6963 -1.7834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8559 -4.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8559 -4.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8559 -4.7536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8559 -4.7536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3280 -4.7547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3280 -4.7547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6959 -3.8030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6959 -3.8030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2213 -3.8031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2213 -3.8031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3280 -4.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3280 -4.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2621 -3.5443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2621 -3.5443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7918 3.5172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7918 3.5172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5245 3.0942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5245 3.0942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8559 -3.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8559 -3.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3501 -2.8565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3501 -2.8565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 18 1 0 0 0 0 | + | 12 18 1 0 0 0 0 |
| − | 5 19 1 0 0 0 0 | + | 5 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 35 34 1 1 0 0 0 | + | 35 34 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 34 39 1 0 0 0 0 | + | 34 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 36 28 1 0 0 0 0 | + | 36 28 1 0 0 0 0 |
| − | 24 18 1 0 0 0 0 | + | 24 18 1 0 0 0 0 |
| − | 48 42 1 1 0 0 0 | + | 48 42 1 1 0 0 0 |
| − | 47 42 1 1 0 0 0 | + | 47 42 1 1 0 0 0 |
| − | 46 48 1 1 0 0 0 | + | 46 48 1 1 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 48 44 1 0 0 0 0 | + | 48 44 1 0 0 0 0 |
| − | 49 46 1 0 0 0 0 | + | 49 46 1 0 0 0 0 |
| − | 45 50 1 0 0 0 0 | + | 45 50 1 0 0 0 0 |
| − | 47 49 1 0 0 0 0 | + | 47 49 1 0 0 0 0 |
| − | 42 45 1 0 0 0 0 | + | 42 45 1 0 0 0 0 |
| − | 31 46 1 0 0 0 0 | + | 31 46 1 0 0 0 0 |
| − | 56 51 1 1 0 0 0 | + | 56 51 1 1 0 0 0 |
| − | 55 51 1 1 0 0 0 | + | 55 51 1 1 0 0 0 |
| − | 54 56 1 1 0 0 0 | + | 54 56 1 1 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 56 53 1 0 0 0 0 | + | 56 53 1 0 0 0 0 |
| − | 57 54 1 0 0 0 0 | + | 57 54 1 0 0 0 0 |
| − | 55 57 1 0 0 0 0 | + | 55 57 1 0 0 0 0 |
| − | 50 54 1 0 0 0 0 | + | 50 54 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 14 58 1 0 0 0 0 | + | 14 58 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 51 60 1 0 0 0 0 | + | 51 60 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 58 59 | + | M SAL 1 2 58 59 |
| − | M SBL 1 1 65 | + | M SBL 1 1 65 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 65 0.7144 -0.4125 | + | M SBV 1 65 0.7144 -0.4125 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 60 61 | + | M SAL 2 2 60 61 |
| − | M SBL 2 1 67 | + | M SBL 2 1 67 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 67 0.0000 -1.0213 | + | M SBV 2 67 0.0000 -1.0213 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FCAGS0012 | + | ID FL5FCAGS0012 |
| − | FORMULA C38H48O23 | + | FORMULA C38H48O23 |
| − | EXACTMASS 872.258637842 | + | EXACTMASS 872.258637842 |
| − | AVERAGEMASS 872.7739200000001 | + | AVERAGEMASS 872.7739200000001 |
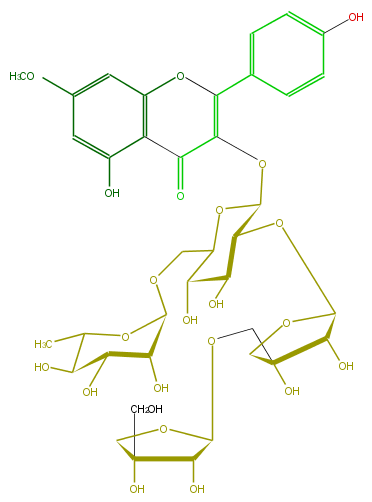
| − | SMILES Oc(c1)c(C(=O)2)c(OC(c(c7)ccc(c7)O)=C2OC(C(OC(O6)C(O)C(C6)(O)COC(C5O)OCC(O)(CO)5)4)OC(C(C4O)O)COC(C3O)OC(C(C(O)3)O)C)cc(OC)1 | + | SMILES Oc(c1)c(C(=O)2)c(OC(c(c7)ccc(c7)O)=C2OC(C(OC(O6)C(O)C(C6)(O)COC(C5O)OCC(O)(CO)5)4)OC(C(C4O)O)COC(C3O)OC(C(C(O)3)O)C)cc(OC)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
61 67 0 0 0 0 0 0 0 0999 V2000
1.4949 4.3422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4949 3.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2093 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9237 3.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9237 4.3422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2093 4.7547 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7804 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0660 3.5172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6485 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6485 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0660 1.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7804 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3630 3.5172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0774 3.1047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0774 2.2797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3630 1.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0660 1.1246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7069 1.7641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5245 4.6891 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3630 1.1678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1990 -0.6248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0192 -0.5336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1479 0.2813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6740 0.9171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8538 0.8260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7249 0.0109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0982 -0.0097 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4587 -0.5666 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1990 -1.3737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7198 -1.0523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0575 0.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8458 -1.6598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1021 -2.4442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3724 -2.0586 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5481 -2.0986 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2112 -1.2678 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0216 -1.6997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3813 -2.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8029 -2.8041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6335 -2.3018 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5543 -1.8496 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9202 -2.2459 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2264 -2.7762 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3084 -2.2916 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5007 -1.5566 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1744 -1.2639 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4370 -2.0740 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9931 -1.7456 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1969 -1.4343 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6963 -1.7834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8559 -4.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8559 -4.7536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3280 -4.7547 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6959 -3.8030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2213 -3.8031 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3280 -4.1630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2621 -3.5443 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7918 3.5172 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5245 3.0942 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8559 -3.1418 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3501 -2.8565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
11 17 2 0 0 0 0
12 18 1 0 0 0 0
5 19 1 0 0 0 0
16 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
21 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
35 34 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
35 38 1 0 0 0 0
34 39 1 0 0 0 0
33 40 1 0 0 0 0
32 41 1 0 0 0 0
36 28 1 0 0 0 0
24 18 1 0 0 0 0
48 42 1 1 0 0 0
47 42 1 1 0 0 0
46 48 1 1 0 0 0
42 43 1 0 0 0 0
48 44 1 0 0 0 0
49 46 1 0 0 0 0
45 50 1 0 0 0 0
47 49 1 0 0 0 0
42 45 1 0 0 0 0
31 46 1 0 0 0 0
56 51 1 1 0 0 0
55 51 1 1 0 0 0
54 56 1 1 0 0 0
51 52 1 0 0 0 0
56 53 1 0 0 0 0
57 54 1 0 0 0 0
55 57 1 0 0 0 0
50 54 1 0 0 0 0
58 59 1 0 0 0 0
14 58 1 0 0 0 0
60 61 1 0 0 0 0
51 60 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 58 59
M SBL 1 1 65
M SMT 1 ^ OCH3
M SBV 1 65 0.7144 -0.4125
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 60 61
M SBL 2 1 67
M SMT 2 CH2OH
M SBV 2 67 0.0000 -1.0213
S SKP 5
ID FL5FCAGS0012
FORMULA C38H48O23
EXACTMASS 872.258637842
AVERAGEMASS 872.7739200000001
SMILES Oc(c1)c(C(=O)2)c(OC(c(c7)ccc(c7)O)=C2OC(C(OC(O6)C(O)C(C6)(O)COC(C5O)OCC(O)(CO)5)4)OC(C(C4O)O)COC(C3O)OC(C(C(O)3)O)C)cc(OC)1
M END