Mol:FL5FALNS0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 28 30 0 0 0 0 0 0 0 0999 V2000 | + | 28 30 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.4197 -0.0290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4197 -0.0290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4197 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4197 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8634 -0.9925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8634 -0.9925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3070 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3070 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3070 -0.0290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3070 -0.0290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8634 0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8634 0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7507 -0.9925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7507 -0.9925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1944 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1944 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1944 -0.0290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1944 -0.0290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7507 0.2922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7507 0.2922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7507 -1.4933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7507 -1.4933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3617 0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3617 0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9286 -0.0352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9286 -0.0352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4956 0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4956 0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4956 0.9468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4956 0.9468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9286 1.2741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9286 1.2741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3617 0.9468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3617 0.9468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0624 -0.0352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0624 -0.0352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6716 -1.1713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6716 -1.1713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5377 -1.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5377 -1.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7769 0.5898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7769 0.5898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2768 1.4559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2768 1.4559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2998 -1.1530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2998 -1.1530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5853 -1.5655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5853 -1.5655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0044 1.5655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0044 1.5655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4957 2.4315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4957 2.4315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0624 1.2740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0624 1.2740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7769 0.8615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7769 0.8615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 1 21 1 0 0 0 0 | + | 1 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 3 23 1 0 0 0 0 | + | 3 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 17 25 1 0 0 0 0 | + | 17 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 15 27 1 0 0 0 0 | + | 15 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | M STY 1 5 SUP | + | M STY 1 5 SUP |
| − | M SLB 1 5 5 | + | M SLB 1 5 5 |
| − | M SAL 5 2 27 28 | + | M SAL 5 2 27 28 |
| − | M SBL 5 1 29 | + | M SBL 5 1 29 |
| − | M SMT 5 OCH3 | + | M SMT 5 OCH3 |
| − | M SVB 5 29 2.0624 1.274 | + | M SVB 5 29 2.0624 1.274 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 25 26 | + | M SAL 4 2 25 26 |
| − | M SBL 4 1 27 | + | M SBL 4 1 27 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SVB 4 27 0.0044 1.5655 | + | M SVB 4 27 0.0044 1.5655 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 23 24 | + | M SAL 3 2 23 24 |
| − | M SBL 3 1 25 | + | M SBL 3 1 25 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SVB 3 25 -2.2998 -1.153 | + | M SVB 3 25 -2.2998 -1.153 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 21 22 | + | M SAL 2 2 21 22 |
| − | M SBL 2 1 23 | + | M SBL 2 1 23 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SVB 2 23 -2.7769 0.5898 | + | M SVB 2 23 -2.7769 0.5898 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 19 20 | + | M SAL 1 2 19 20 |
| − | M SBL 1 1 21 | + | M SBL 1 1 21 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SVB 1 21 0.0044 -0.8776 | + | M SVB 1 21 0.0044 -0.8776 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FALNS0014 | + | ID FL5FALNS0014 |
| − | KNApSAcK_ID C00004755 | + | KNApSAcK_ID C00004755 |
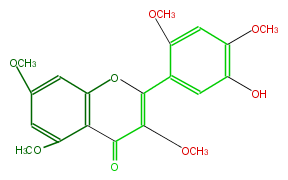
| − | NAME 5'-hydroxy-3,5,7,2',4'-pentamethoxyflavone | + | NAME 5'-hydroxy-3,5,7,2',4'-pentamethoxyflavone |
| − | CAS_RN 129554-05-8 | + | CAS_RN 129554-05-8 |
| − | FORMULA C20H20O8 | + | FORMULA C20H20O8 |
| − | EXACTMASS 388.11581761599996 | + | EXACTMASS 388.11581761599996 |
| − | AVERAGEMASS 388.368 | + | AVERAGEMASS 388.368 |
| − | SMILES COc(c1)c(C(O2)=C(C(c(c3OC)c2cc(c3)OC)=O)OC)cc(O)c1OC | + | SMILES COc(c1)c(C(O2)=C(C(c(c3OC)c2cc(c3)OC)=O)OC)cc(O)c1OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
28 30 0 0 0 0 0 0 0 0999 V2000
-2.4197 -0.0290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4197 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8634 -0.9925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3070 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3070 -0.0290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8634 0.2922 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7507 -0.9925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1944 -0.6713 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1944 -0.0290 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7507 0.2922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7507 -1.4933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3617 0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9286 -0.0352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4956 0.2921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4956 0.9468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9286 1.2741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3617 0.9468 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0624 -0.0352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6716 -1.1713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5377 -1.6712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7769 0.5898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2768 1.4559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2998 -1.1530 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5853 -1.5655 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0044 1.5655 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4957 2.4315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0624 1.2740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7769 0.8615 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
14 18 1 0 0 0 0
8 19 1 0 0 0 0
19 20 1 0 0 0 0
1 21 1 0 0 0 0
21 22 1 0 0 0 0
3 23 1 0 0 0 0
23 24 1 0 0 0 0
17 25 1 0 0 0 0
25 26 1 0 0 0 0
15 27 1 0 0 0 0
27 28 1 0 0 0 0
M STY 1 5 SUP
M SLB 1 5 5
M SAL 5 2 27 28
M SBL 5 1 29
M SMT 5 OCH3
M SVB 5 29 2.0624 1.274
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 25 26
M SBL 4 1 27
M SMT 4 OCH3
M SVB 4 27 0.0044 1.5655
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 23 24
M SBL 3 1 25
M SMT 3 OCH3
M SVB 3 25 -2.2998 -1.153
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 21 22
M SBL 2 1 23
M SMT 2 OCH3
M SVB 2 23 -2.7769 0.5898
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 19 20
M SBL 1 1 21
M SMT 1 OCH3
M SVB 1 21 0.0044 -0.8776
S SKP 8
ID FL5FALNS0014
KNApSAcK_ID C00004755
NAME 5'-hydroxy-3,5,7,2',4'-pentamethoxyflavone
CAS_RN 129554-05-8
FORMULA C20H20O8
EXACTMASS 388.11581761599996
AVERAGEMASS 388.368
SMILES COc(c1)c(C(O2)=C(C(c(c3OC)c2cc(c3)OC)=O)OC)cc(O)c1OC
M END