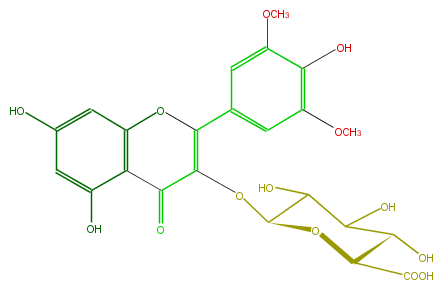
Mol:FL5FAIGS0003
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 37 40 0 0 0 0 0 0 0 0999 V2000 | + | 37 40 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.6836 0.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6836 0.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6837 -0.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6837 -0.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9825 -0.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9825 -0.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2816 -0.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2816 -0.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2815 0.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2815 0.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9825 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9825 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5804 -0.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5804 -0.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8795 -0.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8795 -0.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8794 0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8794 0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5804 0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5804 0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5804 -1.7507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5804 -1.7507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1786 0.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1786 0.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5359 0.2226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5359 0.2226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2504 0.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2504 0.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2503 1.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2503 1.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5359 1.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5359 1.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1786 1.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1786 1.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3844 0.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3844 0.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0077 -1.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0077 -1.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3603 -1.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3603 -1.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5600 -1.6231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5600 -1.6231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5249 -1.7769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5249 -1.7769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2680 -2.4195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2680 -2.4195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0685 -1.8592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0685 -1.8592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1034 -1.7054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1034 -1.7054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5989 -0.9030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5989 -0.9030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8936 -1.2888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8936 -1.2888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6530 -2.2978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6530 -2.2978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0077 1.8974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0077 1.8974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9825 -1.7295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9825 -1.7295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3521 -2.6770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3521 -2.6770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3844 -1.9543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3844 -1.9543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4427 -3.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4427 -3.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9700 0.2196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9700 0.2196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2302 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2302 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5359 2.5820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5359 2.5820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0921 3.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0921 3.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 20 21 1 0 0 0 0 | + | 20 21 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 25 27 1 0 0 0 0 | + | 25 27 1 0 0 0 0 |
| − | 24 28 1 0 0 0 0 | + | 24 28 1 0 0 0 0 |
| − | 21 19 1 0 0 0 0 | + | 21 19 1 0 0 0 0 |
| − | 19 8 1 0 0 0 0 | + | 19 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 15 29 1 0 0 0 0 | + | 15 29 1 0 0 0 0 |
| − | 3 30 1 0 0 0 0 | + | 3 30 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 14 34 1 0 0 0 0 | + | 14 34 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 16 36 1 0 0 0 0 | + | 16 36 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 3 31 32 33 | + | M SAL 1 3 31 32 33 |
| − | M SBL 1 1 36 | + | M SBL 1 1 36 |
| − | M SMT 1 COOH | + | M SMT 1 COOH |
| − | M SBV 1 36 -1.0842 0.2575 | + | M SBV 1 36 -1.0842 0.2575 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 34 35 | + | M SAL 2 2 34 35 |
| − | M SBL 2 1 38 | + | M SBL 2 1 38 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 38 -0.7196 0.4155 | + | M SBV 2 38 -0.7196 0.4155 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 36 37 | + | M SAL 3 2 36 37 |
| − | M SBL 3 1 40 | + | M SBL 3 1 40 |
| − | M SMT 3 OCH3 | + | M SMT 3 OCH3 |
| − | M SBV 3 40 0.0000 -0.7093 | + | M SBV 3 40 0.0000 -0.7093 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAIGS0003 | + | ID FL5FAIGS0003 |
| − | FORMULA C23H22O14 | + | FORMULA C23H22O14 |
| − | EXACTMASS 522.100955412 | + | EXACTMASS 522.100955412 |
| − | AVERAGEMASS 522.41238 | + | AVERAGEMASS 522.41238 |
| − | SMILES c(C2=O)(c4O)c(cc(c4)O)OC(=C(OC(C(O)3)OC(C(O)C3O)C(O)=O)2)c(c1)cc(c(c1OC)O)OC | + | SMILES c(C2=O)(c4O)c(cc(c4)O)OC(=C(OC(C(O)3)OC(C(O)C3O)C(O)=O)2)c(c1)cc(c(c1OC)O)OC |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
37 40 0 0 0 0 0 0 0 0999 V2000
-3.6836 0.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6837 -0.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9825 -0.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2816 -0.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2815 0.2306 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9825 0.6352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5804 -0.9838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8795 -0.5791 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8794 0.2305 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5804 0.6352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5804 -1.7507 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1786 0.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5359 0.2226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2504 0.6351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2503 1.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5359 1.8727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1786 1.4601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3844 0.6350 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0077 -1.0686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3603 -1.0627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5600 -1.6231 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5249 -1.7769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2680 -2.4195 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0685 -1.8592 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1034 -1.7054 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5989 -0.9030 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8936 -1.2888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6530 -2.2978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0077 1.8974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9825 -1.7295 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3521 -2.6770 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3844 -1.9543 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4427 -3.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9700 0.2196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2302 0.2197 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5359 2.5820 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0921 3.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
20 21 1 0 0 0 0
21 22 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
25 27 1 0 0 0 0
24 28 1 0 0 0 0
21 19 1 0 0 0 0
19 8 1 0 0 0 0
23 22 1 1 0 0 0
15 29 1 0 0 0 0
3 30 1 0 0 0 0
31 32 2 0 0 0 0
31 33 1 0 0 0 0
23 31 1 0 0 0 0
34 35 1 0 0 0 0
14 34 1 0 0 0 0
36 37 1 0 0 0 0
16 36 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 3 31 32 33
M SBL 1 1 36
M SMT 1 COOH
M SBV 1 36 -1.0842 0.2575
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 34 35
M SBL 2 1 38
M SMT 2 OCH3
M SBV 2 38 -0.7196 0.4155
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 36 37
M SBL 3 1 40
M SMT 3 OCH3
M SBV 3 40 0.0000 -0.7093
S SKP 5
ID FL5FAIGS0003
FORMULA C23H22O14
EXACTMASS 522.100955412
AVERAGEMASS 522.41238
SMILES c(C2=O)(c4O)c(cc(c4)O)OC(=C(OC(C(O)3)OC(C(O)C3O)C(O)=O)2)c(c1)cc(c(c1OC)O)OC
M END