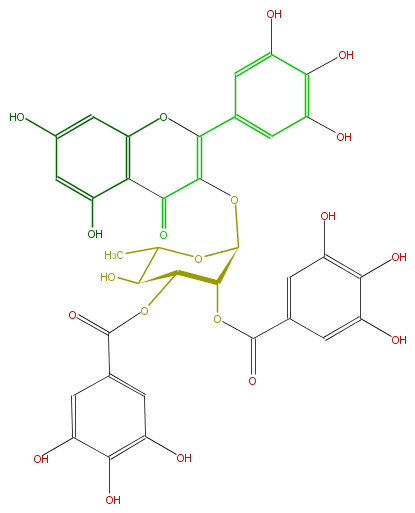
Mol:FL5FAGGS0033
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.2497 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2497 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5352 2.7954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5352 2.7954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5352 3.6204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5352 3.6204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2497 4.0329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2497 4.0329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9642 3.6204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9642 3.6204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9642 2.7954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9642 2.7954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1792 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1792 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8937 2.7954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8937 2.7954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6082 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6082 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6082 1.5579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6082 1.5579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8937 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8937 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1792 1.5579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1792 1.5579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3226 2.7954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3226 2.7954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0371 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0371 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0371 1.5579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0371 1.5579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3226 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3226 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8937 0.4384 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8937 0.4384 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7516 2.7954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7516 2.7954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5352 1.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5352 1.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3226 0.4544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3226 0.4544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6786 4.0329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6786 4.0329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2497 4.8579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2497 4.8579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6421 2.4040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6421 2.4040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9926 0.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9926 0.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4052 -0.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4052 -0.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6117 -0.3283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6117 -0.3283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1866 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1866 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5993 0.1771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5993 0.1771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1943 -0.0496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1943 -0.0496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2721 -1.0759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2721 -1.0759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9222 -0.4048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9222 -0.4048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7683 0.0355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7683 0.0355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6091 -0.4230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6091 -0.4230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6091 -1.2480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6091 -1.2480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3236 -1.6605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3236 -1.6605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0381 -1.2480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0381 -1.2480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0381 -0.4230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0381 -0.4230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3236 -0.0105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3236 -0.0105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3236 0.8134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3236 0.8134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7516 -0.0111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7516 -0.0111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7516 -1.6599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7516 -1.6599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8956 -1.6599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8956 -1.6599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8956 -2.4838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8956 -2.4838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1821 -1.2480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1821 -1.2480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2734 -2.7924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2734 -2.7924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9903 -2.3840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9903 -2.3840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7023 -2.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7023 -2.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6975 -3.6257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6975 -3.6257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9806 -4.0340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9806 -4.0340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2686 -3.6174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2686 -3.6174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5527 -4.0251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5527 -4.0251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9758 -4.8579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9758 -4.8579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4086 -4.0418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4086 -4.0418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9951 -1.5602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9951 -1.5602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7110 -1.1524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7110 -1.1524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 1 0 0 0 | + | 25 26 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 28 27 1 1 0 0 0 | + | 28 27 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 49 52 1 0 0 0 0 | + | 49 52 1 0 0 0 0 |
| − | 48 53 1 0 0 0 0 | + | 48 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 30 54 1 0 0 0 0 | + | 30 54 1 0 0 0 0 |
| − | 44 27 1 0 0 0 0 | + | 44 27 1 0 0 0 0 |
| − | 28 19 1 0 0 0 0 | + | 28 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAGGS0033 | + | ID FL5FAGGS0033 |
| − | FORMULA C35H28O20 | + | FORMULA C35H28O20 |
| − | EXACTMASS 768.117393336 | + | EXACTMASS 768.117393336 |
| − | AVERAGEMASS 768.58482 | + | AVERAGEMASS 768.58482 |
| − | SMILES C(OC(c(c6)cc(c(O)c(O)6)O)=O)(C(OC(c(c5)cc(c(O)c5O)O)=O)1)C(OC(C4=O)=C(Oc(c34)cc(O)cc3O)c(c2)cc(c(c(O)2)O)O)OC(C)C(O)1 | + | SMILES C(OC(c(c6)cc(c(O)c(O)6)O)=O)(C(OC(c(c5)cc(c(O)c5O)O)=O)1)C(OC(C4=O)=C(Oc(c34)cc(O)cc3O)c(c2)cc(c(c(O)2)O)O)OC(C)C(O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
1.2497 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5352 2.7954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5352 3.6204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2497 4.0329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9642 3.6204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9642 2.7954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1792 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8937 2.7954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6082 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6082 1.5579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8937 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1792 1.5579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3226 2.7954 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0371 2.3829 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0371 1.5579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3226 1.1454 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8937 0.4384 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7516 2.7954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5352 1.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3226 0.4544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6786 4.0329 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2497 4.8579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6421 2.4040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9926 0.1596 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4052 -0.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6117 -0.3283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1866 -0.5376 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5993 0.1771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1943 -0.0496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2721 -1.0759 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9222 -0.4048 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7683 0.0355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6091 -0.4230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6091 -1.2480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3236 -1.6605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0381 -1.2480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0381 -0.4230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3236 -0.0105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3236 0.8134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7516 -0.0111 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7516 -1.6599 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8956 -1.6599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8956 -2.4838 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1821 -1.2480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2734 -2.7924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9903 -2.3840 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7023 -2.8007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6975 -3.6257 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9806 -4.0340 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2686 -3.6174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5527 -4.0251 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9758 -4.8579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4086 -4.0418 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9951 -1.5602 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7110 -1.1524 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
6 23 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 1 0 0 0
27 26 1 1 0 0 0
28 27 1 1 0 0 0
28 29 1 0 0 0 0
29 24 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
24 32 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
37 40 1 0 0 0 0
36 41 1 0 0 0 0
34 42 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 45 1 0 0 0 0
50 51 1 0 0 0 0
49 52 1 0 0 0 0
48 53 1 0 0 0 0
46 54 1 0 0 0 0
54 55 2 0 0 0 0
30 54 1 0 0 0 0
44 27 1 0 0 0 0
28 19 1 0 0 0 0
S SKP 5
ID FL5FAGGS0033
FORMULA C35H28O20
EXACTMASS 768.117393336
AVERAGEMASS 768.58482
SMILES C(OC(c(c6)cc(c(O)c(O)6)O)=O)(C(OC(c(c5)cc(c(O)c5O)O)=O)1)C(OC(C4=O)=C(Oc(c34)cc(O)cc3O)c(c2)cc(c(c(O)2)O)O)OC(C)C(O)1
M END