Mol:FL5FAGGS0029
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 45 49 0 0 0 0 0 0 0 0999 V2000 | + | 45 49 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.3240 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3240 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6096 2.3723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6096 2.3723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6096 3.1973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6096 3.1973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3240 3.6098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3240 3.6098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0385 3.1973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0385 3.1973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0385 2.3723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0385 2.3723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1049 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1049 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8194 2.3723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8194 2.3723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5339 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5339 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5339 1.1348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5339 1.1348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8194 0.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8194 0.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1049 1.1348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1049 1.1348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2483 2.3723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2483 2.3723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9628 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9628 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9628 1.1348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9628 1.1348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2483 0.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2483 0.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8194 0.0153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8194 0.0153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6773 2.3723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6773 2.3723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6096 0.7223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6096 0.7223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2483 0.0313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2483 0.0313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7530 3.6098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7530 3.6098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3240 4.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3240 4.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7164 1.9809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7164 1.9809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5991 -1.6359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5991 -1.6359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0096 -1.9911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0096 -1.9911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3682 -2.5100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3682 -2.5100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4968 -3.3249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4968 -3.3249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2668 -3.6210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2668 -3.6210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9083 -3.1021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9083 -3.1021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7796 -2.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7796 -2.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4202 -1.7691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4202 -1.7691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6773 -3.3978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6773 -3.3978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3953 -4.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3953 -4.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5992 -2.2143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5992 -2.2143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0414 -2.7325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0414 -2.7325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9079 -1.2086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9079 -1.2086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1439 -1.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1439 -1.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3590 -0.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3590 -0.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1252 -0.5598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1252 -0.5598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3613 -0.2475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3613 -0.2475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1418 -0.9018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1418 -0.9018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6247 -0.5349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6247 -0.5349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1737 -0.6098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1737 -0.6098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9938 -1.8865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9938 -1.8865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6573 -1.5909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6573 -1.5909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 25 1 0 0 0 0 | + | 30 25 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 29 32 1 0 0 0 0 | + | 29 32 1 0 0 0 0 |
| − | 28 33 1 0 0 0 0 | + | 28 33 1 0 0 0 0 |
| − | 26 34 1 0 0 0 0 | + | 26 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 24 34 1 0 0 0 0 | + | 24 34 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 39 38 1 1 0 0 0 | + | 39 38 1 1 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 45 1 0 0 0 0 | + | 37 45 1 0 0 0 0 |
| − | 39 19 1 0 0 0 0 | + | 39 19 1 0 0 0 0 |
| − | 24 38 1 0 0 0 0 | + | 24 38 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAGGS0029 | + | ID FL5FAGGS0029 |
| − | KNApSAcK_ID C00013971 | + | KNApSAcK_ID C00013971 |
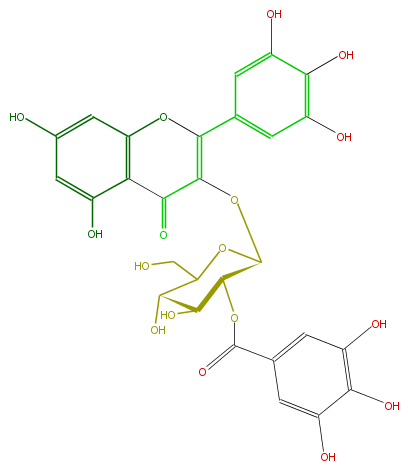
| − | NAME Myricetin 3-(2''-galloylglucoside);5,7-Dihydroxy-3-[[2-O-(3,4,5-trihydroxybenzoyl)-beta-D-glucopyranosyl]oxy]-2-(3,4,5-trihydroxyphenyl)-4H-1-benzopyran-4-one | + | NAME Myricetin 3-(2''-galloylglucoside);5,7-Dihydroxy-3-[[2-O-(3,4,5-trihydroxybenzoyl)-beta-D-glucopyranosyl]oxy]-2-(3,4,5-trihydroxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 334475-75-1 | + | CAS_RN 334475-75-1 |
| − | FORMULA C28H24O17 | + | FORMULA C28H24O17 |
| − | EXACTMASS 632.101349342 | + | EXACTMASS 632.101349342 |
| − | AVERAGEMASS 632.47996 | + | AVERAGEMASS 632.47996 |
| − | SMILES O=C(c(c5)cc(O)c(O)c5O)OC(C(OC(C3=O)=C(c(c4)cc(c(c4O)O)O)Oc(c23)cc(cc2O)O)1)C(O)C(C(CO)O1)O | + | SMILES O=C(c(c5)cc(O)c(O)c5O)OC(C(OC(C3=O)=C(c(c4)cc(c(c4O)O)O)Oc(c23)cc(cc2O)O)1)C(O)C(C(CO)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
45 49 0 0 0 0 0 0 0 0999 V2000
1.3240 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6096 2.3723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6096 3.1973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3240 3.6098 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0385 3.1973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0385 2.3723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1049 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8194 2.3723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5339 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5339 1.1348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8194 0.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1049 1.1348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2483 2.3723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9628 1.9598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9628 1.1348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2483 0.7223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8194 0.0153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6773 2.3723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6096 0.7223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2483 0.0313 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7530 3.6098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3240 4.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7164 1.9809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5991 -1.6359 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0096 -1.9911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3682 -2.5100 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4968 -3.3249 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2668 -3.6210 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9083 -3.1021 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7796 -2.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4202 -1.7691 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6773 -3.3978 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3953 -4.4348 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5992 -2.2143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0414 -2.7325 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9079 -1.2086 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1439 -1.5208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3590 -0.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1252 -0.5598 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3613 -0.2475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1418 -0.9018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6247 -0.5349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1737 -0.6098 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9938 -1.8865 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6573 -1.5909 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
6 23 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 25 1 0 0 0 0
30 31 1 0 0 0 0
29 32 1 0 0 0 0
28 33 1 0 0 0 0
26 34 1 0 0 0 0
34 35 2 0 0 0 0
24 34 1 0 0 0 0
36 37 1 1 0 0 0
37 38 1 1 0 0 0
39 38 1 1 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 36 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
36 44 1 0 0 0 0
37 45 1 0 0 0 0
39 19 1 0 0 0 0
24 38 1 0 0 0 0
S SKP 8
ID FL5FAGGS0029
KNApSAcK_ID C00013971
NAME Myricetin 3-(2''-galloylglucoside);5,7-Dihydroxy-3-[[2-O-(3,4,5-trihydroxybenzoyl)-beta-D-glucopyranosyl]oxy]-2-(3,4,5-trihydroxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 334475-75-1
FORMULA C28H24O17
EXACTMASS 632.101349342
AVERAGEMASS 632.47996
SMILES O=C(c(c5)cc(O)c(O)c5O)OC(C(OC(C3=O)=C(c(c4)cc(c(c4O)O)O)Oc(c23)cc(cc2O)O)1)C(O)C(C(CO)O1)O
M END