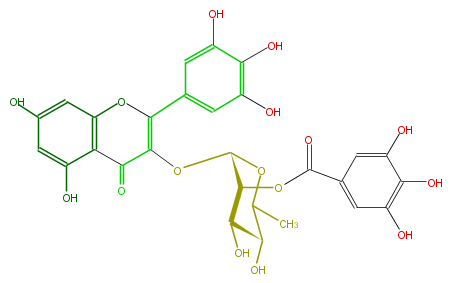
Mol:FL5FAGGS0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.6883 0.4757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6883 0.4757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6883 -0.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6883 -0.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1320 -0.4878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1320 -0.4878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5757 -0.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5757 -0.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5757 0.4757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5757 0.4757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1320 0.7969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1320 0.7969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0194 -0.4878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0194 -0.4878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4631 -0.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4631 -0.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4631 0.4757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4631 0.4757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0194 0.7969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0194 0.7969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0194 -0.9886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0194 -0.9886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7626 0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7626 0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1956 0.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1956 0.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3714 0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3714 0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3714 1.5752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3714 1.5752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1956 1.9026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1956 1.9026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7626 1.5752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7626 1.5752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1320 -1.1299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1320 -1.1299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9782 1.9256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9782 1.9256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1832 0.7969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1832 0.7969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8998 -0.5761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8998 -0.5761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1956 2.5571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1956 2.5571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1530 -1.6287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1530 -1.6287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7812 -1.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7812 -1.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5818 -1.2939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5818 -1.2939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7812 -0.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7812 -0.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1530 -0.2294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1530 -0.2294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3522 -0.9269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3522 -0.9269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1226 -0.9269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1226 -0.9269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3143 -2.2309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3143 -2.2309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6296 -2.5571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6296 -2.5571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2021 -1.6520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2021 -1.6520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9382 0.5933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9382 0.5933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3591 -0.8139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3591 -0.8139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6632 -1.3407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6632 -1.3407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2714 -1.3407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2714 -1.3407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5755 -0.8139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5755 -0.8139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2714 -0.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2714 -0.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6632 -0.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6632 -0.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5752 0.2391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5752 0.2391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1832 -0.8139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1832 -0.8139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5752 -1.8670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5752 -1.8670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7263 -0.5784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7263 -0.5784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7263 0.0451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7263 0.0451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 23 1 1 0 0 0 | + | 28 23 1 1 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 25 32 1 0 0 0 0 | + | 25 32 1 0 0 0 0 |
| − | 27 21 1 0 0 0 0 | + | 27 21 1 0 0 0 0 |
| − | 14 33 1 0 0 0 0 | + | 14 33 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 38 40 1 0 0 0 0 | + | 38 40 1 0 0 0 0 |
| − | 37 41 1 0 0 0 0 | + | 37 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 29 43 1 0 0 0 0 | + | 29 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 34 1 0 0 0 0 | + | 43 34 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAGGS0013 | + | ID FL5FAGGS0013 |
| − | KNApSAcK_ID C00006042 | + | KNApSAcK_ID C00006042 |
| − | NAME Desmanthin 1 | + | NAME Desmanthin 1 |
| − | CAS_RN 56939-52-7 | + | CAS_RN 56939-52-7 |
| − | FORMULA C28H24O16 | + | FORMULA C28H24O16 |
| − | EXACTMASS 616.1064347199999 | + | EXACTMASS 616.1064347199999 |
| − | AVERAGEMASS 616.48056 | + | AVERAGEMASS 616.48056 |
| − | SMILES C(OC(C4OC(c(c5)cc(O)c(O)c(O)5)=O)OC(C(C4O)O)C)(C(=O)2)=C(Oc(c3)c2c(O)cc3O)c(c1)cc(c(c(O)1)O)O | + | SMILES C(OC(C4OC(c(c5)cc(O)c(O)c(O)5)=O)OC(C(C4O)O)C)(C(=O)2)=C(Oc(c3)c2c(O)cc3O)c(c1)cc(c(c(O)1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-3.6883 0.4757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6883 -0.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1320 -0.4878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5757 -0.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5757 0.4757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1320 0.7969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0194 -0.4878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4631 -0.1666 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4631 0.4757 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0194 0.7969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0194 -0.9886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7626 0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1956 0.5932 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3714 0.9205 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3714 1.5752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1956 1.9026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7626 1.5752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1320 -1.1299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9782 1.9256 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1832 0.7969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8998 -0.5761 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1956 2.5571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1530 -1.6287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7812 -1.9914 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5818 -1.2939 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7812 -0.5922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1530 -0.2294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3522 -0.9269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1226 -0.9269 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3143 -2.2309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6296 -2.5571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2021 -1.6520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9382 0.5933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3591 -0.8139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6632 -1.3407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2714 -1.3407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5755 -0.8139 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2714 -0.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6632 -0.2872 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5752 0.2391 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1832 -0.8139 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5752 -1.8670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7263 -0.5784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7263 0.0451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
16 22 1 0 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 1 0 0 0
28 23 1 1 0 0 0
28 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
25 32 1 0 0 0 0
27 21 1 0 0 0 0
14 33 1 0 0 0 0
34 35 2 0 0 0 0
35 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 34 1 0 0 0 0
38 40 1 0 0 0 0
37 41 1 0 0 0 0
36 42 1 0 0 0 0
29 43 1 0 0 0 0
43 44 2 0 0 0 0
43 34 1 0 0 0 0
S SKP 8
ID FL5FAGGS0013
KNApSAcK_ID C00006042
NAME Desmanthin 1
CAS_RN 56939-52-7
FORMULA C28H24O16
EXACTMASS 616.1064347199999
AVERAGEMASS 616.48056
SMILES C(OC(C4OC(c(c5)cc(O)c(O)c(O)5)=O)OC(C(C4O)O)C)(C(=O)2)=C(Oc(c3)c2c(O)cc3O)c(c1)cc(c(c(O)1)O)O
M END