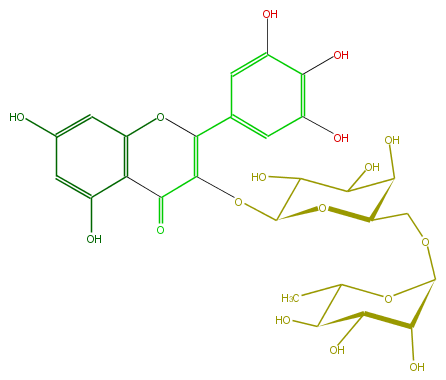
Mol:FL5FAGGA0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.4413 1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4413 1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4413 0.2541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4413 0.2541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7402 -0.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7402 -0.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0392 0.2541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0392 0.2541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0392 1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0392 1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7402 1.4682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7402 1.4682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3382 -0.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3382 -0.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6372 0.2541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6372 0.2541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6372 1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6372 1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3382 1.4682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3382 1.4682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3382 -0.7817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3382 -0.7817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0636 1.4681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0636 1.4681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7781 1.0557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7781 1.0557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4926 1.4681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4926 1.4681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4926 2.2931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4926 2.2931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7781 2.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7781 2.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0636 2.2931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0636 2.2931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7402 -0.9598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7402 -0.9598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2702 -1.8999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2702 -1.8999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7816 -2.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7816 -2.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7210 -2.4774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7210 -2.4774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6662 -2.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6662 -2.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1420 -1.7965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1420 -1.7965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2153 -2.1683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2153 -2.1683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3629 -2.1430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3629 -2.1430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4503 0.2301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4503 0.2301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9619 -0.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9619 -0.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9013 -0.3474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9013 -0.3474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8463 -0.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8463 -0.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3350 0.2301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3350 0.2301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3955 -0.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3955 -0.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2256 -0.2221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2256 -0.2221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8263 0.4640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8263 0.4640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2126 1.0140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2126 1.0140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5784 -0.4729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5784 -0.4729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9575 -1.0310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9575 -1.0310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1725 -2.5828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1725 -2.5828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1086 -3.1944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1086 -3.1944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7088 -3.5303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7088 -3.5303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6884 0.2686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6884 0.2686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1420 1.4681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1420 1.4681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2068 2.7055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2068 2.7055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7781 3.5303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7781 3.5303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2068 1.0558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2068 1.0558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 30 34 1 0 0 0 0 | + | 30 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 23 36 1 0 0 0 0 | + | 23 36 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 20 37 1 0 0 0 0 | + | 20 37 1 0 0 0 0 |
| − | 21 38 1 0 0 0 0 | + | 21 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 32 8 1 0 0 0 0 | + | 32 8 1 0 0 0 0 |
| − | 27 32 1 0 0 0 0 | + | 27 32 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | 1 41 1 0 0 0 0 | + | 1 41 1 0 0 0 0 |
| − | 15 42 1 0 0 0 0 | + | 15 42 1 0 0 0 0 |
| − | 16 43 1 0 0 0 0 | + | 16 43 1 0 0 0 0 |
| − | 14 44 1 0 0 0 0 | + | 14 44 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAGGA0002 | + | ID FL5FAGGA0002 |
| − | FORMULA C27H30O17 | + | FORMULA C27H30O17 |
| − | EXACTMASS 626.148299534 | + | EXACTMASS 626.148299534 |
| − | AVERAGEMASS 626.5169000000001 | + | AVERAGEMASS 626.5169000000001 |
| − | SMILES O(C1C)C(OCC(O2)C(C(O)C(C(OC(=C4c(c5)cc(c(c(O)5)O)O)C(=O)c(c3O)c(O4)cc(c3)O)2)O)O)C(C(O)C(O)1)O | + | SMILES O(C1C)C(OCC(O2)C(C(O)C(C(OC(=C4c(c5)cc(c(c(O)5)O)O)C(=O)c(c3O)c(O4)cc(c3)O)2)O)O)C(C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-3.4413 1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4413 0.2541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7402 -0.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0392 0.2541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0392 1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7402 1.4682 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3382 -0.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6372 0.2541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6372 1.0635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3382 1.4682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3382 -0.7817 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0636 1.4681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7781 1.0557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4926 1.4681 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4926 2.2931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7781 2.7056 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0636 2.2931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7402 -0.9598 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2702 -1.8999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7816 -2.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7210 -2.4774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6662 -2.7460 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1420 -1.7965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2153 -2.1683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3629 -2.1430 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4503 0.2301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9619 -0.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9013 -0.3474 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8463 -0.6159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3350 0.2301 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3955 -0.0382 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2256 -0.2221 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8263 0.4640 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2126 1.0140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5784 -0.4729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9575 -1.0310 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1725 -2.5828 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1086 -3.1944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7088 -3.5303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6884 0.2686 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1420 1.4681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2068 2.7055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7781 3.5303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2068 1.0558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
26 27 1 0 0 0 0
27 28 1 1 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
31 33 1 0 0 0 0
30 34 1 0 0 0 0
29 35 1 0 0 0 0
23 36 1 0 0 0 0
35 36 1 0 0 0 0
20 37 1 0 0 0 0
21 38 1 0 0 0 0
22 39 1 0 0 0 0
32 8 1 0 0 0 0
27 32 1 0 0 0 0
26 40 1 0 0 0 0
1 41 1 0 0 0 0
15 42 1 0 0 0 0
16 43 1 0 0 0 0
14 44 1 0 0 0 0
S SKP 5
ID FL5FAGGA0002
FORMULA C27H30O17
EXACTMASS 626.148299534
AVERAGEMASS 626.5169000000001
SMILES O(C1C)C(OCC(O2)C(C(O)C(C(OC(=C4c(c5)cc(c(c(O)5)O)O)C(=O)c(c3O)c(O4)cc(c3)O)2)O)O)C(C(O)C(O)1)O
M END