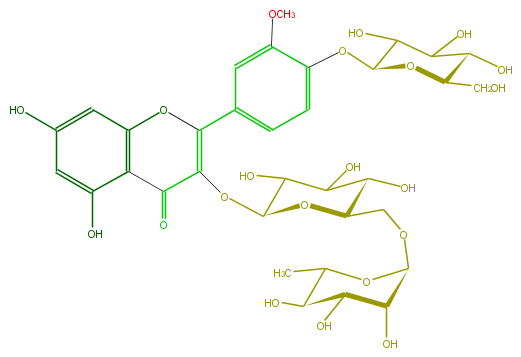
Mol:FL5FADGL0029
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.0963 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0963 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0963 -0.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0963 -0.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3819 -0.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3819 -0.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6674 -0.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6674 -0.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6674 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6674 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3819 0.7868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3819 0.7868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9529 -0.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9529 -0.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2385 -0.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2385 -0.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2385 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2385 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9529 0.7868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9529 0.7868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9529 -1.5064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9529 -1.5064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5243 0.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5243 0.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2038 0.3662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2038 0.3662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9320 0.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9320 0.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9320 1.6274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9320 1.6274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2038 2.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2038 2.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5243 1.6274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5243 1.6274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8105 0.7867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8105 0.7867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7323 -0.9502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7323 -0.9502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3819 -1.6879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3819 -1.6879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2753 -2.4013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2753 -2.4013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8442 -3.1480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8442 -3.1480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6733 -2.9110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6733 -2.9110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5073 -3.1480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5073 -3.1480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9386 -2.4013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9386 -2.4013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1095 -2.6381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1095 -2.6381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5391 -2.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5391 -2.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4699 -0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4699 -0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0388 -1.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0388 -1.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8679 -1.1080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8679 -1.1080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7020 -1.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7020 -1.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1332 -0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1332 -0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3041 -0.8351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3041 -0.8351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2057 -0.5254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2057 -0.5254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7706 -0.3483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7706 -0.3483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8789 -0.7695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8789 -0.7695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4321 -1.2226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4321 -1.2226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8470 -1.7017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8470 -1.7017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2864 -3.0561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2864 -3.0561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1960 -3.5161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1960 -3.5161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5073 -3.8755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5073 -3.8755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6346 1.9780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6346 1.9780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7187 2.1692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7187 2.1692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2309 1.5879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2309 1.5879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9857 1.6667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9857 1.6667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6721 1.3339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6721 1.3339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1599 1.9151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1599 1.9151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4053 1.8366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4053 1.8366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9745 2.3291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9745 2.3291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9953 2.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9953 2.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8105 1.5900 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8105 1.5900 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3101 1.2395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3101 1.2395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1262 0.1962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1262 0.1962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2199 2.7231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2199 2.7231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7868 3.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7868 3.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 24 41 1 0 0 0 0 | + | 24 41 1 0 0 0 0 |
| − | 29 19 1 0 0 0 0 | + | 29 19 1 0 0 0 0 |
| − | 42 15 1 0 0 0 0 | + | 42 15 1 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 47 46 1 1 0 0 0 | + | 47 46 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 48 50 1 0 0 0 0 | + | 48 50 1 0 0 0 0 |
| − | 47 51 1 0 0 0 0 | + | 47 51 1 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 16 54 1 0 0 0 0 | + | 16 54 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 52 53 | + | M SAL 1 2 52 53 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 58 -0.6380 0.0943 | + | M SBV 1 58 -0.6380 0.0943 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 54 55 | + | M SAL 2 2 54 55 |
| − | M SBL 2 1 60 | + | M SBL 2 1 60 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 60 -0.0161 -0.6753 | + | M SBV 2 60 -0.0161 -0.6753 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FADGL0029 | + | ID FL5FADGL0029 |
| − | FORMULA C34H42O21 | + | FORMULA C34H42O21 |
| − | EXACTMASS 786.2218584059999 | + | EXACTMASS 786.2218584059999 |
| − | AVERAGEMASS 786.68468 | + | AVERAGEMASS 786.68468 |
| − | SMILES OC(C1O)C(O)C(Oc(c2OC)ccc(C(=C(OC(O5)C(O)C(C(O)C5COC(C6O)OC(C)C(C6O)O)O)3)Oc(c4)c(c(cc4O)O)C3=O)c2)OC1CO | + | SMILES OC(C1O)C(O)C(Oc(c2OC)ccc(C(=C(OC(O5)C(O)C(C(O)C5COC(C6O)OC(C)C(C6O)O)O)3)Oc(c4)c(c(cc4O)O)C3=O)c2)OC1CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
-4.0963 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0963 -0.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3819 -0.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6674 -0.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6674 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3819 0.7868 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9529 -0.8631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2385 -0.4507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2385 0.3743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9529 0.7868 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9529 -1.5064 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5243 0.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2038 0.3662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9320 0.7867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9320 1.6274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2038 2.0479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5243 1.6274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8105 0.7867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7323 -0.9502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3819 -1.6879 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2753 -2.4013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8442 -3.1480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6733 -2.9110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5073 -3.1480 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9386 -2.4013 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1095 -2.6381 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5391 -2.4696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4699 -0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0388 -1.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8679 -1.1080 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7020 -1.3450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1332 -0.5983 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3041 -0.8351 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2057 -0.5254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7706 -0.3483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8789 -0.7695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4321 -1.2226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8470 -1.7017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2864 -3.0561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1960 -3.5161 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5073 -3.8755 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6346 1.9780 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7187 2.1692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2309 1.5879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9857 1.6667 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6721 1.3339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1599 1.9151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4053 1.8366 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9745 2.3291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9953 2.3124 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8105 1.5900 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.3101 1.2395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1262 0.1962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2199 2.7231 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7868 3.8755 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
25 38 1 0 0 0 0
37 38 1 0 0 0 0
22 39 1 0 0 0 0
23 40 1 0 0 0 0
24 41 1 0 0 0 0
29 19 1 0 0 0 0
42 15 1 0 0 0 0
43 44 1 0 0 0 0
44 45 1 1 0 0 0
45 46 1 1 0 0 0
47 46 1 1 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
43 49 1 0 0 0 0
48 50 1 0 0 0 0
47 51 1 0 0 0 0
42 44 1 0 0 0 0
52 53 1 0 0 0 0
46 52 1 0 0 0 0
54 55 1 0 0 0 0
16 54 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 52 53
M SBL 1 1 58
M SMT 1 CH2OH
M SBV 1 58 -0.6380 0.0943
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 54 55
M SBL 2 1 60
M SMT 2 OCH3
M SBV 2 60 -0.0161 -0.6753
S SKP 5
ID FL5FADGL0029
FORMULA C34H42O21
EXACTMASS 786.2218584059999
AVERAGEMASS 786.68468
SMILES OC(C1O)C(O)C(Oc(c2OC)ccc(C(=C(OC(O5)C(O)C(C(O)C5COC(C6O)OC(C)C(C6O)O)O)3)Oc(c4)c(c(cc4O)O)C3=O)c2)OC1CO
M END