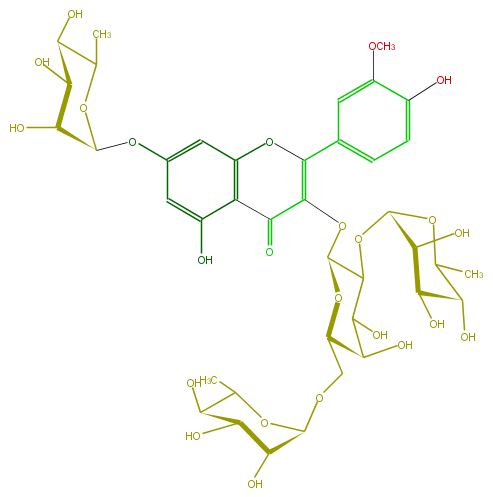
Mol:FL5FADGA0019
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 64 70 0 0 0 0 0 0 0 0999 V2000 | + | 64 70 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.5404 1.5158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5404 1.5158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5404 0.7264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5404 0.7264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8568 0.3318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8568 0.3318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1732 0.7264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1732 0.7264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1732 1.5158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1732 1.5158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8568 1.9105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8568 1.9105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5104 0.3318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5104 0.3318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1940 0.7264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1940 0.7264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1940 1.5158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1940 1.5158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5104 1.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5104 1.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5104 -0.2837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5104 -0.2837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8774 1.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8774 1.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5742 1.5081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5742 1.5081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2709 1.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2709 1.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2709 2.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2709 2.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5742 3.1171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5742 3.1171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8774 2.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8774 2.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2238 1.9104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2238 1.9104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9686 0.2266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9686 0.2266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8568 -0.4574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8568 -0.4574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9240 3.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9240 3.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3853 -0.8363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3853 -0.8363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6691 -0.4227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6691 -0.4227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8964 -1.2179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8964 -1.2179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6691 -2.0179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6691 -2.0179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3853 -2.4314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3853 -2.4314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1581 -1.6362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1581 -1.6362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2840 -0.0285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2840 -0.0285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6585 -1.9549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6585 -1.9549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1578 -2.1533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1578 -2.1533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9530 -2.7473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9530 -2.7473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7370 2.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7370 2.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9651 1.7263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9651 1.7263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2100 2.5835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2100 2.5835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9651 3.4457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9651 3.4457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7370 3.8916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7370 3.8916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4921 3.0343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4921 3.0343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4444 4.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4444 4.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9541 4.0649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9541 4.0649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1027 3.5077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1027 3.5077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4703 2.1815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4703 2.1815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4746 -1.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4746 -1.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3524 -1.2787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3524 -1.2787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8291 -0.5570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8291 -0.5570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7643 0.3372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7643 0.3372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8864 0.4921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8864 0.4921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4098 -0.2297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4098 -0.2297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2993 0.0941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2993 0.0941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7917 -1.7322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7917 -1.7322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4152 -1.9954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4152 -1.9954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4703 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4703 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5091 -3.2110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5091 -3.2110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1713 -3.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1713 -3.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8858 -3.5278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8858 -3.5278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0854 -3.7281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0854 -3.7281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4924 -4.3235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4924 -4.3235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2070 -3.9111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2070 -3.9111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4066 -3.7106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4066 -3.7106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6031 -2.8485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6031 -2.8485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0761 -2.9104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0761 -2.9104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9469 -3.9642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9469 -3.9642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1444 -4.9263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1444 -4.9263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5732 3.8237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5732 3.8237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1156 4.9263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1156 4.9263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 33 18 1 0 0 0 0 | + | 33 18 1 0 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 47 42 1 1 0 0 0 | + | 47 42 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 42 49 1 0 0 0 0 | + | 42 49 1 0 0 0 0 |
| − | 43 50 1 0 0 0 0 | + | 43 50 1 0 0 0 0 |
| − | 44 51 1 0 0 0 0 | + | 44 51 1 0 0 0 0 |
| − | 28 46 1 0 0 0 0 | + | 28 46 1 0 0 0 0 |
| − | 31 52 1 0 0 0 0 | + | 31 52 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 57 56 1 1 0 0 0 | + | 57 56 1 1 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 53 1 0 0 0 0 | + | 58 53 1 0 0 0 0 |
| − | 53 59 1 0 0 0 0 | + | 53 59 1 0 0 0 0 |
| − | 54 60 1 0 0 0 0 | + | 54 60 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 56 62 1 0 0 0 0 | + | 56 62 1 0 0 0 0 |
| − | 57 52 1 0 0 0 0 | + | 57 52 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 16 63 1 0 0 0 0 | + | 16 63 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 63 64 | + | M SAL 1 2 63 64 |
| − | M SBL 1 1 70 | + | M SBL 1 1 70 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 70 0.0010 -0.7066 | + | M SBV 1 70 0.0010 -0.7066 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FADGA0019 | + | ID FL5FADGA0019 |
| − | FORMULA C40H52O24 | + | FORMULA C40H52O24 |
| − | EXACTMASS 916.284852592 | + | EXACTMASS 916.284852592 |
| − | AVERAGEMASS 916.82648 | + | AVERAGEMASS 916.82648 |
| − | SMILES OC(C(O)2)C(OC(OC(=C(c(c7)cc(c(c7)O)OC)4)C(=O)c(c6O)c(cc(c6)OC(C(O)5)OC(C)C(O)C(O)5)O4)C2OC(C3O)OC(C)C(C3O)O)COC(O1)C(O)C(C(O)C(C)1)O | + | SMILES OC(C(O)2)C(OC(OC(=C(c(c7)cc(c(c7)O)OC)4)C(=O)c(c6O)c(cc(c6)OC(C(O)5)OC(C)C(O)C(O)5)O4)C2OC(C3O)OC(C)C(C3O)O)COC(O1)C(O)C(C(O)C(C)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
64 70 0 0 0 0 0 0 0 0999 V2000
-1.5404 1.5158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5404 0.7264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8568 0.3318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1732 0.7264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1732 1.5158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8568 1.9105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5104 0.3318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1940 0.7264 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1940 1.5158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5104 1.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5104 -0.2837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8774 1.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5742 1.5081 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2709 1.9104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2709 2.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5742 3.1171 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8774 2.7149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2238 1.9104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9686 0.2266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8568 -0.4574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9240 3.1423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3853 -0.8363 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6691 -0.4227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8964 -1.2179 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6691 -2.0179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3853 -2.4314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1581 -1.6362 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2840 -0.0285 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6585 -1.9549 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1578 -2.1533 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9530 -2.7473 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7370 2.1720 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9651 1.7263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2100 2.5835 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9651 3.4457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7370 3.8916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4921 3.0343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4444 4.4727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9541 4.0649 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1027 3.5077 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4703 2.1815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4746 -1.1239 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3524 -1.2787 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8291 -0.5570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7643 0.3372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8864 0.4921 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4098 -0.2297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2993 0.0941 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7917 -1.7322 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4152 -1.9954 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4703 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5091 -3.2110 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1713 -3.1153 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8858 -3.5278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0854 -3.7281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4924 -4.3235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2070 -3.9111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4066 -3.7106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6031 -2.8485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0761 -2.9104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9469 -3.9642 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1444 -4.9263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5732 3.8237 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1156 4.9263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
23 19 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
37 40 1 0 0 0 0
32 41 1 0 0 0 0
33 18 1 0 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
47 42 1 1 0 0 0
47 48 1 0 0 0 0
42 49 1 0 0 0 0
43 50 1 0 0 0 0
44 51 1 0 0 0 0
28 46 1 0 0 0 0
31 52 1 0 0 0 0
53 54 1 0 0 0 0
54 55 1 1 0 0 0
55 56 1 1 0 0 0
57 56 1 1 0 0 0
57 58 1 0 0 0 0
58 53 1 0 0 0 0
53 59 1 0 0 0 0
54 60 1 0 0 0 0
55 61 1 0 0 0 0
56 62 1 0 0 0 0
57 52 1 0 0 0 0
63 64 1 0 0 0 0
16 63 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 63 64
M SBL 1 1 70
M SMT 1 OCH3
M SBV 1 70 0.0010 -0.7066
S SKP 5
ID FL5FADGA0019
FORMULA C40H52O24
EXACTMASS 916.284852592
AVERAGEMASS 916.82648
SMILES OC(C(O)2)C(OC(OC(=C(c(c7)cc(c(c7)O)OC)4)C(=O)c(c6O)c(cc(c6)OC(C(O)5)OC(C)C(O)C(O)5)O4)C2OC(C3O)OC(C)C(C3O)O)COC(O1)C(O)C(C(O)C(C)1)O
M END