Mol:FL5FADGA0014
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.9377 0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9377 0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9377 -0.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9377 -0.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2233 -0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2233 -0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5089 -0.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5089 -0.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5089 0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5089 0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2233 1.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2233 1.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7944 -0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7944 -0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0800 -0.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0800 -0.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0800 0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0800 0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7944 1.1105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7944 1.1105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7944 -1.1826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7944 -1.1826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6342 1.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6342 1.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3623 0.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3623 0.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0905 1.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0905 1.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0905 1.9511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0905 1.9511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3623 2.3715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3623 2.3715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6342 1.9511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6342 1.9511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6519 1.1104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6519 1.1104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6545 -0.5247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6545 -0.5247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2233 -1.3641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2233 -1.3641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7917 2.4061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7917 2.4061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9897 -1.6906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9897 -1.6906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2413 -1.2584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2413 -1.2584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4787 -2.0894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4787 -2.0894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2413 -2.9255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2413 -2.9255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9897 -3.3576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9897 -3.3576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7523 -2.5266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7523 -2.5266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9410 -0.8688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9410 -0.8688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2020 -2.8496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2020 -2.8496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6488 -3.0698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6488 -3.0698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3341 -3.5653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3341 -3.5653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6825 -3.8518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6825 -3.8518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0072 -4.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0072 -4.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2214 -3.4920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2214 -3.4920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0072 -2.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0072 -2.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6825 -2.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6825 -2.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4683 -3.0976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4683 -3.0976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4375 -1.8553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4375 -1.8553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9954 -2.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9954 -2.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1362 -2.6061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1362 -2.6061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2722 -3.8617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2722 -3.8617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1967 -3.9435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1967 -3.9435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4701 -1.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4701 -1.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4017 -1.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4017 -1.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7321 -1.1690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7321 -1.1690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5033 -0.2605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5033 -0.2605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5715 -0.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5715 -0.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2411 -0.9083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2411 -0.9083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0979 -0.4137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0979 -0.4137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9345 -2.3431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9345 -2.3431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6390 -2.4209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6390 -2.4209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6519 -1.1690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6519 -1.1690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3770 3.0894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3770 3.0894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9439 4.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9439 4.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 15 1 0 0 0 0 | + | 21 15 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 23 19 1 0 0 0 0 | + | 23 19 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 42 31 1 0 0 0 0 | + | 42 31 1 0 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 1 0 0 0 | + | 47 48 1 1 0 0 0 |
| − | 48 43 1 1 0 0 0 | + | 48 43 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 43 50 1 0 0 0 0 | + | 43 50 1 0 0 0 0 |
| − | 44 51 1 0 0 0 0 | + | 44 51 1 0 0 0 0 |
| − | 45 52 1 0 0 0 0 | + | 45 52 1 0 0 0 0 |
| − | 47 28 1 0 0 0 0 | + | 47 28 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 16 53 1 0 0 0 0 | + | 16 53 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 59 | + | M SBL 1 1 59 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 59 -0.0147 -0.7179 | + | M SBV 1 59 -0.0147 -0.7179 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FADGA0014 | + | ID FL5FADGA0014 |
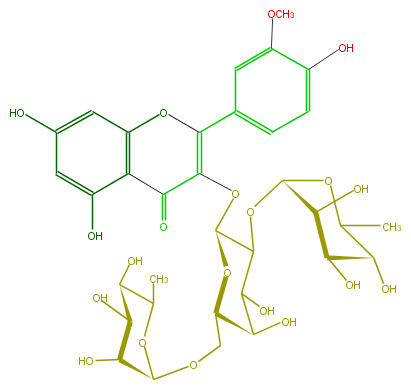
| − | FORMULA C34H42O20 | + | FORMULA C34H42O20 |
| − | EXACTMASS 770.226943784 | + | EXACTMASS 770.226943784 |
| − | AVERAGEMASS 770.6852799999999 | + | AVERAGEMASS 770.6852799999999 |
| − | SMILES C(C(O)1)(C(O)C(C)OC1OC(C3OC(=C5c(c6)cc(OC)c(O)c6)C(=O)c(c(O5)4)c(cc(c4)O)O)C(C(C(O3)COC(O2)C(O)C(C(C2C)O)O)O)O)O | + | SMILES C(C(O)1)(C(O)C(C)OC1OC(C3OC(=C5c(c6)cc(OC)c(O)c6)C(=O)c(c(O5)4)c(cc(c4)O)O)C(C(C(O3)COC(O2)C(O)C(C(C2C)O)O)O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-2.9377 0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9377 -0.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2233 -0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5089 -0.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5089 0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2233 1.1105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7944 -0.5395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0800 -0.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0800 0.6980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7944 1.1105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7944 -1.1826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6342 1.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3623 0.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0905 1.1104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0905 1.9511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3623 2.3715 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6342 1.9511 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6519 1.1104 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6545 -0.5247 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2233 -1.3641 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7917 2.4061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9897 -1.6906 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2413 -1.2584 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4787 -2.0894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2413 -2.9255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9897 -3.3576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7523 -2.5266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9410 -0.8688 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2020 -2.8496 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6488 -3.0698 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3341 -3.5653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6825 -3.8518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0072 -4.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2214 -3.4920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0072 -2.7378 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6825 -2.3477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4683 -3.0976 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4375 -1.8553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9954 -2.2224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1362 -2.6061 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2722 -3.8617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1967 -3.9435 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4701 -1.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4017 -1.8168 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7321 -1.1690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5033 -0.2605 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5715 -0.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2411 -0.9083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0979 -0.4137 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9345 -2.3431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6390 -2.4209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.6519 -1.1690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3770 3.0894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9439 4.2417 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 15 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
25 31 1 0 0 0 0
23 19 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
37 40 1 0 0 0 0
32 41 1 0 0 0 0
33 42 1 0 0 0 0
42 31 1 0 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 0 0 0 0
47 48 1 1 0 0 0
48 43 1 1 0 0 0
48 49 1 0 0 0 0
43 50 1 0 0 0 0
44 51 1 0 0 0 0
45 52 1 0 0 0 0
47 28 1 0 0 0 0
53 54 1 0 0 0 0
16 53 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 59
M SMT 1 OCH3
M SBV 1 59 -0.0147 -0.7179
S SKP 5
ID FL5FADGA0014
FORMULA C34H42O20
EXACTMASS 770.226943784
AVERAGEMASS 770.6852799999999
SMILES C(C(O)1)(C(O)C(C)OC1OC(C3OC(=C5c(c6)cc(OC)c(O)c6)C(=O)c(c(O5)4)c(cc(c4)O)O)C(C(C(O3)COC(O2)C(O)C(C(C2C)O)O)O)O)O
M END