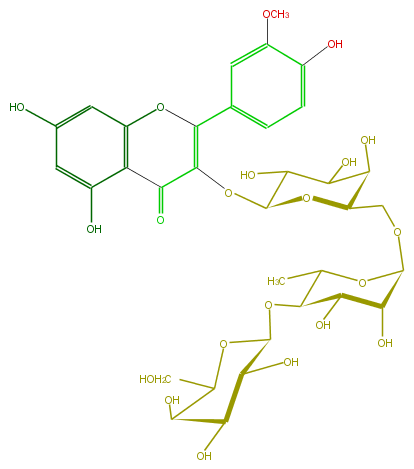
Mol:FL5FADGA0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.1229 1.5723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1229 1.5723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1229 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1229 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4219 0.3580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4219 0.3580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7209 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7209 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7209 1.5723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7209 1.5723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4219 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4219 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0199 0.3580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0199 0.3580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3189 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3189 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3189 1.5723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3189 1.5723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0199 1.9769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0199 1.9769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0199 -0.2730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0199 -0.2730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3819 1.9768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3819 1.9768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0964 1.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0964 1.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8108 1.9768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8108 1.9768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8108 2.8018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8108 2.8018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0964 3.2143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0964 3.2143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3819 2.8018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3819 2.8018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8236 1.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8236 1.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3442 0.2713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3442 0.2713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4219 -0.4511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4219 -0.4511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1917 -1.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1917 -1.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7686 -2.0286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7686 -2.0286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5822 -1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5822 -1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4005 -2.0286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4005 -2.0286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8236 -1.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8236 -1.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0101 -1.5283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0101 -1.5283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4097 -1.4947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4097 -1.4947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5155 0.6760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5155 0.6760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0925 -0.0568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0925 -0.0568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9060 0.1757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9060 0.1757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7243 -0.0568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7243 -0.0568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1475 0.6760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1475 0.6760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3340 0.4435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3340 0.4435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7931 0.6291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7931 0.6291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6703 0.8779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6703 0.8779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0620 1.3259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0620 1.3259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4088 0.0039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4088 0.0039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7101 -0.5223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7101 -0.5223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1383 -1.9733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1383 -1.9733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1536 -2.3676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1536 -2.3676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4005 -2.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4005 -2.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3879 3.2448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3879 3.2448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8060 -4.2756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8060 -4.2756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2685 -4.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2685 -4.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5914 -3.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5914 -3.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1642 -2.6806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1642 -2.6806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2398 -2.7614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2398 -2.7614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0447 -3.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0447 -3.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8649 -3.8696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8649 -3.8696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2269 -4.9858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2269 -4.9858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5236 -3.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5236 -3.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7558 -3.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7558 -3.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2756 -2.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2756 -2.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1056 3.8550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1056 3.8550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6618 4.9858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6618 4.9858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 8 19 1 0 0 0 0 | + | 8 19 1 0 0 0 0 |
| − | 3 20 1 0 0 0 0 | + | 3 20 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 28 1 0 0 0 0 | + | 33 28 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 32 36 1 0 0 0 0 | + | 32 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 25 38 1 0 0 0 0 | + | 25 38 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 22 39 1 0 0 0 0 | + | 22 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 24 41 1 0 0 0 0 | + | 24 41 1 0 0 0 0 |
| − | 29 19 1 0 0 0 0 | + | 29 19 1 0 0 0 0 |
| − | 42 15 1 0 0 0 0 | + | 42 15 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 39 1 0 0 0 0 | + | 46 39 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 16 54 1 0 0 0 0 | + | 16 54 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 52 53 | + | M SAL 1 2 52 53 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 58 0.8005 -0.2087 | + | M SBV 1 58 0.8005 -0.2087 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 54 55 | + | M SAL 2 2 54 55 |
| − | M SBL 2 1 60 | + | M SBL 2 1 60 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 60 -0.0092 -0.6407 | + | M SBV 2 60 -0.0092 -0.6407 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FADGA0012 | + | ID FL5FADGA0012 |
| − | FORMULA C34H42O21 | + | FORMULA C34H42O21 |
| − | EXACTMASS 786.2218584059999 | + | EXACTMASS 786.2218584059999 |
| − | AVERAGEMASS 786.68468 | + | AVERAGEMASS 786.68468 |
| − | SMILES c(c(O)6)c(c(c4c6)C(C(=C(c(c5)cc(OC)c(c5)O)O4)OC(C3O)OC(C(C3O)O)COC(O2)C(O)C(C(C2C)OC(C(O)1)OC(C(O)C1O)CO)O)=O)O | + | SMILES c(c(O)6)c(c(c4c6)C(C(=C(c(c5)cc(OC)c(c5)O)O4)OC(C3O)OC(C(C3O)O)COC(O2)C(O)C(C(C2C)OC(C(O)1)OC(C(O)C1O)CO)O)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
-3.1229 1.5723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1229 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4219 0.3580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7209 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7209 1.5723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4219 1.9769 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0199 0.3580 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3189 0.7628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3189 1.5723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0199 1.9769 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0199 -0.2730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3819 1.9768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0964 1.5643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8108 1.9768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8108 2.8018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0964 3.2143 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3819 2.8018 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8236 1.9768 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3442 0.2713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4219 -0.4511 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1917 -1.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7686 -2.0286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5822 -1.7961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4005 -2.0286 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8236 -1.2958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0101 -1.5283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4097 -1.4947 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5155 0.6760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0925 -0.0568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9060 0.1757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7243 -0.0568 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1475 0.6760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3340 0.4435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7931 0.6291 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6703 0.8779 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0620 1.3259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4088 0.0039 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7101 -0.5223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1383 -1.9733 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1536 -2.3676 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4005 -2.7424 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3879 3.2448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8060 -4.2756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2685 -4.3291 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5914 -3.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1642 -2.6806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2398 -2.7614 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0447 -3.6662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8649 -3.8696 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2269 -4.9858 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5236 -3.1146 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7558 -3.4574 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2756 -2.5572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1056 3.8550 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6618 4.9858 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
8 19 1 0 0 0 0
3 20 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 28 1 0 0 0 0
28 34 1 0 0 0 0
33 35 1 0 0 0 0
32 36 1 0 0 0 0
31 37 1 0 0 0 0
25 38 1 0 0 0 0
37 38 1 0 0 0 0
22 39 1 0 0 0 0
23 40 1 0 0 0 0
24 41 1 0 0 0 0
29 19 1 0 0 0 0
42 15 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 39 1 0 0 0 0
52 53 1 0 0 0 0
48 52 1 0 0 0 0
54 55 1 0 0 0 0
16 54 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 52 53
M SBL 1 1 58
M SMT 1 ^ CH2OH
M SBV 1 58 0.8005 -0.2087
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 54 55
M SBL 2 1 60
M SMT 2 OCH3
M SBV 2 60 -0.0092 -0.6407
S SKP 5
ID FL5FADGA0012
FORMULA C34H42O21
EXACTMASS 786.2218584059999
AVERAGEMASS 786.68468
SMILES c(c(O)6)c(c(c4c6)C(C(=C(c(c5)cc(OC)c(c5)O)O4)OC(C3O)OC(C(C3O)O)COC(O2)C(O)C(C(C2C)OC(C(O)1)OC(C(O)C1O)CO)O)=O)O
M END