Mol:FL5FACGS0084
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.5799 2.2549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5799 2.2549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5799 1.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5799 1.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1345 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1345 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8490 1.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8490 1.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8490 2.2549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8490 2.2549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1345 2.6674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1345 2.6674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2944 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2944 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0089 1.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0089 1.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7234 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7234 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7234 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7234 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0089 -0.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0089 -0.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2944 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2944 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4378 1.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4378 1.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1523 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1523 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1523 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1523 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4378 -0.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4378 -0.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0089 -1.0451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0089 -1.0451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8668 1.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8668 1.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5799 -0.2201 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5799 -0.2201 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4378 -1.0451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4378 -1.0451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4409 2.5966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4409 2.5966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4776 1.0670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4776 1.0670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2985 -2.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2985 -2.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4897 -2.4268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4897 -2.4268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1178 -1.6900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1178 -1.6900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4223 -1.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4223 -1.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2311 -1.0815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2311 -1.0815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6032 -1.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6032 -1.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1461 -1.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1461 -1.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6714 -1.7240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6714 -1.7240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2564 -2.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2564 -2.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9809 -2.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9809 -2.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7518 -2.2423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7518 -2.2423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9077 -1.4611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9077 -1.4611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6642 -1.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6642 -1.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5461 -0.3146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5461 -0.3146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8606 0.4484 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8606 0.4484 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1041 0.1187 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1041 0.1187 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2221 -0.6981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2221 -0.6981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6224 -0.7883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6224 -0.7883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2932 -1.2340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2932 -1.2340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3335 -1.9955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3335 -1.9955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3564 -1.5481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3564 -1.5481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1627 -0.5569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1627 -0.5569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7449 2.7121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7449 2.7121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3322 1.9974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3322 1.9974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1257 2.2242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1257 2.2242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9240 2.0150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9240 2.0150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3368 2.7297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3368 2.7297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5432 2.5030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5432 2.5030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2232 2.4109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2232 2.4109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9850 2.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9850 2.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8668 2.1997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8668 2.1997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4786 2.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4786 2.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7518 1.6904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7518 1.6904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 50 52 1 0 0 0 0 | + | 50 52 1 0 0 0 0 |
| − | 49 53 1 0 0 0 0 | + | 49 53 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 46 21 1 0 0 0 0 | + | 46 21 1 0 0 0 0 |
| − | 37 22 1 0 0 0 0 | + | 37 22 1 0 0 0 0 |
| − | 19 26 1 0 0 0 0 | + | 19 26 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0084 | + | ID FL5FACGS0084 |
| − | KNApSAcK_ID C00013856 | + | KNApSAcK_ID C00013856 |
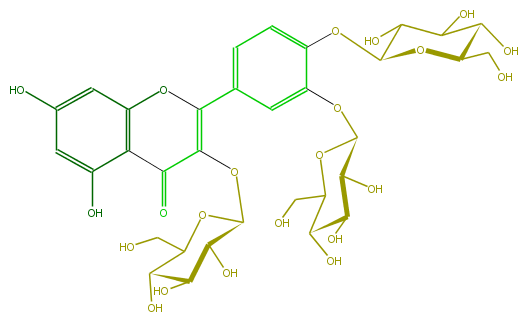
| − | NAME Quercetin 3,3',4'-triglucoside;2-[3,4-Bis(beta-D-glucopyranosyloxy)phenyl]-3-(beta-D-glucopyranosyloxy)-5,7-dihydroxy-4H-1-benzopyran-4-one | + | NAME Quercetin 3,3',4'-triglucoside;2-[3,4-Bis(beta-D-glucopyranosyloxy)phenyl]-3-(beta-D-glucopyranosyloxy)-5,7-dihydroxy-4H-1-benzopyran-4-one |
| − | CAS_RN 352639-75-9 | + | CAS_RN 352639-75-9 |
| − | FORMULA C33H40O22 | + | FORMULA C33H40O22 |
| − | EXACTMASS 788.201122964 | + | EXACTMASS 788.201122964 |
| − | AVERAGEMASS 788.6575 | + | AVERAGEMASS 788.6575 |
| − | SMILES OC(C1O)C(OC(C(=O)3)=C(c(c5)cc(OC(C(O)6)OC(C(C6O)O)CO)c(c5)OC(C4O)OC(C(C4O)O)CO)Oc(c32)cc(O)cc2O)OC(C1O)CO | + | SMILES OC(C1O)C(OC(C(=O)3)=C(c(c5)cc(OC(C(O)6)OC(C(C6O)O)CO)c(c5)OC(C4O)OC(C(C4O)O)CO)Oc(c32)cc(O)cc2O)OC(C1O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
-0.5799 2.2549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5799 1.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1345 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8490 1.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8490 2.2549 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1345 2.6674 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2944 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0089 1.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7234 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7234 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0089 -0.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2944 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4378 1.4299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1523 1.0174 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1523 0.1924 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4378 -0.2201 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0089 -1.0451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8668 1.4299 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5799 -0.2201 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4378 -1.0451 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4409 2.5966 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4776 1.0670 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2985 -2.2627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4897 -2.4268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1178 -1.6900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4223 -1.2457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2311 -1.0815 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6032 -1.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1461 -1.5479 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6714 -1.7240 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2564 -2.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9809 -2.5914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7518 -2.2423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9077 -1.4611 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6642 -1.1314 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5461 -0.3146 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8606 0.4484 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1041 0.1187 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2221 -0.6981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6224 -0.7883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2932 -1.2340 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3335 -1.9955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3564 -1.5481 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1627 -0.5569 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7449 2.7121 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3322 1.9974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1257 2.2242 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9240 2.0150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3368 2.7297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5432 2.5030 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2232 2.4109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9850 2.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8668 2.1997 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.4786 2.1636 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7518 1.6904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
49 48 1 1 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
45 51 1 0 0 0 0
50 52 1 0 0 0 0
49 53 1 0 0 0 0
48 54 1 0 0 0 0
54 55 1 0 0 0 0
46 21 1 0 0 0 0
37 22 1 0 0 0 0
19 26 1 0 0 0 0
S SKP 8
ID FL5FACGS0084
KNApSAcK_ID C00013856
NAME Quercetin 3,3',4'-triglucoside;2-[3,4-Bis(beta-D-glucopyranosyloxy)phenyl]-3-(beta-D-glucopyranosyloxy)-5,7-dihydroxy-4H-1-benzopyran-4-one
CAS_RN 352639-75-9
FORMULA C33H40O22
EXACTMASS 788.201122964
AVERAGEMASS 788.6575
SMILES OC(C1O)C(OC(C(=O)3)=C(c(c5)cc(OC(C(O)6)OC(C(C6O)O)CO)c(c5)OC(C4O)OC(C(C4O)O)CO)Oc(c32)cc(O)cc2O)OC(C1O)CO
M END