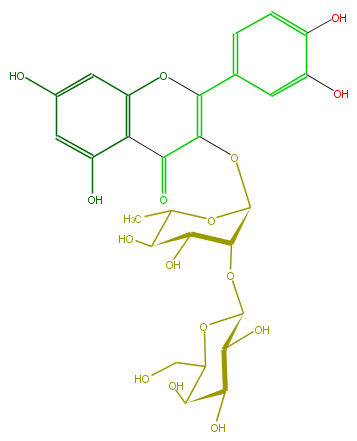
Mol:FL5FACGS0069
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.1147 3.7227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1147 3.7227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1147 2.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1147 2.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8290 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8290 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5435 2.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5435 2.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5435 3.7227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5435 3.7227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8290 4.1352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8290 4.1352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4002 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4002 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3143 2.8977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3143 2.8977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0288 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0288 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0288 1.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0288 1.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3143 1.2478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3143 1.2478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4002 1.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4002 1.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7431 2.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7431 2.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4576 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4576 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4576 1.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4576 1.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7431 1.2478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7431 1.2478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3143 0.4228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3143 0.4228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1721 2.8977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1721 2.8977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1147 1.2478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1147 1.2478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7431 0.4228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7431 0.4228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1354 4.0645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1354 4.0645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1721 2.5349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1721 2.5349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1649 0.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1649 0.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5585 -0.5322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5585 -0.5322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2288 -0.2846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2288 -0.2846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0322 -0.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0322 -0.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4259 0.2525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4259 0.2525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6386 0.0051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6386 0.0051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0226 -1.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0226 -1.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1884 -0.8921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1884 -0.8921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9946 -0.3675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9946 -0.3675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8123 0.0322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8123 0.0322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1265 -3.6690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1265 -3.6690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9142 -3.4226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9142 -3.4226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8845 -2.5979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8845 -2.5979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2792 -1.8732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2792 -1.8732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4916 -2.1196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4916 -2.1196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5212 -2.9444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5212 -2.9444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0605 -2.8599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0605 -2.8599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6662 -3.1551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6662 -3.1551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1415 -3.2957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1415 -3.2957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7064 -4.1352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7064 -4.1352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5822 -2.1802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5822 -2.1802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 23 32 1 0 0 0 0 | + | 23 32 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 29 1 0 0 0 0 | + | 36 29 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGS0069 | + | ID FL5FACGS0069 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES c(c5O)c(cc(c53)OC(c(c4)ccc(O)c4O)=C(C3=O)OC(C(OC(O2)C(C(O)C(C2CO)O)O)1)OC(C)C(O)C1O)O | + | SMILES c(c5O)c(cc(c53)OC(c(c4)ccc(O)c4O)=C(C3=O)OC(C(OC(O2)C(C(O)C(C2CO)O)O)1)OC(C)C(O)C1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
1.1147 3.7227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1147 2.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8290 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5435 2.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5435 3.7227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8290 4.1352 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4002 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3143 2.8977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0288 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0288 1.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3143 1.2478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4002 1.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7431 2.8977 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4576 2.4853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4576 1.6603 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7431 1.2478 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3143 0.4228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1721 2.8977 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1147 1.2478 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7431 0.4228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1354 4.0645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1721 2.5349 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1649 0.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5585 -0.5322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2288 -0.2846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0322 -0.4727 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4259 0.2525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6386 0.0051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0226 -1.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1884 -0.8921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9946 -0.3675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8123 0.0322 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1265 -3.6690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9142 -3.4226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8845 -2.5979 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2792 -1.8732 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4916 -2.1196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5212 -2.9444 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0605 -2.8599 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6662 -3.1551 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1415 -3.2957 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7064 -4.1352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5822 -2.1802 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
26 29 1 0 0 0 0
25 30 1 0 0 0 0
24 31 1 0 0 0 0
23 32 1 0 0 0 0
27 19 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 29 1 0 0 0 0
S SKP 5
ID FL5FACGS0069
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES c(c5O)c(cc(c53)OC(c(c4)ccc(O)c4O)=C(C3=O)OC(C(OC(O2)C(C(O)C(C2CO)O)O)1)OC(C)C(O)C1O)O
M END