Mol:FL5FACGS0067
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.1147 3.3712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1147 3.3712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1147 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1147 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8290 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8290 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5435 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5435 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5435 3.3712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5435 3.3712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8290 3.7837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8290 3.7837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4002 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4002 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3143 2.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3143 2.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0288 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0288 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0288 1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0288 1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3143 0.8962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3143 0.8962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4002 1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4002 1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7431 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7431 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4576 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4576 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4576 1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4576 1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7431 0.8962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7431 0.8962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3143 0.0713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3143 0.0713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1721 2.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1721 2.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1147 0.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1147 0.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7431 0.0713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7431 0.0713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1354 3.7130 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1354 3.7130 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1721 2.1833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1721 2.1833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1443 -0.2204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1443 -0.2204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5379 -0.9457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5379 -0.9457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2494 -0.6981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2494 -0.6981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0529 -0.8862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0529 -0.8862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4465 -0.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4465 -0.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6593 -0.4084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6593 -0.4084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0638 -1.5796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0638 -1.5796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2090 -1.2849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2090 -1.2849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9740 -0.7809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9740 -0.7809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8535 -0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8535 -0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3502 -2.4450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3502 -2.4450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7438 -3.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7438 -3.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0434 -2.9227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0434 -2.9227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8469 -3.1108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8469 -3.1108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2406 -2.3855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2406 -2.3855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4533 -2.6330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4533 -2.6330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9198 -3.7837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9198 -3.7837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4562 -3.5301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4562 -3.5301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1799 -3.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1799 -3.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9976 -2.5852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9976 -2.5852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 16 20 1 0 0 0 0 | + | 16 20 1 0 0 0 0 |
| − | 5 21 1 0 0 0 0 | + | 5 21 1 0 0 0 0 |
| − | 4 22 1 0 0 0 0 | + | 4 22 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 27 26 1 1 0 0 0 | + | 27 26 1 1 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 23 32 1 0 0 0 0 | + | 23 32 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 37 29 1 0 0 0 0 | + | 37 29 1 0 0 0 0 |
| − | 27 19 1 0 0 0 0 | + | 27 19 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
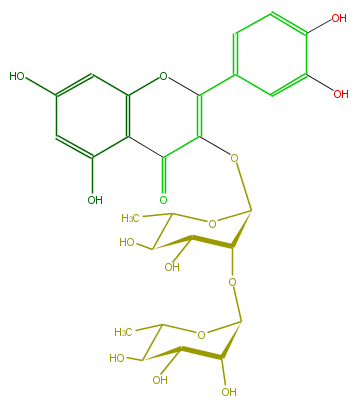
| − | ID FL5FACGS0067 | + | ID FL5FACGS0067 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES c(c5O)c(cc(c53)OC(c(c4)ccc(O)c4O)=C(C3=O)OC(C1OC(O2)C(C(O)C(O)C(C)2)O)OC(C)C(C1O)O)O | + | SMILES c(c5O)c(cc(c53)OC(c(c4)ccc(O)c4O)=C(C3=O)OC(C1OC(O2)C(C(O)C(O)C(C)2)O)OC(C)C(C1O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
1.1147 3.3712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1147 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8290 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5435 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5435 3.3712 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8290 3.7837 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4002 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3143 2.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0288 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0288 1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3143 0.8962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4002 1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7431 2.5462 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4576 2.1337 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4576 1.3087 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7431 0.8962 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3143 0.0713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1721 2.5462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1147 0.8962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7431 0.0713 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1354 3.7130 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1721 2.1833 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1443 -0.2204 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5379 -0.9457 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2494 -0.6981 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0529 -0.8862 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4465 -0.1609 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6593 -0.4084 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0638 -1.5796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2090 -1.2849 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9740 -0.7809 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8535 -0.2987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3502 -2.4450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7438 -3.1703 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0434 -2.9227 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8469 -3.1108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2406 -2.3855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4533 -2.6330 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9198 -3.7837 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4562 -3.5301 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1799 -3.0881 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9976 -2.5852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
16 20 1 0 0 0 0
5 21 1 0 0 0 0
4 22 1 0 0 0 0
23 24 1 0 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
27 26 1 1 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
26 29 1 0 0 0 0
25 30 1 0 0 0 0
24 31 1 0 0 0 0
23 32 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
34 41 1 0 0 0 0
33 42 1 0 0 0 0
37 29 1 0 0 0 0
27 19 1 0 0 0 0
S SKP 5
ID FL5FACGS0067
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES c(c5O)c(cc(c53)OC(c(c4)ccc(O)c4O)=C(C3=O)OC(C1OC(O2)C(C(O)C(O)C(C)2)O)OC(C)C(C1O)O)O
M END