Mol:FL5FACGS0059
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 36 39 0 0 0 0 0 0 0 0999 V2000 | + | 36 39 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.3722 -1.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3722 -1.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3722 -1.9270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3722 -1.9270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8159 -2.2482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8159 -2.2482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2596 -1.9270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2596 -1.9270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2596 -1.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2596 -1.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8159 -0.9635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8159 -0.9635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2967 -2.2482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2967 -2.2482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8530 -1.9270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8530 -1.9270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8530 -1.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8530 -1.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2967 -0.9635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2967 -0.9635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2967 -2.7490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2967 -2.7490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5535 -0.8398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5535 -0.8398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1205 -1.1672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1205 -1.1672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6875 -0.8398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6875 -0.8398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6875 -0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6875 -0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1205 0.1422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1205 0.1422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5535 -0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5535 -0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0367 -0.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0367 -0.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8159 -2.8109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8159 -2.8109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4370 -2.3159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4370 -2.3159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6458 -1.2527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6458 -1.2527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6458 -1.9553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6458 -1.9553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2543 -0.9014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2543 -0.9014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1205 0.7967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1205 0.7967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2543 0.1421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2543 0.1421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9111 2.4612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9111 2.4612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6724 1.8948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6724 1.8948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1196 1.5359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1196 1.5359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3823 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3823 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5196 1.6040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5196 1.6040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0720 1.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0720 1.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3972 2.7579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3972 2.7579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3587 2.3818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3587 2.3818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8275 1.0136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8275 1.0136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2005 2.8109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2005 2.8109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9974 2.5974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9974 2.5974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 3 1 0 0 0 0 | + | 19 3 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 18 21 1 0 0 0 0 | + | 18 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 21 23 2 0 0 0 0 | + | 21 23 2 0 0 0 0 |
| − | 16 24 1 0 0 0 0 | + | 16 24 1 0 0 0 0 |
| − | 15 25 1 0 0 0 0 | + | 15 25 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 29 28 1 1 0 0 0 | + | 29 28 1 1 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 26 1 0 0 0 0 | + | 31 26 1 0 0 0 0 |
| − | 26 32 1 0 0 0 0 | + | 26 32 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 24 1 0 0 0 0 | + | 29 24 1 0 0 0 0 |
| − | 31 35 1 0 0 0 0 | + | 31 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 35 36 | + | M SAL 1 2 35 36 |
| − | M SBL 1 1 38 | + | M SBL 1 1 38 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 38 -6.7877 6.0963 | + | M SBV 1 38 -6.7877 6.0963 |
| − | S SKP 8 | + | S SKP 8 |
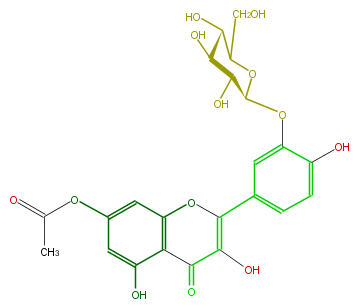
| − | ID FL5FACGS0059 | + | ID FL5FACGS0059 |
| − | KNApSAcK_ID C00006062 | + | KNApSAcK_ID C00006062 |
| − | NAME Quercetin 3'-glucoside-7-acetate | + | NAME Quercetin 3'-glucoside-7-acetate |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C23H22O13 | + | FORMULA C23H22O13 |
| − | EXACTMASS 506.10604078999995 | + | EXACTMASS 506.10604078999995 |
| − | AVERAGEMASS 506.41298000000006 | + | AVERAGEMASS 506.41298000000006 |
| − | SMILES OC(C4O)C(OC(CO)C4O)Oc(c1)c(O)ccc1C(=C3O)Oc(c2)c(C3=O)c(O)cc2OC(C)=O | + | SMILES OC(C4O)C(OC(CO)C4O)Oc(c1)c(O)ccc1C(=C3O)Oc(c2)c(C3=O)c(O)cc2OC(C)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
36 39 0 0 0 0 0 0 0 0999 V2000
-1.3722 -1.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3722 -1.9270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8159 -2.2482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2596 -1.9270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2596 -1.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8159 -0.9635 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2967 -2.2482 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8530 -1.9270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8530 -1.2847 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2967 -0.9635 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2967 -2.7490 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5535 -0.8398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1205 -1.1672 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6875 -0.8398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6875 -0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1205 0.1422 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5535 -0.1852 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0367 -0.9010 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8159 -2.8109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4370 -2.3159 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6458 -1.2527 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6458 -1.9553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2543 -0.9014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1205 0.7967 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2543 0.1421 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9111 2.4612 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6724 1.8948 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1196 1.5359 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3823 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5196 1.6040 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0720 1.8855 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3972 2.7579 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3587 2.3818 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8275 1.0136 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2005 2.8109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9974 2.5974 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
4 3 1 0 0 0 0
1 18 1 0 0 0 0
19 3 1 0 0 0 0
8 20 1 0 0 0 0
18 21 1 0 0 0 0
21 22 1 0 0 0 0
21 23 2 0 0 0 0
16 24 1 0 0 0 0
15 25 1 0 0 0 0
26 27 1 1 0 0 0
27 28 1 1 0 0 0
29 28 1 1 0 0 0
29 30 1 0 0 0 0
30 31 1 0 0 0 0
31 26 1 0 0 0 0
26 32 1 0 0 0 0
27 33 1 0 0 0 0
28 34 1 0 0 0 0
29 24 1 0 0 0 0
31 35 1 0 0 0 0
35 36 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 35 36
M SBL 1 1 38
M SMT 1 CH2OH
M SBV 1 38 -6.7877 6.0963
S SKP 8
ID FL5FACGS0059
KNApSAcK_ID C00006062
NAME Quercetin 3'-glucoside-7-acetate
CAS_RN -
FORMULA C23H22O13
EXACTMASS 506.10604078999995
AVERAGEMASS 506.41298000000006
SMILES OC(C4O)C(OC(CO)C4O)Oc(c1)c(O)ccc1C(=C3O)Oc(c2)c(C3=O)c(O)cc2OC(C)=O
M END