Mol:FL5FACGS0055
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 65 71 0 0 0 0 0 0 0 0999 V2000 | + | 65 71 0 0 0 0 0 0 0 0999 V2000 |
| − | -4.0026 -0.8474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0026 -0.8474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0026 -1.4898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0026 -1.4898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4463 -1.8109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4463 -1.8109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8900 -1.4898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8900 -1.4898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8900 -0.8474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8900 -0.8474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4463 -0.5262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4463 -0.5262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3337 -1.8109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3337 -1.8109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7774 -1.4898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7774 -1.4898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7774 -0.8474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7774 -0.8474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3337 -0.5262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3337 -0.5262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3337 -2.3118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3337 -2.3118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2213 -0.5263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2213 -0.5263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6543 -0.8537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6543 -0.8537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0874 -0.5263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0874 -0.5263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0874 0.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0874 0.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6543 0.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6543 0.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2213 0.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2213 0.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5587 -0.5263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5587 -0.5263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4795 0.4556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4795 0.4556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3832 -1.8786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3832 -1.8786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4463 -2.4531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4463 -2.4531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6515 -0.9775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6515 -0.9775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0493 -0.4092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0493 -0.4092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6236 -0.4594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6236 -0.4594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7564 0.2188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7564 0.2188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0820 0.6838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0820 0.6838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8426 1.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8426 1.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1479 1.6332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1479 1.6332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9230 2.1155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9230 2.1155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3928 2.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3928 2.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0875 1.7259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0875 1.7259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3125 1.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3125 1.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1679 2.6441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1679 2.6441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6543 1.1102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6543 1.1102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5945 -2.9205 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -0.5945 -2.9205 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.0591 -3.2296 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0591 -3.2296 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.2290 -2.6352 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -0.2290 -2.6352 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.0591 -2.0372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0591 -2.0372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5945 -1.7280 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5945 -1.7280 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.4247 -2.3225 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.4247 -2.3225 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 0.2318 -2.3225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2318 -2.3225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9703 -3.2962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9703 -3.2962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0276 -3.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0276 -3.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2996 -2.9404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2996 -2.9404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1816 -1.9964 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 1.1816 -1.9964 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 1.5833 -2.5266 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5833 -2.5266 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.1617 -2.3016 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.1617 -2.3016 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.7199 -2.2956 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.7199 -2.2956 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3143 -1.8899 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.3143 -1.8899 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.8082 -2.1570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8082 -2.1570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9570 -2.5571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9570 -2.5571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4932 -2.8580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4932 -2.8580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7850 -1.6704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7850 -1.6704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1119 -2.3647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1119 -2.3647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1615 3.1156 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 0.1615 3.1156 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 0.4886 2.5951 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 0.4886 2.5951 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.0391 2.7556 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.0391 2.7556 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.5535 2.7162 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 1.5535 2.7162 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.2128 3.1223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2128 3.1223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7255 2.9172 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 0.7255 2.9172 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -0.4226 3.2186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4226 3.2186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0994 2.1660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0994 2.1660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2995 2.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2995 2.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7541 3.3277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7541 3.3277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0751 3.8867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0751 3.8867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 23 25 1 0 0 0 0 | + | 23 25 1 0 0 0 0 |
| − | 25 26 2 0 0 0 0 | + | 25 26 2 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 2 0 0 0 0 | + | 27 28 2 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 2 0 0 0 0 | + | 29 30 2 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 30 33 1 0 0 0 0 | + | 30 33 1 0 0 0 0 |
| − | 16 34 1 0 0 0 0 | + | 16 34 1 0 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 1 0 0 0 | + | 39 40 1 1 0 0 0 |
| − | 40 35 1 1 0 0 0 | + | 40 35 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 35 42 1 0 0 0 0 | + | 35 42 1 0 0 0 0 |
| − | 36 43 1 0 0 0 0 | + | 36 43 1 0 0 0 0 |
| − | 37 44 1 0 0 0 0 | + | 37 44 1 0 0 0 0 |
| − | 39 20 1 0 0 0 0 | + | 39 20 1 0 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 48 47 1 1 0 0 0 | + | 48 47 1 1 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 46 51 1 0 0 0 0 | + | 46 51 1 0 0 0 0 |
| − | 47 52 1 0 0 0 0 | + | 47 52 1 0 0 0 0 |
| − | 49 53 1 0 0 0 0 | + | 49 53 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 41 45 1 0 0 0 0 | + | 41 45 1 0 0 0 0 |
| − | 53 22 1 0 0 0 0 | + | 53 22 1 0 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 58 57 1 1 0 0 0 | + | 58 57 1 1 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 55 1 0 0 0 0 | + | 60 55 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 56 62 1 0 0 0 0 | + | 56 62 1 0 0 0 0 |
| − | 57 63 1 0 0 0 0 | + | 57 63 1 0 0 0 0 |
| − | 58 33 1 0 0 0 0 | + | 58 33 1 0 0 0 0 |
| − | 60 64 1 0 0 0 0 | + | 60 64 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 64 65 | + | M SAL 1 2 64 65 |
| − | M SBL 1 1 70 | + | M SBL 1 1 70 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 70 3.1053 3.4982 | + | M SVB 1 70 3.1053 3.4982 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FACGS0055 | + | ID FL5FACGS0055 |
| − | KNApSAcK_ID C00005979 | + | KNApSAcK_ID C00005979 |
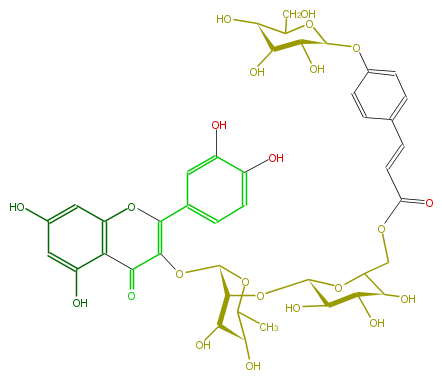
| − | NAME Quercetin 3-(6-[4-glucosyl-p-coumaryl]glucosyl)(1->2)-rhamnoside | + | NAME Quercetin 3-(6-[4-glucosyl-p-coumaryl]glucosyl)(1->2)-rhamnoside |
| − | CAS_RN 142997-33-9 | + | CAS_RN 142997-33-9 |
| − | FORMULA C42H46O23 | + | FORMULA C42H46O23 |
| − | EXACTMASS 918.242987778 | + | EXACTMASS 918.242987778 |
| − | AVERAGEMASS 918.8008399999999 | + | AVERAGEMASS 918.8008399999999 |
| − | SMILES C([C@H]5O)(COC(=O)C=Cc(c7)ccc(c7)O[C@H](O6)[C@H]([C@@H](O)[C@@H](O)C6CO)O)O[C@@H]([C@H](O)[C@@H]5O)O[C@@H]([C@H](O)4)[C@H](OC(C)[C@H]4O)OC(=C2c(c3)cc(c(O)c3)O)C(c(c1O2)c(O)cc(c1)O)=O | + | SMILES C([C@H]5O)(COC(=O)C=Cc(c7)ccc(c7)O[C@H](O6)[C@H]([C@@H](O)[C@@H](O)C6CO)O)O[C@@H]([C@H](O)[C@@H]5O)O[C@@H]([C@H](O)4)[C@H](OC(C)[C@H]4O)OC(=C2c(c3)cc(c(O)c3)O)C(c(c1O2)c(O)cc(c1)O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
65 71 0 0 0 0 0 0 0 0999 V2000
-4.0026 -0.8474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0026 -1.4898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4463 -1.8109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8900 -1.4898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8900 -0.8474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4463 -0.5262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3337 -1.8109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7774 -1.4898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7774 -0.8474 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3337 -0.5262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3337 -2.3118 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2213 -0.5263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6543 -0.8537 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0874 -0.5263 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0874 0.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6543 0.4557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2213 0.1284 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5587 -0.5263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4795 0.4556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3832 -1.8786 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4463 -2.4531 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6515 -0.9775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0493 -0.4092 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6236 -0.4594 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7564 0.2188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0820 0.6838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8426 1.1972 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1479 1.6332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.9230 2.1155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3928 2.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0875 1.7259 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3125 1.2436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1679 2.6441 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6543 1.1102 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5945 -2.9205 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.0591 -3.2296 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.2290 -2.6352 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.0591 -2.0372 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5945 -1.7280 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.4247 -2.3225 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
0.2318 -2.3225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9703 -3.2962 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0276 -3.7211 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2996 -2.9404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1816 -1.9964 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
1.5833 -2.5266 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.1617 -2.3016 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.7199 -2.2956 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3143 -1.8899 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.8082 -2.1570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9570 -2.5571 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4932 -2.8580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7850 -1.6704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1119 -2.3647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1615 3.1156 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
0.4886 2.5951 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.0391 2.7556 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.5535 2.7162 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.2128 3.1223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7255 2.9172 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-0.4226 3.2186 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0994 2.1660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2995 2.2168 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7541 3.3277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0751 3.8867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
23 25 1 0 0 0 0
25 26 2 0 0 0 0
26 27 1 0 0 0 0
27 28 2 0 0 0 0
28 29 1 0 0 0 0
29 30 2 0 0 0 0
30 31 1 0 0 0 0
31 32 2 0 0 0 0
32 27 1 0 0 0 0
30 33 1 0 0 0 0
16 34 1 0 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 1 0 0 0
40 35 1 1 0 0 0
40 41 1 0 0 0 0
35 42 1 0 0 0 0
36 43 1 0 0 0 0
37 44 1 0 0 0 0
39 20 1 0 0 0 0
45 46 1 1 0 0 0
46 47 1 1 0 0 0
48 47 1 1 0 0 0
48 49 1 0 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
46 51 1 0 0 0 0
47 52 1 0 0 0 0
49 53 1 0 0 0 0
48 54 1 0 0 0 0
41 45 1 0 0 0 0
53 22 1 0 0 0 0
55 56 1 1 0 0 0
56 57 1 1 0 0 0
58 57 1 1 0 0 0
58 59 1 0 0 0 0
59 60 1 0 0 0 0
60 55 1 0 0 0 0
55 61 1 0 0 0 0
56 62 1 0 0 0 0
57 63 1 0 0 0 0
58 33 1 0 0 0 0
60 64 1 0 0 0 0
64 65 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 64 65
M SBL 1 1 70
M SMT 1 CH2OH
M SVB 1 70 3.1053 3.4982
S SKP 8
ID FL5FACGS0055
KNApSAcK_ID C00005979
NAME Quercetin 3-(6-[4-glucosyl-p-coumaryl]glucosyl)(1->2)-rhamnoside
CAS_RN 142997-33-9
FORMULA C42H46O23
EXACTMASS 918.242987778
AVERAGEMASS 918.8008399999999
SMILES C([C@H]5O)(COC(=O)C=Cc(c7)ccc(c7)O[C@H](O6)[C@H]([C@@H](O)[C@@H](O)C6CO)O)O[C@@H]([C@H](O)[C@@H]5O)O[C@@H]([C@H](O)4)[C@H](OC(C)[C@H]4O)OC(=C2c(c3)cc(c(O)c3)O)C(c(c1O2)c(O)cc(c1)O)=O
M END