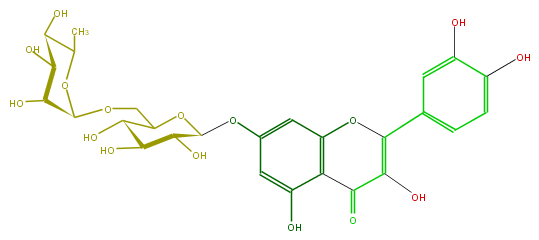
Mol:FL5FACGS0038
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.2244 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2244 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2244 -1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2244 -1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4904 -1.6533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4904 -1.6533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2052 -1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2052 -1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2052 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2052 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4904 -0.0027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4904 -0.0027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9199 -1.6533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9199 -1.6533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6347 -1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6347 -1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6347 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6347 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9199 -0.0027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9199 -0.0027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9199 -2.2968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9199 -2.2968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5346 0.1562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5346 0.1562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2631 -0.2644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2631 -0.2644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9916 0.1562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9916 0.1562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9916 0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9916 0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2631 1.4180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2631 1.4180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5346 0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5346 0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4904 -2.4783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4904 -2.4783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7713 1.4475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7713 1.4475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8603 -0.0027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8603 -0.0027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3584 -1.7668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3584 -1.7668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2631 2.2589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2631 2.2589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4021 -0.0157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4021 -0.0157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9252 -0.6452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9252 -0.6452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2384 -0.3781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2384 -0.3781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5757 -0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5757 -0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0573 0.1106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0573 0.1106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6581 -0.2065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6581 -0.2065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0623 -0.3969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0623 -0.3969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6686 -0.6813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6686 -0.6813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7184 -0.8145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7184 -0.8145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1972 0.4453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1972 0.4453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5159 0.0519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5159 0.0519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7320 0.8082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7320 0.8082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5159 1.5693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5159 1.5693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1972 1.9627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1972 1.9627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9810 1.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9810 1.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9262 2.4783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9262 2.4783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5159 2.0601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5159 2.0601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5718 1.6371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5718 1.6371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7713 0.3927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7713 0.3927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0805 0.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0805 0.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7438 0.2558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7438 0.2558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 20 1 1 0 0 0 0 | + | 20 1 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 16 22 1 0 0 0 0 | + | 16 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 25 31 1 0 0 0 0 | + | 25 31 1 0 0 0 0 |
| − | 26 20 1 0 0 0 0 | + | 26 20 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 36 38 1 0 0 0 0 | + | 36 38 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 28 42 1 0 0 0 0 | + | 28 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 33 1 0 0 0 0 | + | 43 33 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGS0038 | + | ID FL5FACGS0038 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES O(C1C)C(OCC(O2)C(O)C(C(C(Oc(c3)cc(O)c(C4=O)c3OC(c(c5)ccc(c5O)O)=C4O)2)O)O)C(C(O)C(O)1)O | + | SMILES O(C1C)C(OCC(O2)C(O)C(C(C(Oc(c3)cc(O)c(C4=O)c3OC(c(c5)ccc(c5O)O)=C4O)2)O)O)C(C(O)C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
-0.2244 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2244 -1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4904 -1.6533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2052 -1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2052 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4904 -0.0027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9199 -1.6533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6347 -1.2407 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6347 -0.4154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9199 -0.0027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9199 -2.2968 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5346 0.1562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2631 -0.2644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9916 0.1562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9916 0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2631 1.4180 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5346 0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4904 -2.4783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.7713 1.4475 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8603 -0.0027 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3584 -1.7668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2631 2.2589 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4021 -0.0157 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9252 -0.6452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2384 -0.3781 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5757 -0.3709 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0573 0.1106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6581 -0.2065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0623 -0.3969 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6686 -0.6813 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7184 -0.8145 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1972 0.4453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5159 0.0519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7320 0.8082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5159 1.5693 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1972 1.9627 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9810 1.2062 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9262 2.4783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5159 2.0601 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5718 1.6371 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.7713 0.3927 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0805 0.2572 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7438 0.2558 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
20 1 1 0 0 0 0
21 8 1 0 0 0 0
4 3 1 0 0 0 0
16 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
23 29 1 0 0 0 0
24 30 1 0 0 0 0
25 31 1 0 0 0 0
26 20 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
36 38 1 0 0 0 0
35 39 1 0 0 0 0
37 40 1 0 0 0 0
32 41 1 0 0 0 0
28 42 1 0 0 0 0
42 43 1 0 0 0 0
43 33 1 0 0 0 0
S SKP 5
ID FL5FACGS0038
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES O(C1C)C(OCC(O2)C(O)C(C(C(Oc(c3)cc(O)c(C4=O)c3OC(c(c5)ccc(c5O)O)=C4O)2)O)O)C(C(O)C(O)1)O
M END