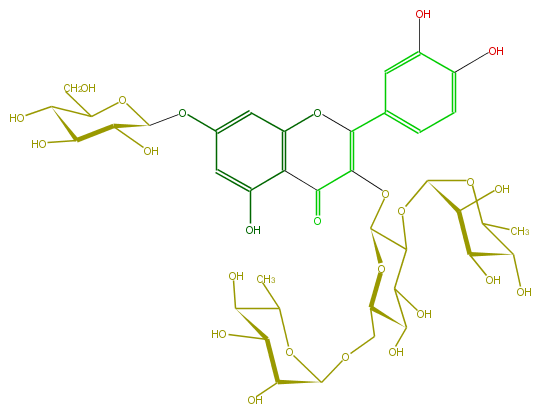
Mol:FL5FACGL0051
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 64 70 0 0 0 0 0 0 0 0999 V2000 | + | 64 70 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.1296 1.5509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1296 1.5509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1296 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1296 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4286 0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4286 0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2724 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2724 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2724 1.5509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2724 1.5509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4286 1.9556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4286 1.9556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9735 0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9735 0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6745 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6745 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6745 1.5509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6745 1.5509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9735 1.9556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9735 1.9556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9735 -0.2943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9735 -0.2943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3752 1.9555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3752 1.9555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0896 1.5431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0896 1.5431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8041 1.9555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8041 1.9555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8041 2.7805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8041 2.7805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0896 3.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0896 3.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3752 2.7805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3752 2.7805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3993 0.2707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3993 0.2707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4286 -0.4724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4286 -0.4724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6029 3.2417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6029 3.2417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8077 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8077 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0733 -0.4650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0733 -0.4650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3063 -1.2803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3063 -1.2803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0733 -2.1007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0733 -2.1007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8077 -2.5248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8077 -2.5248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5747 -1.7094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5747 -1.7094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7219 -0.0822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7219 -0.0822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1164 -2.2321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1164 -2.2321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5369 -3.0106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5369 -3.0106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0896 4.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0896 4.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8304 1.9555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8304 1.9555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1295 -0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1295 -0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0436 -0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0436 -0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3866 -0.3618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3866 -0.3618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1620 0.5296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1620 0.5296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2479 0.5298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2479 0.5298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9048 -0.1060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9048 -0.1060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7456 0.3794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7456 0.3794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5421 -1.5130 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5421 -1.5130 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1948 -1.7734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1948 -1.7734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0545 -0.4957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0545 -0.4957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1419 -3.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1419 -3.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0562 -3.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0562 -3.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3991 -3.0151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3991 -3.0151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1744 -2.1236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1744 -2.1236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7397 -2.1235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7397 -2.1235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0828 -2.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0828 -2.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7803 -1.4351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7803 -1.4351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2279 -1.4996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2279 -1.4996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9982 -2.6434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9982 -2.6434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3875 -4.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3875 -4.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5598 -3.1262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5598 -3.1262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1475 -2.7010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1475 -2.7010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5678 2.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5678 2.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0369 1.3721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0369 1.3721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2725 1.6694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2725 1.6694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5349 1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5349 1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0709 2.2135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0709 2.2135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7396 1.8606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7396 1.8606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1948 1.8504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1948 1.8504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7429 1.3091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7429 1.3091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5622 1.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5622 1.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3368 2.4695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3368 2.4695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4653 3.3797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4653 3.3797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 18 1 0 0 0 0 | + | 22 18 1 0 0 0 0 |
| − | 16 30 1 0 0 0 0 | + | 16 30 1 0 0 0 0 |
| − | 1 31 1 0 0 0 0 | + | 1 31 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 36 27 1 0 0 0 0 | + | 36 27 1 0 0 0 0 |
| − | 43 42 1 1 0 0 0 | + | 43 42 1 1 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 47 42 1 1 0 0 0 | + | 47 42 1 1 0 0 0 |
| − | 46 48 1 0 0 0 0 | + | 46 48 1 0 0 0 0 |
| − | 45 49 1 0 0 0 0 | + | 45 49 1 0 0 0 0 |
| − | 47 50 1 0 0 0 0 | + | 47 50 1 0 0 0 0 |
| − | 42 51 1 0 0 0 0 | + | 42 51 1 0 0 0 0 |
| − | 43 52 1 0 0 0 0 | + | 43 52 1 0 0 0 0 |
| − | 24 53 1 0 0 0 0 | + | 24 53 1 0 0 0 0 |
| − | 53 52 1 0 0 0 0 | + | 53 52 1 0 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 57 56 1 1 0 0 0 | + | 57 56 1 1 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 54 1 0 0 0 0 | + | 59 54 1 0 0 0 0 |
| − | 54 60 1 0 0 0 0 | + | 54 60 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 56 62 1 0 0 0 0 | + | 56 62 1 0 0 0 0 |
| − | 57 31 1 0 0 0 0 | + | 57 31 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 59 63 1 0 0 0 0 | + | 59 63 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 63 64 | + | M SAL 1 2 63 64 |
| − | M SBL 1 1 70 | + | M SBL 1 1 70 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 70 0.5972 -0.6090 | + | M SBV 1 70 0.5972 -0.6090 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGL0051 | + | ID FL5FACGL0051 |
| − | FORMULA C39H50O25 | + | FORMULA C39H50O25 |
| − | EXACTMASS 918.26411715 | + | EXACTMASS 918.26411715 |
| − | AVERAGEMASS 918.7993 | + | AVERAGEMASS 918.7993 |
| − | SMILES c(c31)c(OC(C7O)OC(CO)C(C7O)O)cc(O)c(C(C(OC(O4)C(OC(C6O)OC(C)C(C(O)6)O)C(C(O)C(COC(O5)C(C(O)C(O)C5C)O)4)O)=C(O3)c(c2)cc(O)c(c2)O)=O)1 | + | SMILES c(c31)c(OC(C7O)OC(CO)C(C7O)O)cc(O)c(C(C(OC(O4)C(OC(C6O)OC(C)C(C(O)6)O)C(C(O)C(COC(O5)C(C(O)C(O)C5C)O)4)O)=C(O3)c(c2)cc(O)c(c2)O)=O)1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
64 70 0 0 0 0 0 0 0 0999 V2000
-1.1296 1.5509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1296 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4286 0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2724 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2724 1.5509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4286 1.9556 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9735 0.3367 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6745 0.7415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6745 1.5509 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9735 1.9556 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9735 -0.2943 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3752 1.9555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0896 1.5431 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8041 1.9555 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8041 2.7805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0896 3.1930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3752 2.7805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3993 0.2707 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4286 -0.4724 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6029 3.2417 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8077 -0.8890 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0733 -0.4650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3063 -1.2803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0733 -2.1007 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8077 -2.5248 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5747 -1.7094 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7219 -0.0822 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1164 -2.2321 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5369 -3.0106 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0896 4.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8304 1.9555 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1295 -0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.0436 -0.9973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3866 -0.3618 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1620 0.5296 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.2479 0.5298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9048 -0.1060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7456 0.3794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5421 -1.5130 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1948 -1.7734 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0545 -0.4957 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1419 -3.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0562 -3.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3991 -3.0151 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1744 -2.1236 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7397 -2.1235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0828 -2.7593 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7803 -1.4351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2279 -1.4996 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9982 -2.6434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3875 -4.0177 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5598 -3.1262 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1475 -2.7010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5678 2.0729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0369 1.3721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2725 1.6694 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5349 1.6773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0709 2.2135 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7396 1.8606 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1948 1.8504 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7429 1.3091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5622 1.1568 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3368 2.4695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4653 3.3797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
8 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 18 1 0 0 0 0
16 30 1 0 0 0 0
1 31 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
37 38 1 0 0 0 0
32 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
36 27 1 0 0 0 0
43 42 1 1 0 0 0
43 44 1 0 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
47 42 1 1 0 0 0
46 48 1 0 0 0 0
45 49 1 0 0 0 0
47 50 1 0 0 0 0
42 51 1 0 0 0 0
43 52 1 0 0 0 0
24 53 1 0 0 0 0
53 52 1 0 0 0 0
54 55 1 1 0 0 0
55 56 1 1 0 0 0
57 56 1 1 0 0 0
57 58 1 0 0 0 0
58 59 1 0 0 0 0
59 54 1 0 0 0 0
54 60 1 0 0 0 0
55 61 1 0 0 0 0
56 62 1 0 0 0 0
57 31 1 0 0 0 0
63 64 1 0 0 0 0
59 63 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 63 64
M SBL 1 1 70
M SMT 1 ^ CH2OH
M SBV 1 70 0.5972 -0.6090
S SKP 5
ID FL5FACGL0051
FORMULA C39H50O25
EXACTMASS 918.26411715
AVERAGEMASS 918.7993
SMILES c(c31)c(OC(C7O)OC(CO)C(C7O)O)cc(O)c(C(C(OC(O4)C(OC(C6O)OC(C)C(C(O)6)O)C(C(O)C(COC(O5)C(C(O)C(O)C5C)O)4)O)=C(O3)c(c2)cc(O)c(c2)O)=O)1
M END