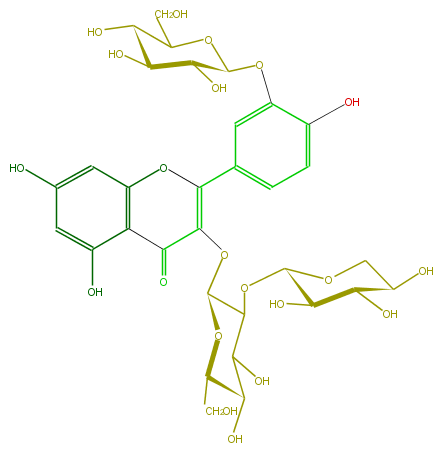
Mol:FL5FACGL0044
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3057 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3057 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3057 -0.5937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3057 -0.5937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5912 -1.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5912 -1.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8768 -0.5937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8768 -0.5937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8768 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8768 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5912 0.6437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5912 0.6437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1623 -1.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1623 -1.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4478 -0.5937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4478 -0.5937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4478 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4478 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1623 0.6437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1623 0.6437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1623 -1.6494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1623 -1.6494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2663 0.6436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2663 0.6436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9945 0.2232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9945 0.2232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7227 0.6436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7227 0.6436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7227 1.4844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7227 1.4844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9945 1.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9945 1.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2663 1.4844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2663 1.4844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0585 -1.0932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0585 -1.0932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5912 -1.8309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5912 -1.8309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5369 1.9545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5369 1.9545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4550 -2.3202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4550 -2.3202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2934 -1.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2934 -1.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0560 -2.7190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0560 -2.7190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2934 -3.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2934 -3.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4550 -3.9874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4550 -3.9874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2175 -3.1563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2175 -3.1563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4515 -1.7695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4515 -1.7695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7486 -3.6259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7486 -3.6259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1993 -4.7797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1993 -4.7797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2580 -1.3818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2580 -1.3818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8261 -2.1318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8261 -2.1318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6443 -1.8136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6443 -1.8136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4339 -1.8050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4339 -1.8050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8602 -1.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8602 -1.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1443 -1.6091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1443 -1.6091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1744 -2.0727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1744 -2.0727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3037 -2.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3037 -2.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0199 -1.4292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0199 -1.4292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7670 2.7017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7670 2.7017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0199 0.6436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0199 0.6436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7767 3.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7767 3.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4389 2.6355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4389 2.6355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6080 2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6080 2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1203 2.5398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1203 2.5398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2660 3.2089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2660 3.2089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0175 3.0379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0175 3.0379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4632 3.3660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4632 3.3660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0488 2.9189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0488 2.9189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1174 2.2489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1174 2.2489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3663 3.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3663 3.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1030 4.7797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1030 4.7797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3506 -4.2246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3506 -4.2246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6271 -4.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6271 -4.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 8 18 1 0 0 0 0 | + | 8 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 22 18 1 0 0 0 0 | + | 22 18 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 16 39 1 0 0 0 0 | + | 16 39 1 0 0 0 0 |
| − | 1 40 1 0 0 0 0 | + | 1 40 1 0 0 0 0 |
| − | 41 42 1 1 0 0 0 | + | 41 42 1 1 0 0 0 |
| − | 42 43 1 1 0 0 0 | + | 42 43 1 1 0 0 0 |
| − | 44 43 1 1 0 0 0 | + | 44 43 1 1 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 41 1 0 0 0 0 | + | 46 41 1 0 0 0 0 |
| − | 41 47 1 0 0 0 0 | + | 41 47 1 0 0 0 0 |
| − | 42 48 1 0 0 0 0 | + | 42 48 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 39 1 0 0 0 0 | + | 44 39 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 46 50 1 0 0 0 0 | + | 46 50 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 24 52 1 0 0 0 0 | + | 24 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 56 0.3488 -0.6899 | + | M SBV 1 56 0.3488 -0.6899 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 52 53 | + | M SAL 2 2 52 53 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 58 0.0572 0.6696 | + | M SBV 2 58 0.0572 0.6696 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGL0044 | + | ID FL5FACGL0044 |
| − | FORMULA C32H38O21 | + | FORMULA C32H38O21 |
| − | EXACTMASS 758.190558278 | + | EXACTMASS 758.190558278 |
| − | AVERAGEMASS 758.6315199999999 | + | AVERAGEMASS 758.6315199999999 |
| − | SMILES c(c(O)6)c(c(c1c6)C(C(OC(C4OC(O5)C(C(O)C(O)C5)O)OC(C(O)C(O)4)CO)=C(c(c2)ccc(c(OC(O3)C(O)C(O)C(C3CO)O)2)O)O1)=O)O | + | SMILES c(c(O)6)c(c(c1c6)C(C(OC(C4OC(O5)C(C(O)C(O)C5)O)OC(C(O)C(O)4)CO)=C(c(c2)ccc(c(OC(O3)C(O)C(O)C(C3CO)O)2)O)O1)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-3.3057 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3057 -0.5937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5912 -1.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8768 -0.5937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8768 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5912 0.6437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1623 -1.0063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4478 -0.5937 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4478 0.2311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1623 0.6437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1623 -1.6494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2663 0.6436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9945 0.2232 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7227 0.6436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7227 1.4844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9945 1.9048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2663 1.4844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0585 -1.0932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5912 -1.8309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5369 1.9545 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4550 -2.3202 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2934 -1.8881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0560 -2.7190 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2934 -3.5551 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4550 -3.9874 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2175 -3.1563 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4515 -1.7695 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7486 -3.6259 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1993 -4.7797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2580 -1.3818 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8261 -2.1318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6443 -1.8136 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4339 -1.8050 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8602 -1.2313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1443 -1.6091 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1744 -2.0727 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3037 -2.2867 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0199 -1.4292 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7670 2.7017 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0199 0.6436 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7767 3.4654 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4389 2.6355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6080 2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1203 2.5398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2660 3.2089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0175 3.0379 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4632 3.3660 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0488 2.9189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1174 2.2489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3663 3.7277 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1030 4.7797 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3506 -4.2246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6271 -4.0830 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
8 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
22 18 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
31 36 1 0 0 0 0
32 37 1 0 0 0 0
27 30 1 0 0 0 0
33 38 1 0 0 0 0
16 39 1 0 0 0 0
1 40 1 0 0 0 0
41 42 1 1 0 0 0
42 43 1 1 0 0 0
44 43 1 1 0 0 0
44 45 1 0 0 0 0
45 46 1 0 0 0 0
46 41 1 0 0 0 0
41 47 1 0 0 0 0
42 48 1 0 0 0 0
43 49 1 0 0 0 0
44 39 1 0 0 0 0
50 51 1 0 0 0 0
46 50 1 0 0 0 0
52 53 1 0 0 0 0
24 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 56
M SMT 1 ^ CH2OH
M SBV 1 56 0.3488 -0.6899
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 52 53
M SBL 2 1 58
M SMT 2 ^ CH2OH
M SBV 2 58 0.0572 0.6696
S SKP 5
ID FL5FACGL0044
FORMULA C32H38O21
EXACTMASS 758.190558278
AVERAGEMASS 758.6315199999999
SMILES c(c(O)6)c(c(c1c6)C(C(OC(C4OC(O5)C(C(O)C(O)C5)O)OC(C(O)C(O)4)CO)=C(c(c2)ccc(c(OC(O3)C(O)C(O)C(C3CO)O)2)O)O1)=O)O
M END