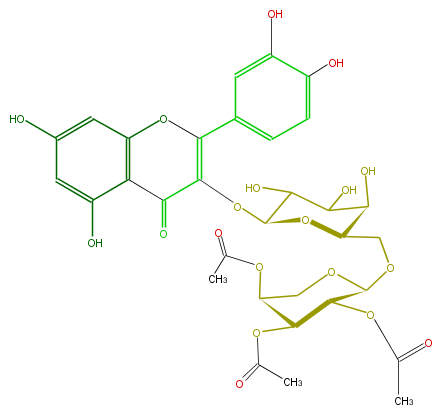
Mol:FL5FACGA0036
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 55 0 0 0 0 0 0 0 0999 V2000 | + | 51 55 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.3680 1.3521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3680 1.3521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3680 0.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3680 0.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6535 0.1145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6535 0.1145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9390 0.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9390 0.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9390 1.3521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9390 1.3521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6535 1.7645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6535 1.7645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2244 0.1145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2244 0.1145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5099 0.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5099 0.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5099 1.3521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5099 1.3521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2244 1.7645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2244 1.7645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2244 -0.5288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2244 -0.5288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2043 1.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2043 1.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9326 1.3440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9326 1.3440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6608 1.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6608 1.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6608 2.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6608 2.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9326 3.0258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9326 3.0258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2043 2.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2043 2.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0822 1.7644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0822 1.7644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6535 -0.7103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6535 -0.7103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1484 2.8869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1484 2.8869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3306 0.2751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3306 0.2751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8134 -0.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8134 -0.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6137 -0.2578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6137 -0.2578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3418 -0.6108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3418 -0.6108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8591 0.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8591 0.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0587 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0587 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6451 0.3959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6451 0.3959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4098 0.3405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4098 0.3405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8033 0.7193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8033 0.7193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0874 -0.6331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0874 -0.6331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6871 -1.8419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6871 -1.8419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4042 -2.3990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4042 -2.3990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0874 -1.8980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0874 -1.8980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8213 -1.6929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8213 -1.6929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1431 -1.3014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1431 -1.3014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4389 -1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4389 -1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7027 -1.1939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7027 -1.1939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6441 -2.5196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6441 -2.5196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9252 -2.1610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9252 -2.1610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3149 -1.2183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3149 -1.2183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2553 0.0083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2553 0.0083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9326 3.8665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9326 3.8665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0020 -0.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0020 -0.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1238 -0.5153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1238 -0.5153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2573 -1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2573 -1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6647 -3.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6647 -3.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2410 -3.4980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2410 -3.4980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2997 -3.5303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2997 -3.5303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4664 -3.0784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4664 -3.0784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0822 -2.7228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0822 -2.7228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4664 -3.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4664 -3.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 3 19 1 0 0 0 0 | + | 3 19 1 0 0 0 0 |
| − | 20 15 1 0 0 0 0 | + | 20 15 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 24 30 1 0 0 0 0 | + | 24 30 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 40 30 1 0 0 0 0 | + | 40 30 1 0 0 0 0 |
| − | 22 41 1 0 0 0 0 | + | 22 41 1 0 0 0 0 |
| − | 41 8 1 0 0 0 0 | + | 41 8 1 0 0 0 0 |
| − | 16 42 1 0 0 0 0 | + | 16 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 38 46 1 0 0 0 0 | + | 38 46 1 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 46 48 2 0 0 0 0 | + | 46 48 2 0 0 0 0 |
| − | 39 49 1 0 0 0 0 | + | 39 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 49 51 1 0 0 0 0 | + | 49 51 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FACGA0036 | + | ID FL5FACGA0036 |
| − | FORMULA C32H34O19 | + | FORMULA C32H34O19 |
| − | EXACTMASS 722.1694289059999 | + | EXACTMASS 722.1694289059999 |
| − | AVERAGEMASS 722.60096 | + | AVERAGEMASS 722.60096 |
| − | SMILES c(c12)(OC(c(c5)ccc(O)c5O)=C(OC(C3O)OC(COC(O4)C(OC(C)=O)C(OC(C)=O)C(OC(C)=O)C4)C(C3O)O)C2=O)cc(cc1O)O | + | SMILES c(c12)(OC(c(c5)ccc(O)c5O)=C(OC(C3O)OC(COC(O4)C(OC(C)=O)C(OC(C)=O)C(OC(C)=O)C4)C(C3O)O)C2=O)cc(cc1O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 55 0 0 0 0 0 0 0 0999 V2000
-3.3680 1.3521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3680 0.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6535 0.1145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9390 0.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9390 1.3521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6535 1.7645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2244 0.1145 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5099 0.5270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5099 1.3521 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2244 1.7645 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.2244 -0.5288 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2043 1.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9326 1.3440 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6608 1.7644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6608 2.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9326 3.0258 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2043 2.6053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0822 1.7644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6535 -0.7103 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1484 2.8869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3306 0.2751 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8134 -0.3413 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6137 -0.2578 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3418 -0.6108 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8591 0.0055 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0587 -0.0778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6451 0.3959 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4098 0.3405 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8033 0.7193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0874 -0.6331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6871 -1.8419 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4042 -2.3990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0874 -1.8980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8213 -1.6929 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1431 -1.3014 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4389 -1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7027 -1.1939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6441 -2.5196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9252 -2.1610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3149 -1.2183 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2553 0.0083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9326 3.8665 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0020 -0.9846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1238 -0.5153 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2573 -1.4336 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6647 -3.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2410 -3.4980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2997 -3.5303 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4664 -3.0784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0822 -2.7228 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4664 -3.8665 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
3 19 1 0 0 0 0
20 15 1 0 0 0 0
21 22 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
24 30 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
40 30 1 0 0 0 0
22 41 1 0 0 0 0
41 8 1 0 0 0 0
16 42 1 0 0 0 0
37 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
38 46 1 0 0 0 0
46 47 1 0 0 0 0
46 48 2 0 0 0 0
39 49 1 0 0 0 0
49 50 2 0 0 0 0
49 51 1 0 0 0 0
S SKP 5
ID FL5FACGA0036
FORMULA C32H34O19
EXACTMASS 722.1694289059999
AVERAGEMASS 722.60096
SMILES c(c12)(OC(c(c5)ccc(O)c5O)=C(OC(C3O)OC(COC(O4)C(OC(C)=O)C(OC(C)=O)C(OC(C)=O)C4)C(C3O)O)C2=O)cc(cc1O)O
M END