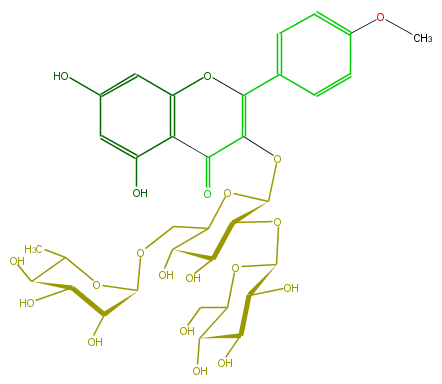
Mol:FL5FABGS0012
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.2766 3.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2766 3.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2766 2.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2766 2.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9909 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9909 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7054 2.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7054 2.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7054 3.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7054 3.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9909 3.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9909 3.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5621 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5621 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1524 2.3294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1524 2.3294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8667 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8667 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8667 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8667 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1524 0.6795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1524 0.6795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5621 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5621 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5812 2.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5812 2.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2956 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2956 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2956 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2956 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5812 0.6795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5812 0.6795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1524 -0.0386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1524 -0.0386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0101 2.3294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0101 2.3294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2397 0.6572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2397 0.6572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3060 3.5011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3060 3.5011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5812 -0.0199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5812 -0.0199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0341 3.0809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0341 3.0809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8569 -1.1613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8569 -1.1613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0539 -1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0539 -1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3421 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3421 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0518 -0.2067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0518 -0.2067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2489 -0.0160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2489 -0.0160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1473 -0.7400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1473 -0.7400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2508 -0.6015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2508 -0.6015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0589 -1.6455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0589 -1.6455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5113 -1.7089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5113 -1.7089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8489 -0.6958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8489 -0.6958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4867 -1.2163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4867 -1.2163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0020 -1.3293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0020 -1.3293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6802 -1.7995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6802 -1.7995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8659 -1.9331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8659 -1.9331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2333 -2.4630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2333 -2.4630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5551 -1.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5551 -1.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3694 -1.8592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3694 -1.8592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0341 -1.3803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0341 -1.3803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5226 -1.1331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5226 -1.1331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7007 -2.2092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7007 -2.2092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4828 -2.9888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4828 -2.9888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3217 -2.9390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3217 -2.9390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5018 -2.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5018 -2.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6676 -2.0768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6676 -2.0768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2221 -1.4657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2221 -1.4657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3986 -1.5194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3986 -1.5194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2328 -2.3278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2328 -2.3278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2988 -2.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2988 -2.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6358 -2.7659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6358 -2.7659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3690 -3.5668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3690 -3.5668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1343 -3.4218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1343 -3.4218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4556 -1.9170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4556 -1.9170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 20 22 1 0 0 0 0 | + | 20 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 23 30 1 0 0 0 0 | + | 23 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 28 32 1 0 0 0 0 | + | 28 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 38 37 1 1 0 0 0 | + | 38 37 1 1 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 26 19 1 0 0 0 0 | + | 26 19 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 47 46 1 1 0 0 0 | + | 47 46 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 44 52 1 0 0 0 0 | + | 44 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 47 29 1 0 0 0 0 | + | 47 29 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FABGS0012 | + | ID FL5FABGS0012 |
| − | FORMULA C34H42O20 | + | FORMULA C34H42O20 |
| − | EXACTMASS 770.226943784 | + | EXACTMASS 770.226943784 |
| − | AVERAGEMASS 770.6852799999999 | + | AVERAGEMASS 770.6852799999999 |
| − | SMILES C(C1O)(C(C(OC(OCC(O2)C(C(C(OC(O6)C(C(O)C(O)C6CO)O)C2OC(C5=O)=C(Oc(c54)cc(O)cc4O)c(c3)ccc(OC)c3)O)O)1)C)O)O | + | SMILES C(C1O)(C(C(OC(OCC(O2)C(C(C(OC(O6)C(C(O)C(O)C6CO)O)C2OC(C5=O)=C(Oc(c54)cc(O)cc4O)c(c3)ccc(OC)c3)O)O)1)C)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
1.2766 3.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2766 2.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9909 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7054 2.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7054 3.1544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9909 3.5668 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5621 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1524 2.3294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8667 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8667 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1524 0.6795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5621 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5812 2.3294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2956 1.9169 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2956 1.0920 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5812 0.6795 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1524 -0.0386 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0101 2.3294 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2397 0.6572 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3060 3.5011 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5812 -0.0199 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0341 3.0809 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8569 -1.1613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0539 -1.3520 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3421 -0.6279 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0518 -0.2067 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2489 -0.0160 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1473 -0.7400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2508 -0.6015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0589 -1.6455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.5113 -1.7089 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8489 -0.6958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4867 -1.2163 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0020 -1.3293 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6802 -1.7995 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8659 -1.9331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2333 -2.4630 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5551 -1.9928 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3694 -1.8592 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0341 -1.3803 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5226 -1.1331 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7007 -2.2092 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4828 -2.9888 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3217 -2.9390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5018 -2.8853 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6676 -2.0768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2221 -1.4657 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3986 -1.5194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2328 -2.3278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2988 -2.3138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6358 -2.7659 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3690 -3.5668 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1343 -3.4218 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4556 -1.9170 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
20 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
25 29 1 0 0 0 0
23 30 1 0 0 0 0
24 31 1 0 0 0 0
28 32 1 0 0 0 0
32 33 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
38 37 1 1 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
35 40 1 0 0 0 0
34 41 1 0 0 0 0
36 42 1 0 0 0 0
37 43 1 0 0 0 0
33 38 1 0 0 0 0
26 19 1 0 0 0 0
44 45 1 1 0 0 0
45 46 1 1 0 0 0
47 46 1 1 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
44 52 1 0 0 0 0
45 53 1 0 0 0 0
46 54 1 0 0 0 0
47 29 1 0 0 0 0
S SKP 5
ID FL5FABGS0012
FORMULA C34H42O20
EXACTMASS 770.226943784
AVERAGEMASS 770.6852799999999
SMILES C(C1O)(C(C(OC(OCC(O2)C(C(C(OC(O6)C(C(O)C(O)C6CO)O)C2OC(C5=O)=C(Oc(c54)cc(O)cc4O)c(c3)ccc(OC)c3)O)O)1)C)O)O
M END