Mol:FL5FAAGS0118
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.1252 2.6149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1252 2.6149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1252 1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1252 1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8395 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8395 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5540 1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5540 1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5540 2.6149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5540 2.6149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8395 3.0274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8395 3.0274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4107 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4107 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6962 1.7899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6962 1.7899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0182 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0182 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0182 0.5525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0182 0.5525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6962 0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6962 0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4107 0.5525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4107 0.5525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7327 1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7327 1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4471 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4471 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4471 0.5525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4471 0.5525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7327 0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7327 0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6962 -0.5783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6962 -0.5783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1616 1.7899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1616 1.7899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0170 0.2024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0170 0.2024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1547 2.9617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1547 2.9617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7327 -0.5596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7327 -0.5596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6333 -2.2442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6333 -2.2442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4528 -2.1471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4528 -2.1471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5757 -1.3311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5757 -1.3311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0972 -0.6916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0972 -0.6916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2777 -0.7886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2777 -0.7886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1547 -1.6046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1547 -1.6046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5572 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5572 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1166 -1.3996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1166 -1.3996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7257 -3.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7257 -3.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2495 -2.6690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2495 -2.6690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3448 -1.0619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3448 -1.0619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4217 -1.7688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4217 -1.7688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5095 -2.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5095 -2.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8758 -2.0607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8758 -2.0607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0613 -1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0613 -1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9732 -1.1082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9732 -1.1082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6070 -1.6368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6070 -1.6368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5109 -1.2536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5109 -1.2536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2514 -2.3028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2514 -2.3028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0762 -2.8675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0762 -2.8675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9078 -2.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9078 -2.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1547 -2.7167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1547 -2.7167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0753 -1.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0753 -1.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 40 1 0 0 0 0 | + | 34 40 1 0 0 0 0 |
| − | 35 41 1 0 0 0 0 | + | 35 41 1 0 0 0 0 |
| − | 36 29 1 0 0 0 0 | + | 36 29 1 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
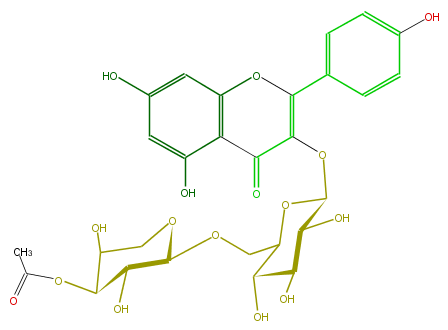
| − | ID FL5FAAGS0118 | + | ID FL5FAAGS0118 |
| − | FORMULA C28H30O16 | + | FORMULA C28H30O16 |
| − | EXACTMASS 622.153384912 | + | EXACTMASS 622.153384912 |
| − | AVERAGEMASS 622.5282 | + | AVERAGEMASS 622.5282 |
| − | SMILES C(C1O)(O)C(C(COC(C(O)5)OCC(O)C5OC(C)=O)OC1OC(=C3c(c4)ccc(O)c4)C(c(c(O3)2)c(cc(O)c2)O)=O)O | + | SMILES C(C1O)(O)C(C(COC(C(O)5)OCC(O)C5OC(C)=O)OC1OC(=C3c(c4)ccc(O)c4)C(c(c(O3)2)c(cc(O)c2)O)=O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
2.1252 2.6149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1252 1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8395 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5540 1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5540 2.6149 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8395 3.0274 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4107 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6962 1.7899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0182 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0182 0.5525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6962 0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4107 0.5525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7327 1.7899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4471 1.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4471 0.5525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7327 0.1400 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6962 -0.5783 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1616 1.7899 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0170 0.2024 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1547 2.9617 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7327 -0.5596 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6333 -2.2442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4528 -2.1471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5757 -1.3311 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0972 -0.6916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2777 -0.7886 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1547 -1.6046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5572 -1.8093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1166 -1.3996 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7257 -3.0274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2495 -2.6690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3448 -1.0619 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4217 -1.7688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5095 -2.5893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8758 -2.0607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0613 -1.9288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9732 -1.1082 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6070 -1.6368 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5109 -1.2536 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2514 -2.3028 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0762 -2.8675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9078 -2.1871 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1547 -2.7167 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0753 -1.7268 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 19 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
33 39 1 0 0 0 0
34 40 1 0 0 0 0
35 41 1 0 0 0 0
36 29 1 0 0 0 0
40 42 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
S SKP 5
ID FL5FAAGS0118
FORMULA C28H30O16
EXACTMASS 622.153384912
AVERAGEMASS 622.5282
SMILES C(C1O)(O)C(C(COC(C(O)5)OCC(O)C5OC(C)=O)OC1OC(=C3c(c4)ccc(O)c4)C(c(c(O3)2)c(cc(O)c2)O)=O)O
M END