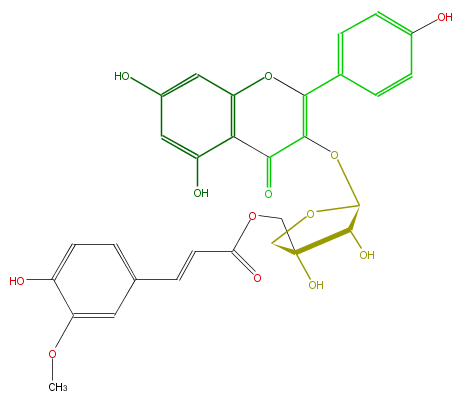
Mol:FL5FAAGS0107
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.1798 3.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1798 3.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1798 2.4892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1798 2.4892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8943 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8943 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6088 2.4892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6088 2.4892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6088 3.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6088 3.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8943 3.7267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8943 3.7267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4654 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4654 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7509 2.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7509 2.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0364 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0364 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0364 1.2517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0364 1.2517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7509 0.8392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7509 0.8392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4654 1.2517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4654 1.2517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6780 2.4892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6780 2.4892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3925 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3925 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3925 1.2517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3925 1.2517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6780 0.8392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6780 0.8392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7509 0.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7509 0.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1070 2.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1070 2.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0717 0.9016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0717 0.9016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2095 3.6610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2095 3.6610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6780 0.1397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6780 0.1397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2814 -1.0705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2814 -1.0705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6399 -1.6913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6399 -1.6913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6473 -1.0856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6473 -1.0856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0054 -0.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0054 -0.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5657 -0.1287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5657 -0.1287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8040 -0.8834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8040 -0.8834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3694 -0.6043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3694 -0.6043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5836 -0.2681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5836 -0.2681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4389 -0.3175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4389 -0.3175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0248 -1.0347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0248 -1.0347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5390 -1.5489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5390 -1.5489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7451 -1.0347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7451 -1.0347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0775 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0775 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9187 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9187 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3312 -0.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3312 -0.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1562 -0.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1562 -0.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5687 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5687 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1562 -2.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1562 -2.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3312 -2.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3312 -2.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2095 -1.6105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2095 -1.6105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5784 -3.0639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5784 -3.0639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5784 -3.7267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5784 -3.7267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 28 22 1 1 0 0 0 | + | 28 22 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 26 28 1 1 0 0 0 | + | 26 28 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 28 24 1 0 0 0 0 | + | 28 24 1 0 0 0 0 |
| − | 29 26 1 0 0 0 0 | + | 29 26 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 22 25 1 0 0 0 0 | + | 22 25 1 0 0 0 0 |
| − | 19 26 1 0 0 0 0 | + | 19 26 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 2 0 0 0 0 | + | 31 32 2 0 0 0 0 |
| − | 31 33 1 0 0 0 0 | + | 31 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 35 1 0 0 0 0 | + | 40 35 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 39 42 1 0 0 0 0 | + | 39 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0107 | + | ID FL5FAAGS0107 |
| − | FORMULA C30H26O13 | + | FORMULA C30H26O13 |
| − | EXACTMASS 594.137340918 | + | EXACTMASS 594.137340918 |
| − | AVERAGEMASS 594.51964 | + | AVERAGEMASS 594.51964 |
| − | SMILES O(C2OC(=C4c(c5)ccc(O)c5)C(c(c(O)3)c(O4)cc(O)c3)=O)CC(C(O)2)(O)COC(=O)C=Cc(c1)cc(OC)c(O)c1 | + | SMILES O(C2OC(=C4c(c5)ccc(O)c5)C(c(c(O)3)c(O4)cc(O)c3)=O)CC(C(O)2)(O)COC(=O)C=Cc(c1)cc(OC)c(O)c1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
2.1798 3.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1798 2.4892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8943 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6088 2.4892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6088 3.3142 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8943 3.7267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4654 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7509 2.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0364 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0364 1.2517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7509 0.8392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4654 1.2517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6780 2.4892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3925 2.0767 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3925 1.2517 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6780 0.8392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7509 0.1209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1070 2.4892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0717 0.9016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2095 3.6610 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6780 0.1397 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2814 -1.0705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6399 -1.6913 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6473 -1.0856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0054 -0.3851 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5657 -0.1287 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8040 -0.8834 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3694 -0.6043 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5836 -0.2681 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4389 -0.3175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0248 -1.0347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5390 -1.5489 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7451 -1.0347 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0775 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9187 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3312 -0.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1562 -0.8960 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5687 -1.6105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1562 -2.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3312 -2.3250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2095 -1.6105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5784 -3.0639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5784 -3.7267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
28 22 1 1 0 0 0
27 22 1 1 0 0 0
26 28 1 1 0 0 0
22 23 1 0 0 0 0
28 24 1 0 0 0 0
29 26 1 0 0 0 0
25 30 1 0 0 0 0
27 29 1 0 0 0 0
22 25 1 0 0 0 0
19 26 1 0 0 0 0
30 31 1 0 0 0 0
31 32 2 0 0 0 0
31 33 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 35 1 0 0 0 0
38 41 1 0 0 0 0
39 42 1 0 0 0 0
42 43 1 0 0 0 0
S SKP 5
ID FL5FAAGS0107
FORMULA C30H26O13
EXACTMASS 594.137340918
AVERAGEMASS 594.51964
SMILES O(C2OC(=C4c(c5)ccc(O)c5)C(c(c(O)3)c(O4)cc(O)c3)=O)CC(C(O)2)(O)COC(=O)C=Cc(c1)cc(OC)c(O)c1
M END