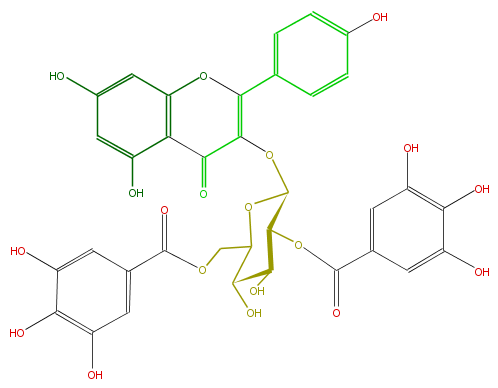
Mol:FL5FAAGS0101
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.5117 3.1958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5117 3.1958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5117 2.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5117 2.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2262 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2262 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9406 2.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9406 2.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9406 3.1958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9406 3.1958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2262 3.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2262 3.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2028 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2028 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9173 2.3708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9173 2.3708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6317 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6317 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6317 1.1333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6317 1.1333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9173 0.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9173 0.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2028 1.1333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2028 1.1333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3462 2.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3462 2.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0607 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0607 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0607 1.1333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0607 1.1333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3462 0.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3462 0.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9173 0.0025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9173 0.0025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7751 2.3708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7751 2.3708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4035 0.7832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4035 0.7832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5413 3.5426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5413 3.5426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3462 0.0212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3462 0.0212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3609 -1.8016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3609 -1.8016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4220 -1.5406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4220 -1.5406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3771 -0.7165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3771 -0.7165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7584 0.0154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7584 0.0154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0245 -0.2455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0245 -0.2455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0203 -1.0696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0203 -1.0696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5851 -1.1060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5851 -1.1060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9527 -1.5206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9527 -1.5206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0157 -2.3718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0157 -2.3718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0783 -1.9282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0783 -1.9282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9697 -1.0128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9697 -1.0128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4378 -2.3768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4378 -2.3768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4321 -1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4321 -1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1437 -1.1344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1437 -1.1344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8610 -1.5420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8610 -1.5420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8667 -2.3670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8667 -2.3670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1551 -2.7844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1551 -2.7844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1607 -3.6083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1607 -3.6083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5830 -2.7740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5830 -2.7740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5717 -1.1252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5717 -1.1252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7158 -1.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7158 -1.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7102 -0.3209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7102 -0.3209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4406 -0.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4406 -0.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4406 -1.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4406 -1.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1550 -1.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1550 -1.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8695 -1.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8695 -1.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8695 -0.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8695 -0.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1550 0.1048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1550 0.1048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1550 0.9287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1550 0.9287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5830 0.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5830 0.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5830 -1.5446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5830 -1.5446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7271 -1.5446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7271 -1.5446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7271 -2.3685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7271 -2.3685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 37 40 1 0 0 0 0 | + | 37 40 1 0 0 0 0 |
| − | 36 41 1 0 0 0 0 | + | 36 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 44 45 2 0 0 0 0 | + | 44 45 2 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 47 52 1 0 0 0 0 | + | 47 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 32 53 1 0 0 0 0 | + | 32 53 1 0 0 0 0 |
| − | 29 42 1 0 0 0 0 | + | 29 42 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGS0101 | + | ID FL5FAAGS0101 |
| − | KNApSAcK_ID C00013768 | + | KNApSAcK_ID C00013768 |
| − | NAME Loropetalin D;Kaempferol 3-(2'',6''-digalloylglucoside);3-[[2,6-bis-O-(3,4,5-Trihydroxybenzoyl)-beta-D-glucopyranosyl]oxy]-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one | + | NAME Loropetalin D;Kaempferol 3-(2'',6''-digalloylglucoside);3-[[2,6-bis-O-(3,4,5-Trihydroxybenzoyl)-beta-D-glucopyranosyl]oxy]-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 197379-54-7 | + | CAS_RN 197379-54-7 |
| − | FORMULA C35H28O19 | + | FORMULA C35H28O19 |
| − | EXACTMASS 752.122478714 | + | EXACTMASS 752.122478714 |
| − | AVERAGEMASS 752.5854200000001 | + | AVERAGEMASS 752.5854200000001 |
| − | SMILES O(C(=O)c(c6)cc(c(c(O)6)O)O)CC(O1)C(O)C(C(OC(c(c5)cc(c(O)c5O)O)=O)C1OC(C4=O)=C(Oc(c43)cc(cc(O)3)O)c(c2)ccc(c2)O)O | + | SMILES O(C(=O)c(c6)cc(c(c(O)6)O)O)CC(O1)C(O)C(C(OC(c(c5)cc(c(O)c5O)O)=O)C1OC(C4=O)=C(Oc(c43)cc(cc(O)3)O)c(c2)ccc(c2)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
0.5117 3.1958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5117 2.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2262 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9406 2.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9406 3.1958 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2262 3.6083 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2028 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9173 2.3708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6317 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6317 1.1333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9173 0.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2028 1.1333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3462 2.3708 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0607 1.9583 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0607 1.1333 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3462 0.7208 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9173 0.0025 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7751 2.3708 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4035 0.7832 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5413 3.5426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3462 0.0212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3609 -1.8016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4220 -1.5406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3771 -0.7165 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7584 0.0154 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0245 -0.2455 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0203 -1.0696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5851 -1.1060 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9527 -1.5206 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0157 -2.3718 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.0783 -1.9282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9697 -1.0128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4378 -2.3768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4321 -1.5518 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1437 -1.1344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8610 -1.5420 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8667 -2.3670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1551 -2.7844 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1607 -3.6083 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5830 -2.7740 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5717 -1.1252 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7158 -1.1448 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7102 -0.3209 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4406 -0.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4406 -1.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1550 -1.5452 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8695 -1.1327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8695 -0.3077 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1550 0.1048 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1550 0.9287 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5830 0.1043 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5830 -1.5446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7271 -1.5446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7271 -2.3685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 19 1 0 0 0 0
33 34 2 0 0 0 0
34 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
37 40 1 0 0 0 0
36 41 1 0 0 0 0
34 42 1 0 0 0 0
42 43 2 0 0 0 0
44 45 2 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
47 48 1 0 0 0 0
48 49 2 0 0 0 0
49 44 1 0 0 0 0
49 50 1 0 0 0 0
48 51 1 0 0 0 0
47 52 1 0 0 0 0
45 53 1 0 0 0 0
53 54 2 0 0 0 0
32 53 1 0 0 0 0
29 42 1 0 0 0 0
S SKP 8
ID FL5FAAGS0101
KNApSAcK_ID C00013768
NAME Loropetalin D;Kaempferol 3-(2'',6''-digalloylglucoside);3-[[2,6-bis-O-(3,4,5-Trihydroxybenzoyl)-beta-D-glucopyranosyl]oxy]-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 197379-54-7
FORMULA C35H28O19
EXACTMASS 752.122478714
AVERAGEMASS 752.5854200000001
SMILES O(C(=O)c(c6)cc(c(c(O)6)O)O)CC(O1)C(O)C(C(OC(c(c5)cc(c(O)c5O)O)=O)C1OC(C4=O)=C(Oc(c43)cc(cc(O)3)O)c(c2)ccc(c2)O)O
M END