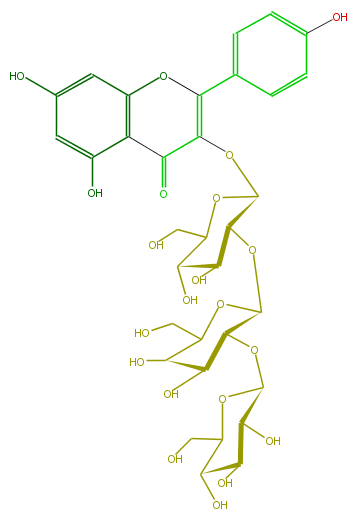
Mol:FL5FAAGS0083
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.1286 4.4980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1286 4.4980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1286 3.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1286 3.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8431 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8431 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5575 3.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5575 3.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5575 4.4980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5575 4.4980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8431 4.9105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8431 4.9105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4141 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4141 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3004 3.6730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3004 3.6730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0148 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0148 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0148 2.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0148 2.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3004 2.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3004 2.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4141 2.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4141 2.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7293 3.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7293 3.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4438 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4438 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4438 2.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4438 2.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7293 2.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7293 2.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3004 1.3047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3004 1.3047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1582 3.6730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1582 3.6730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0204 2.0854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0204 2.0854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1582 4.8448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1582 4.8448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7293 1.3235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7293 1.3235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0317 -0.1591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0317 -0.1591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7935 -0.1485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7935 -0.1485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0012 0.6502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0012 0.6502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5870 1.2316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5870 1.2316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7617 1.2210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7617 1.2210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5539 0.4223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5539 0.4223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0339 0.5717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0339 0.5717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5101 0.2885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5101 0.2885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1537 -0.8169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1537 -0.8169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3482 -0.4133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3482 -0.4133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4757 0.1878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4757 0.1878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4275 -4.3128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4275 -4.3128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2282 -4.1132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2282 -4.1132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2472 -3.2881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2472 -3.2881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6839 -2.5879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6839 -2.5879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8832 -2.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8832 -2.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8641 -3.6126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8641 -3.6126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2577 -3.6020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2577 -3.6020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1409 -3.9869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1409 -3.9869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7588 -4.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7588 -4.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8555 -4.4731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8555 -4.4731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8151 -3.6293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8151 -3.6293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2628 -2.0344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2628 -2.0344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5408 -2.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5408 -2.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9342 -1.4968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9342 -1.4968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6425 -1.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6425 -1.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8388 -0.8852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8388 -0.8852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4453 -1.6106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4453 -1.6106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1281 -1.3037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1281 -1.3037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6586 -1.4644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6586 -1.4644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7543 -2.0502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7543 -2.0502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2203 -2.6949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2203 -2.6949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5123 -1.8062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5123 -1.8062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 33 41 1 0 0 0 0 | + | 33 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 45 46 1 1 0 0 0 | + | 45 46 1 1 0 0 0 |
| − | 47 46 1 1 0 0 0 | + | 47 46 1 1 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 44 1 0 0 0 0 | + | 49 44 1 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 44 52 1 0 0 0 0 | + | 44 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 46 54 1 0 0 0 0 | + | 46 54 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 47 32 1 0 0 0 0 | + | 47 32 1 0 0 0 0 |
| − | 36 54 1 0 0 0 0 | + | 36 54 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGS0083 | + | ID FL5FAAGS0083 |
| − | KNApSAcK_ID C00013750 | + | KNApSAcK_ID C00013750 |
| − | NAME Kaempferol 3-glucosyl-(1->2)-galactosyl-(1->2)-glucoside;3-[(O-beta-D-Glucopyranosyl-(1->2)-O-beta-D-galactopyranosyl-(1->2)-beta-D-glucopyranosyl)oxy]-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one | + | NAME Kaempferol 3-glucosyl-(1->2)-galactosyl-(1->2)-glucoside;3-[(O-beta-D-Glucopyranosyl-(1->2)-O-beta-D-galactopyranosyl-(1->2)-beta-D-glucopyranosyl)oxy]-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 197250-98-9 | + | CAS_RN 197250-98-9 |
| − | FORMULA C33H40O21 | + | FORMULA C33H40O21 |
| − | EXACTMASS 772.206208342 | + | EXACTMASS 772.206208342 |
| − | AVERAGEMASS 772.6581 | + | AVERAGEMASS 772.6581 |
| − | SMILES OC(C6CO)C(C(O)C(O6)OC(C(OC(C2OC(C4=O)=C(c(c5)ccc(O)c5)Oc(c43)cc(cc3O)O)C(O)C(O)C(O2)CO)1)C(C(O)C(CO)O1)O)O | + | SMILES OC(C6CO)C(C(O)C(O6)OC(C(OC(C2OC(C4=O)=C(c(c5)ccc(O)c5)Oc(c43)cc(cc3O)O)C(O)C(O)C(O2)CO)1)C(C(O)C(CO)O1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
1.1286 4.4980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1286 3.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8431 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5575 3.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5575 4.4980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8431 4.9105 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4141 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3004 3.6730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.0148 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0148 2.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3004 2.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4141 2.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7293 3.6730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4438 3.2605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4438 2.4355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7293 2.0230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3004 1.3047 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1582 3.6730 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0204 2.0854 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1582 4.8448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7293 1.3235 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0317 -0.1591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7935 -0.1485 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0012 0.6502 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5870 1.2316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7617 1.2210 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5539 0.4223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0339 0.5717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5101 0.2885 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1537 -0.8169 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3482 -0.4133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4757 0.1878 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4275 -4.3128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2282 -4.1132 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2472 -3.2881 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6839 -2.5879 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8832 -2.7875 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8641 -3.6126 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2577 -3.6020 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1409 -3.9869 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7588 -4.9105 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8555 -4.4731 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8151 -3.6293 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2628 -2.0344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5408 -2.2222 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9342 -1.4968 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6425 -1.0731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8388 -0.8852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.4453 -1.6106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1281 -1.3037 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6586 -1.4644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7543 -2.0502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2203 -2.6949 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5123 -1.8062 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
38 39 1 0 0 0 0
39 40 1 0 0 0 0
33 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
44 45 1 1 0 0 0
45 46 1 1 0 0 0
47 46 1 1 0 0 0
47 48 1 0 0 0 0
48 49 1 0 0 0 0
49 44 1 0 0 0 0
49 50 1 0 0 0 0
50 51 1 0 0 0 0
44 52 1 0 0 0 0
45 53 1 0 0 0 0
46 54 1 0 0 0 0
25 19 1 0 0 0 0
47 32 1 0 0 0 0
36 54 1 0 0 0 0
S SKP 8
ID FL5FAAGS0083
KNApSAcK_ID C00013750
NAME Kaempferol 3-glucosyl-(1->2)-galactosyl-(1->2)-glucoside;3-[(O-beta-D-Glucopyranosyl-(1->2)-O-beta-D-galactopyranosyl-(1->2)-beta-D-glucopyranosyl)oxy]-5,7-dihydroxy-2-(4-hydroxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 197250-98-9
FORMULA C33H40O21
EXACTMASS 772.206208342
AVERAGEMASS 772.6581
SMILES OC(C6CO)C(C(O)C(O6)OC(C(OC(C2OC(C4=O)=C(c(c5)ccc(O)c5)Oc(c43)cc(cc3O)O)C(O)C(O)C(O2)CO)1)C(C(O)C(CO)O1)O)O
M END