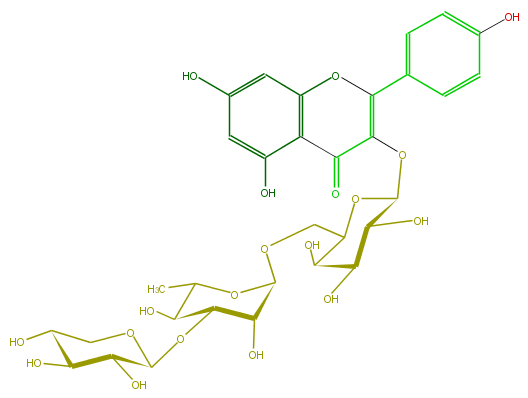
Mol:FL5FAAGS0077
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 51 56 0 0 0 0 0 0 0 0999 V2000 | + | 51 56 0 0 0 0 0 0 0 0999 V2000 |
| − | 2.8542 3.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8542 3.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8542 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8542 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5687 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5687 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2831 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2831 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2831 3.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2831 3.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5687 3.7065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5687 3.7065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1397 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1397 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4253 2.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4253 2.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7108 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7108 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7108 1.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7108 1.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4253 0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4253 0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1397 1.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1397 1.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0037 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0037 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7181 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7181 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7181 1.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7181 1.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0037 0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0037 0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4253 0.1007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4253 0.1007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4326 2.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4326 2.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7461 0.8814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7461 0.8814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8839 3.6408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8839 3.6408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0037 0.1195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0037 0.1195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9861 -1.3487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9861 -1.3487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8110 -1.3643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8110 -1.3643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0440 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0440 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6478 -0.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6478 -0.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8229 0.0050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8229 0.0050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5898 -0.7864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5898 -0.7864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9899 -0.5280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9899 -0.5280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0021 -0.9926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0021 -0.9926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8844 -0.9142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8844 -0.9142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2511 -1.9883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2511 -1.9883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0465 -0.4260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0465 -0.4260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3948 -1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3948 -1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8073 -2.4229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8073 -2.4229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0140 -2.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0140 -2.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2160 -2.4053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2160 -2.4053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1966 -1.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1966 -1.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5967 -1.9175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5967 -1.9175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2385 -3.1059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2385 -3.1059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6758 -2.7979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6758 -2.7979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2764 -2.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2764 -2.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0466 -1.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0466 -1.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2823 -2.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2823 -2.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8698 -3.3650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8698 -3.3650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0717 -3.1559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0717 -3.1559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2785 -3.3827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2785 -3.3827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6909 -2.6682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6909 -2.6682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4890 -2.8772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4890 -2.8772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5787 -3.7065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5787 -3.7065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5368 -3.2806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5368 -3.2806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8839 -2.8620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8839 -2.8620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 12 19 1 0 0 0 0 | + | 12 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 25 24 1 1 0 0 0 | + | 25 24 1 1 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 22 30 1 0 0 0 0 | + | 22 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 19 1 0 0 0 0 | + | 25 19 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 33 1 0 0 0 0 | + | 38 33 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 35 40 1 0 0 0 0 | + | 35 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 33 42 1 0 0 0 0 | + | 33 42 1 0 0 0 0 |
| − | 37 29 1 0 0 0 0 | + | 37 29 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 45 49 1 0 0 0 0 | + | 45 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 43 51 1 0 0 0 0 | + | 43 51 1 0 0 0 0 |
| − | 46 40 1 0 0 0 0 | + | 46 40 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0077 | + | ID FL5FAAGS0077 |
| − | FORMULA C32H38O19 | + | FORMULA C32H38O19 |
| − | EXACTMASS 726.200729034 | + | EXACTMASS 726.200729034 |
| − | AVERAGEMASS 726.6327200000001 | + | AVERAGEMASS 726.6327200000001 |
| − | SMILES OC(C1OC(C5=O)=C(Oc(c6)c5c(O)cc6O)c(c4)ccc(O)c4)C(C(C(COC(C2O)OC(C)C(C2OC(C3O)OCC(C3O)O)O)O1)O)O | + | SMILES OC(C1OC(C5=O)=C(Oc(c6)c5c(O)cc6O)c(c4)ccc(O)c4)C(C(C(COC(C2O)OC(C)C(C2OC(C3O)OCC(C3O)O)O)O1)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
51 56 0 0 0 0 0 0 0 0999 V2000
2.8542 3.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8542 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5687 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2831 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2831 3.2940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5687 3.7065 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1397 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4253 2.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7108 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7108 1.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4253 0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1397 1.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0037 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7181 2.0565 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7181 1.2315 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0037 0.8190 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4253 0.1007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4326 2.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7461 0.8814 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.8839 3.6408 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0037 0.1195 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9861 -1.3487 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8110 -1.3643 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0440 -0.5729 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6478 -0.0107 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8229 0.0050 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5898 -0.7864 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9899 -0.5280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0021 -0.9926 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8844 -0.9142 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2511 -1.9883 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0465 -0.4260 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3948 -1.7084 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8073 -2.4229 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0140 -2.1961 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2160 -2.4053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1966 -1.6909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5967 -1.9175 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2385 -3.1059 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6758 -2.7979 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2764 -2.2270 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0466 -1.7912 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2823 -2.6506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8698 -3.3650 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0717 -3.1559 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2785 -3.3827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6909 -2.6682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4890 -2.8772 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5787 -3.7065 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5368 -3.2806 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.8839 -2.8620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
12 19 1 0 0 0 0
5 20 1 0 0 0 0
16 21 1 0 0 0 0
22 23 1 1 0 0 0
23 24 1 1 0 0 0
25 24 1 1 0 0 0
25 26 1 0 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
27 28 1 0 0 0 0
28 29 1 0 0 0 0
22 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 19 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 33 1 0 0 0 0
36 39 1 0 0 0 0
35 40 1 0 0 0 0
34 41 1 0 0 0 0
33 42 1 0 0 0 0
37 29 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
45 49 1 0 0 0 0
44 50 1 0 0 0 0
43 51 1 0 0 0 0
46 40 1 0 0 0 0
S SKP 5
ID FL5FAAGS0077
FORMULA C32H38O19
EXACTMASS 726.200729034
AVERAGEMASS 726.6327200000001
SMILES OC(C1OC(C5=O)=C(Oc(c6)c5c(O)cc6O)c(c4)ccc(O)c4)C(C(C(COC(C2O)OC(C)C(C2OC(C3O)OCC(C3O)O)O)O1)O)O
M END