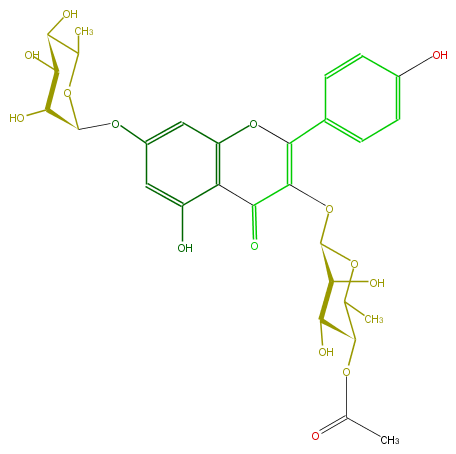
Mol:FL5FAAGS0065
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.6468 1.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6468 1.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6468 0.8296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6468 0.8296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9322 0.4172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9322 0.4172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2177 0.8296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2177 0.8296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2177 1.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2177 1.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9322 2.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9322 2.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4968 0.4172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4968 0.4172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2113 0.8296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2113 0.8296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2113 1.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2113 1.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4968 2.0673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4968 2.0673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5188 -0.3719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5188 -0.3719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9254 2.1316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9254 2.1316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6537 1.7016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6537 1.7016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3818 2.1316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3818 2.1316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3818 2.9919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3818 2.9919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6537 3.4219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6537 3.4219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9254 2.9919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9254 2.9919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9322 -0.4076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9322 -0.4076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1613 3.4522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1613 3.4522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2664 2.0712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2664 2.0712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0197 0.3630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0197 0.3630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6376 2.3303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6376 2.3303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0176 1.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0176 1.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2143 2.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2143 2.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0176 3.4433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0176 3.4433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6376 3.8329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6376 3.8329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4409 3.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4409 3.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2420 4.2632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2420 4.2632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0176 3.9294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0176 3.9294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9971 3.4513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9971 3.4513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1613 2.1775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1613 2.1775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8327 -1.8760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8327 -1.8760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5205 -2.2730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5205 -2.2730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3023 -1.5095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3023 -1.5095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5205 -0.7413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5205 -0.7413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8327 -0.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8327 -0.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0509 -1.1078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0509 -1.1078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8941 -1.1078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8941 -1.1078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8871 -2.4953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8871 -2.4953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3546 -2.8922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3546 -2.8922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7970 -1.8409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7970 -1.8409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3546 -3.8234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3546 -3.8234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7400 -4.1781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7400 -4.1781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1162 -4.2632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1162 -4.2632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 37 1 1 0 0 0 | + | 36 37 1 1 0 0 0 |
| − | 37 32 1 1 0 0 0 | + | 37 32 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 33 40 1 0 0 0 0 | + | 33 40 1 0 0 0 0 |
| − | 34 41 1 0 0 0 0 | + | 34 41 1 0 0 0 0 |
| − | 36 21 1 0 0 0 0 | + | 36 21 1 0 0 0 0 |
| − | 40 42 1 0 0 0 0 | + | 40 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 42 44 1 0 0 0 0 | + | 42 44 1 0 0 0 0 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGS0065 | + | ID FL5FAAGS0065 |
| − | FORMULA C29H32O15 | + | FORMULA C29H32O15 |
| − | EXACTMASS 620.174120354 | + | EXACTMASS 620.174120354 |
| − | AVERAGEMASS 620.55538 | + | AVERAGEMASS 620.55538 |
| − | SMILES OC(C1Oc(c2)cc(c(C(=O)3)c(OC(c(c5)ccc(c5)O)=C(OC(O4)C(O)C(C(OC(C)=O)C(C)4)O)3)2)O)C(C(C(O1)C)O)O | + | SMILES OC(C1Oc(c2)cc(c(C(=O)3)c(OC(c(c5)ccc(c5)O)=C(OC(O4)C(O)C(C(OC(C)=O)C(C)4)O)3)2)O)C(C(C(O1)C)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-1.6468 1.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6468 0.8296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9322 0.4172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2177 0.8296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.2177 1.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9322 2.0673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4968 0.4172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2113 0.8296 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2113 1.6548 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4968 2.0673 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5188 -0.3719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9254 2.1316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6537 1.7016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3818 2.1316 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3818 2.9919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6537 3.4219 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9254 2.9919 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9322 -0.4076 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1613 3.4522 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2664 2.0712 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0197 0.3630 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6376 2.3303 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0176 1.9408 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2143 2.6898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0176 3.4433 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6376 3.8329 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4409 3.0838 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2420 4.2632 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0176 3.9294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9971 3.4513 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1613 2.1775 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8327 -1.8760 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5205 -2.2730 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3023 -1.5095 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5205 -0.7413 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8327 -0.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0509 -1.1078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8941 -1.1078 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8871 -2.4953 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3546 -2.8922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7970 -1.8409 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3546 -3.8234 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7400 -4.1781 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1162 -4.2632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
27 30 1 0 0 0 0
22 31 1 0 0 0 0
23 20 1 0 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 37 1 1 0 0 0
37 32 1 1 0 0 0
37 38 1 0 0 0 0
32 39 1 0 0 0 0
33 40 1 0 0 0 0
34 41 1 0 0 0 0
36 21 1 0 0 0 0
40 42 1 0 0 0 0
42 43 2 0 0 0 0
42 44 1 0 0 0 0
S SKP 5
ID FL5FAAGS0065
FORMULA C29H32O15
EXACTMASS 620.174120354
AVERAGEMASS 620.55538
SMILES OC(C1Oc(c2)cc(c(C(=O)3)c(OC(c(c5)ccc(c5)O)=C(OC(O4)C(O)C(C(OC(C)=O)C(C)4)O)3)2)O)C(C(C(O1)C)O)O
M END