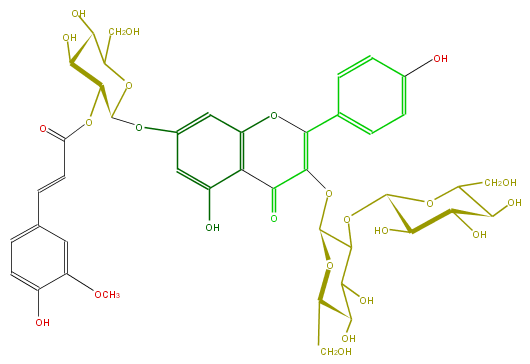
Mol:FL5FAAGL0096
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 67 73 0 0 0 0 0 0 0 0999 V2000 | + | 67 73 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8593 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8593 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8593 0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8593 0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1447 0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1447 0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4302 0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4302 0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4302 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4302 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1447 2.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1447 2.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2844 0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2844 0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9989 0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9989 0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9989 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9989 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2844 2.1253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2844 2.1253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2844 -0.1682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2844 -0.1682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7132 2.1252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7132 2.1252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4413 1.7047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4413 1.7047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1696 2.1252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1696 2.1252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1696 2.9661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1696 2.9661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4413 3.3865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4413 3.3865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7132 2.9661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7132 2.9661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6904 1.8502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6904 1.8502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8976 3.3864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8976 3.3864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5051 0.3882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5051 0.3882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1447 -0.3497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1447 -0.3497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0100 -0.8327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0100 -0.8327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2993 -0.4224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2993 -0.4224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5249 -1.2115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5249 -1.2115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2993 -2.0053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2993 -2.0053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0100 -2.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0100 -2.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7846 -1.6265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7846 -1.6265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9117 -0.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9117 -0.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7819 0.3395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7819 0.3395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3261 -0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3261 -0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2058 -0.0114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2058 -0.0114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1332 -0.1526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1332 -0.1526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3798 0.5188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3798 0.5188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7311 0.1744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7311 0.1744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7423 -0.4234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7423 -0.4234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7581 -0.5225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7581 -0.5225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7141 3.9178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7141 3.9178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2079 3.2498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2079 3.2498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5303 2.7619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5303 2.7619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3052 2.0397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3052 2.0397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9297 2.7910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9297 2.7910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4713 3.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4713 3.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1191 4.3839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1191 4.3839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3076 3.8351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3076 3.8351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8094 1.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8094 1.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3425 1.5558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3425 1.5558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8265 1.8352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8265 1.8352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3425 0.7451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3425 0.7451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9217 0.4106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9217 0.4106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9217 -0.3391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9217 -0.3391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5337 -0.6925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5337 -0.6925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5337 -1.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5337 -1.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9217 -1.7525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9217 -1.7525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3098 -1.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3098 -1.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3098 -0.6925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3098 -0.6925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9217 -2.4590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9217 -2.4590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5337 0.1850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5337 0.1850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2688 -1.9871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2688 -1.9871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8652 -2.8223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8652 -2.8223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0185 0.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0185 0.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1070 1.9220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1070 1.9220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3146 3.9797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3146 3.9797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5029 3.2986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5029 3.2986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2713 -3.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2713 -3.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2063 -4.3839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2063 -4.3839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6086 -1.8365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6086 -1.8365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4962 -2.4789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4962 -2.4789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 30 35 1 0 0 0 0 | + | 30 35 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 38 39 1 1 0 0 0 | + | 38 39 1 1 0 0 0 |
| − | 40 39 1 1 0 0 0 | + | 40 39 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | 37 43 1 0 0 0 0 | + | 37 43 1 0 0 0 0 |
| − | 38 44 1 0 0 0 0 | + | 38 44 1 0 0 0 0 |
| − | 18 40 1 0 0 0 0 | + | 18 40 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 2 0 0 0 0 | + | 46 47 2 0 0 0 0 |
| − | 46 48 1 0 0 0 0 | + | 46 48 1 0 0 0 0 |
| − | 48 49 2 0 0 0 0 | + | 48 49 2 0 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 51 2 0 0 0 0 | + | 50 51 2 0 0 0 0 |
| − | 51 52 1 0 0 0 0 | + | 51 52 1 0 0 0 0 |
| − | 52 53 2 0 0 0 0 | + | 52 53 2 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 54 55 2 0 0 0 0 | + | 54 55 2 0 0 0 0 |
| − | 55 50 1 0 0 0 0 | + | 55 50 1 0 0 0 0 |
| − | 53 56 1 0 0 0 0 | + | 53 56 1 0 0 0 0 |
| − | 45 39 1 0 0 0 0 | + | 45 39 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 32 57 1 0 0 0 0 | + | 32 57 1 0 0 0 0 |
| − | 27 58 1 0 0 0 0 | + | 27 58 1 0 0 0 0 |
| − | 26 59 1 0 0 0 0 | + | 26 59 1 0 0 0 0 |
| − | 60 61 1 0 0 0 0 | + | 60 61 1 0 0 0 0 |
| − | 33 60 1 0 0 0 0 | + | 33 60 1 0 0 0 0 |
| − | 62 63 1 0 0 0 0 | + | 62 63 1 0 0 0 0 |
| − | 42 62 1 0 0 0 0 | + | 42 62 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 25 64 1 0 0 0 0 | + | 25 64 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 54 66 1 0 0 0 0 | + | 54 66 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 60 61 | + | M SAL 1 2 60 61 |
| − | M SBL 1 1 67 | + | M SBL 1 1 67 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 67 -0.6387 -0.1218 | + | M SBV 1 67 -0.6387 -0.1218 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 62 63 | + | M SAL 2 2 62 63 |
| − | M SBL 2 1 69 | + | M SBL 2 1 69 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SBV 2 69 -0.1567 -0.7291 | + | M SBV 2 69 -0.1567 -0.7291 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 64 65 | + | M SAL 3 2 64 65 |
| − | M SBL 3 1 71 | + | M SBL 3 1 71 |
| − | M SMT 3 ^ CH2OH | + | M SMT 3 ^ CH2OH |
| − | M SBV 3 71 0.0280 1.0958 | + | M SBV 3 71 0.0280 1.0958 |
| − | M STY 1 4 SUP | + | M STY 1 4 SUP |
| − | M SLB 1 4 4 | + | M SLB 1 4 4 |
| − | M SAL 4 2 66 67 | + | M SAL 4 2 66 67 |
| − | M SBL 4 1 73 | + | M SBL 4 1 73 |
| − | M SMT 4 OCH3 | + | M SMT 4 OCH3 |
| − | M SBV 4 73 -0.7012 0.4373 | + | M SBV 4 73 -0.7012 0.4373 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0096 | + | ID FL5FAAGL0096 |
| − | FORMULA C43H48O24 | + | FORMULA C43H48O24 |
| − | EXACTMASS 948.253552464 | + | EXACTMASS 948.253552464 |
| − | AVERAGEMASS 948.82682 | + | AVERAGEMASS 948.82682 |
| − | SMILES C(OC(O7)C(O)C(O)C(C7CO)O)(C6O)C(OC(C(O)6)CO)OC(=C4c(c5)ccc(c5)O)C(c(c3O4)c(O)cc(c3)OC(C(OC(C=Cc(c2)cc(c(O)c2)OC)=O)1)OC(CO)C(O)C1O)=O | + | SMILES C(OC(O7)C(O)C(O)C(C7CO)O)(C6O)C(OC(C(O)6)CO)OC(=C4c(c5)ccc(c5)O)C(c(c3O4)c(O)cc(c3)OC(C(OC(C=Cc(c2)cc(c(O)c2)OC)=O)1)OC(CO)C(O)C1O)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
67 73 0 0 0 0 0 0 0 0999 V2000
-1.8593 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8593 0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1447 0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4302 0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4302 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1447 2.1253 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2844 0.4752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9989 0.8878 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9989 1.7128 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2844 2.1253 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2844 -0.1682 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7132 2.1252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4413 1.7047 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1696 2.1252 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1696 2.9661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4413 3.3865 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7132 2.9661 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6904 1.8502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8976 3.3864 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5051 0.3882 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1447 -0.3497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0100 -0.8327 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2993 -0.4224 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5249 -1.2115 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2993 -2.0053 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0100 -2.4155 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7846 -1.6265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9117 -0.1766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7819 0.3395 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3261 -0.4940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2058 -0.0114 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1332 -0.1526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3798 0.5188 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7311 0.1744 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7423 -0.4234 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7581 -0.5225 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7141 3.9178 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.2079 3.2498 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5303 2.7619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3052 2.0397 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9297 2.7910 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4713 3.2506 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1191 4.3839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3076 3.8351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8094 1.9685 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3425 1.5558 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8265 1.8352 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3425 0.7451 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9217 0.4106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9217 -0.3391 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5337 -0.6925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5337 -1.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9217 -1.7525 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3098 -1.3992 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3098 -0.6925 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9217 -2.4590 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5337 0.1850 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2688 -1.9871 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8652 -2.8223 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0185 0.6406 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1070 1.9220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3146 3.9797 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5029 3.2986 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2713 -3.1011 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2063 -4.3839 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6086 -1.8365 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4962 -2.4789 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
23 20 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
30 35 1 0 0 0 0
31 36 1 0 0 0 0
37 38 1 1 0 0 0
38 39 1 1 0 0 0
40 39 1 1 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 37 1 0 0 0 0
37 43 1 0 0 0 0
38 44 1 0 0 0 0
18 40 1 0 0 0 0
45 46 1 0 0 0 0
46 47 2 0 0 0 0
46 48 1 0 0 0 0
48 49 2 0 0 0 0
49 50 1 0 0 0 0
50 51 2 0 0 0 0
51 52 1 0 0 0 0
52 53 2 0 0 0 0
53 54 1 0 0 0 0
54 55 2 0 0 0 0
55 50 1 0 0 0 0
53 56 1 0 0 0 0
45 39 1 0 0 0 0
28 29 1 0 0 0 0
32 57 1 0 0 0 0
27 58 1 0 0 0 0
26 59 1 0 0 0 0
60 61 1 0 0 0 0
33 60 1 0 0 0 0
62 63 1 0 0 0 0
42 62 1 0 0 0 0
64 65 1 0 0 0 0
25 64 1 0 0 0 0
66 67 1 0 0 0 0
54 66 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 60 61
M SBL 1 1 67
M SMT 1 CH2OH
M SBV 1 67 -0.6387 -0.1218
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 62 63
M SBL 2 1 69
M SMT 2 CH2OH
M SBV 2 69 -0.1567 -0.7291
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 64 65
M SBL 3 1 71
M SMT 3 ^ CH2OH
M SBV 3 71 0.0280 1.0958
M STY 1 4 SUP
M SLB 1 4 4
M SAL 4 2 66 67
M SBL 4 1 73
M SMT 4 OCH3
M SBV 4 73 -0.7012 0.4373
S SKP 5
ID FL5FAAGL0096
FORMULA C43H48O24
EXACTMASS 948.253552464
AVERAGEMASS 948.82682
SMILES C(OC(O7)C(O)C(O)C(C7CO)O)(C6O)C(OC(C(O)6)CO)OC(=C4c(c5)ccc(c5)O)C(c(c3O4)c(O)cc(c3)OC(C(OC(C=Cc(c2)cc(c(O)c2)OC)=O)1)OC(CO)C(O)C1O)=O
M END