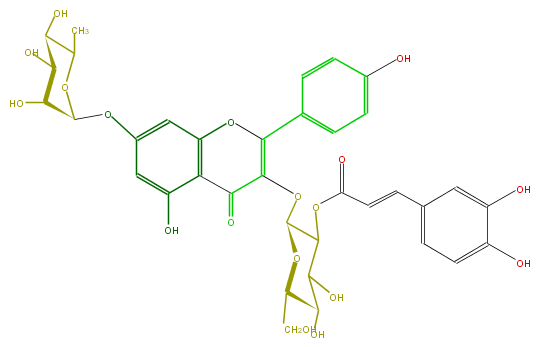
Mol:FL5FAAGL0089
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 54 59 0 0 0 0 0 0 0 0999 V2000 | + | 54 59 0 0 0 0 0 0 0 0999 V2000 |
| − | -3.0267 1.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0267 1.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0267 0.5063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0267 0.5063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3122 0.0938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3122 0.0938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5978 0.5063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5978 0.5063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5978 1.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5978 1.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3122 1.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3122 1.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8834 0.0938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8834 0.0938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1690 0.5063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1690 0.5063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1690 1.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1690 1.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8834 1.7437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8834 1.7437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8834 -0.5493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8834 -0.5493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7305 1.9026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7305 1.9026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4587 1.4821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4587 1.4821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1869 1.9026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1869 1.9026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1869 2.7434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1869 2.7434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4587 3.1637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4587 3.1637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7305 2.7434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7305 2.7434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3122 -0.7308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3122 -0.7308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9661 3.1932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9661 3.1932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6890 1.9140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6890 1.9140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6393 0.0396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6393 0.0396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1154 2.1718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1154 2.1718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4409 1.7822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4409 1.7822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6550 2.5312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6550 2.5312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4409 3.2846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4409 3.2846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1154 3.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1154 3.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9014 2.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9014 2.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8395 4.2189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8395 4.2189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4518 3.8355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4518 3.8355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5067 3.3338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5067 3.3338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6838 2.1621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6838 2.1621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0948 -0.9784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0948 -0.9784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3844 -0.5683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3844 -0.5683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6098 -1.3570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6098 -1.3570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3844 -2.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3844 -2.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0948 -2.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0948 -2.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8694 -1.7721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8694 -1.7721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0365 -0.1973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0365 -0.1973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4354 -2.2426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4354 -2.2426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9912 -3.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9912 -3.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6364 0.1110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6364 0.1110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6351 0.8944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6351 0.8944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2481 -0.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2481 -0.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8833 0.1372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8833 0.1372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4804 -0.2042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4804 -0.2042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4804 -1.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4804 -1.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2151 -1.4768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2151 -1.4768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9498 -1.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9498 -1.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9498 -0.2042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9498 -0.2042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2151 0.2200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2151 0.2200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6838 -1.4764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6838 -1.4764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6838 0.2196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6838 0.2196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2877 -2.9765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2877 -2.9765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0375 -4.2189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0375 -4.2189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 27 1 1 0 0 0 | + | 26 27 1 1 0 0 0 |
| − | 27 22 1 1 0 0 0 | + | 27 22 1 1 0 0 0 |
| − | 26 28 1 0 0 0 0 | + | 26 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 27 30 1 0 0 0 0 | + | 27 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 23 20 1 0 0 0 0 | + | 23 20 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 1 0 0 0 | + | 33 34 1 1 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 36 35 1 1 0 0 0 | + | 36 35 1 1 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 32 1 0 0 0 0 | + | 37 32 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 37 39 1 0 0 0 0 | + | 37 39 1 0 0 0 0 |
| − | 36 40 1 0 0 0 0 | + | 36 40 1 0 0 0 0 |
| − | 33 21 1 0 0 0 0 | + | 33 21 1 0 0 0 0 |
| − | 38 41 1 0 0 0 0 | + | 38 41 1 0 0 0 0 |
| − | 42 41 2 0 0 0 0 | + | 42 41 2 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 44 45 1 0 0 0 0 | + | 44 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 49 52 1 0 0 0 0 | + | 49 52 1 0 0 0 0 |
| − | 53 54 1 0 0 0 0 | + | 53 54 1 0 0 0 0 |
| − | 35 53 1 0 0 0 0 | + | 35 53 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 53 54 | + | M SAL 1 2 53 54 |
| − | M SBL 1 1 59 | + | M SBL 1 1 59 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 59 0.0967 0.8258 | + | M SBV 1 59 0.0967 0.8258 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0089 | + | ID FL5FAAGL0089 |
| − | FORMULA C36H36O18 | + | FORMULA C36H36O18 |
| − | EXACTMASS 756.190164348 | + | EXACTMASS 756.190164348 |
| − | AVERAGEMASS 756.6602399999999 | + | AVERAGEMASS 756.6602399999999 |
| − | SMILES C(C(O)1)(O)C(OC(OC(=C(c(c6)ccc(O)c6)5)C(=O)c(c4O5)c(cc(c4)OC(O3)C(C(C(C3C)O)O)O)O)C1OC(C=Cc(c2)ccc(c2O)O)=O)CO | + | SMILES C(C(O)1)(O)C(OC(OC(=C(c(c6)ccc(O)c6)5)C(=O)c(c4O5)c(cc(c4)OC(O3)C(C(C(C3C)O)O)O)O)C1OC(C=Cc(c2)ccc(c2O)O)=O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
54 59 0 0 0 0 0 0 0 0999 V2000
-3.0267 1.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0267 0.5063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3122 0.0938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5978 0.5063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5978 1.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3122 1.7437 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8834 0.0938 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1690 0.5063 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1690 1.3313 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8834 1.7437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8834 -0.5493 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7305 1.9026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4587 1.4821 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1869 1.9026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1869 2.7434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4587 3.1637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7305 2.7434 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3122 -0.7308 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9661 3.1932 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6890 1.9140 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6393 0.0396 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1154 2.1718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4409 1.7822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6550 2.5312 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4409 3.2846 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1154 3.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.9014 2.9251 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8395 4.2189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4518 3.8355 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5067 3.3338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.6838 2.1621 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.0948 -0.9784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3844 -0.5683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6098 -1.3570 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3844 -2.1507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0948 -2.5610 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8694 -1.7721 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0365 -0.1973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4354 -2.2426 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9912 -3.0914 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6364 0.1110 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6351 0.8944 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2481 -0.1831 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8833 0.1372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4804 -0.2042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4804 -1.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2151 -1.4768 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9498 -1.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9498 -0.2042 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2151 0.2200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.6838 -1.4764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6838 0.2196 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2877 -2.9765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0375 -4.2189 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 27 1 1 0 0 0
27 22 1 1 0 0 0
26 28 1 0 0 0 0
25 29 1 0 0 0 0
27 30 1 0 0 0 0
22 31 1 0 0 0 0
23 20 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 1 0 0 0
34 35 1 1 0 0 0
36 35 1 1 0 0 0
36 37 1 0 0 0 0
37 32 1 0 0 0 0
32 38 1 0 0 0 0
37 39 1 0 0 0 0
36 40 1 0 0 0 0
33 21 1 0 0 0 0
38 41 1 0 0 0 0
42 41 2 0 0 0 0
41 43 1 0 0 0 0
43 44 2 0 0 0 0
44 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 45 1 0 0 0 0
48 51 1 0 0 0 0
49 52 1 0 0 0 0
53 54 1 0 0 0 0
35 53 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 53 54
M SBL 1 1 59
M SMT 1 ^ CH2OH
M SBV 1 59 0.0967 0.8258
S SKP 5
ID FL5FAAGL0089
FORMULA C36H36O18
EXACTMASS 756.190164348
AVERAGEMASS 756.6602399999999
SMILES C(C(O)1)(O)C(OC(OC(=C(c(c6)ccc(O)c6)5)C(=O)c(c4O5)c(cc(c4)OC(O3)C(C(C(C3C)O)O)O)O)C1OC(C=Cc(c2)ccc(c2O)O)=O)CO
M END