Mol:FL5FAAGL0066
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.8036 1.6235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8036 1.6235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8036 0.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8036 0.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2473 0.6600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2473 0.6600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6910 0.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6910 0.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6910 1.6235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6910 1.6235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2473 1.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2473 1.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1347 0.6600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1347 0.6600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5784 0.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5784 0.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5784 1.6235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5784 1.6235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1347 1.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1347 1.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1347 0.1591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1347 0.1591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0223 1.9446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0223 1.9446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5447 1.6172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5447 1.6172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1117 1.9446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1117 1.9446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1117 2.5993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1117 2.5993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5447 2.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5447 2.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0223 2.5993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0223 2.5993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0697 0.4309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0697 0.4309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1152 0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1152 0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7275 0.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7275 0.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4730 0.4639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4730 0.4639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2229 0.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2229 0.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6107 0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6107 0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8652 0.7093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8652 0.7093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3881 0.7274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3881 0.7274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1340 1.1749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1340 1.1749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0970 0.7920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0970 0.7920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6785 2.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6785 2.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2473 0.0178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2473 0.0178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4145 1.8220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4145 1.8220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8519 0.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8519 0.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1975 -0.3477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1975 -0.3477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9508 -1.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9508 -1.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1603 -1.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1603 -1.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4145 -1.7393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4145 -1.7393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8889 -2.3777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8889 -2.3777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2435 -2.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2435 -2.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9265 -1.8285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9265 -1.8285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2925 -1.8285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2925 -1.8285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9755 -2.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9755 -2.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2925 -2.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2925 -2.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9265 -2.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9265 -2.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 19 20 1 0 0 0 0 | + | 19 20 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 20 18 1 0 0 0 0 | + | 20 18 1 0 0 0 0 |
| − | 18 8 1 0 0 0 0 | + | 18 8 1 0 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 15 28 1 0 0 0 0 | + | 15 28 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 1 30 1 0 0 0 0 | + | 1 30 1 0 0 0 0 |
| − | 22 31 1 0 0 0 0 | + | 22 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 2 0 0 0 0 | + | 33 34 2 0 0 0 0 |
| − | 33 35 1 0 0 0 0 | + | 33 35 1 0 0 0 0 |
| − | 35 36 2 0 0 0 0 | + | 35 36 2 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 2 0 0 0 0 | + | 37 38 2 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 40 2 0 0 0 0 | + | 39 40 2 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 2 0 0 0 0 | + | 41 42 2 0 0 0 0 |
| − | 42 37 1 0 0 0 0 | + | 42 37 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FAAGL0066 | + | ID FL5FAAGL0066 |
| − | KNApSAcK_ID C00005848 | + | KNApSAcK_ID C00005848 |
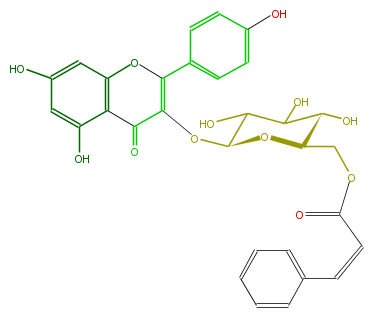
| − | NAME Kaempferol 3-(6''-(Z)-cinnamylglucoside) | + | NAME Kaempferol 3-(6''-(Z)-cinnamylglucoside) |
| − | CAS_RN 134779-23-0 | + | CAS_RN 134779-23-0 |
| − | FORMULA C30H26O12 | + | FORMULA C30H26O12 |
| − | EXACTMASS 578.1424262959999 | + | EXACTMASS 578.1424262959999 |
| − | AVERAGEMASS 578.5202400000001 | + | AVERAGEMASS 578.5202400000001 |
| − | SMILES C(=CC(OCC(O2)C(O)C(O)C(O)C(OC(=C3c(c5)ccc(c5)O)C(c(c(O)4)c(cc(O)c4)O3)=O)2)=O)c(c1)cccc1 | + | SMILES C(=CC(OCC(O2)C(O)C(O)C(O)C(OC(=C3c(c5)ccc(c5)O)C(c(c(O)4)c(cc(O)c4)O3)=O)2)=O)c(c1)cccc1 |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-2.8036 1.6235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8036 0.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2473 0.6600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6910 0.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6910 1.6235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2473 1.9447 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1347 0.6600 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5784 0.9812 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5784 1.6235 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1347 1.9447 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1347 0.1591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0223 1.9446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5447 1.6172 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1117 1.9446 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1117 2.5993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5447 2.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0223 2.5993 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0697 0.4309 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1152 0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7275 0.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4730 0.4639 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2229 0.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6107 0.9223 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8652 0.7093 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3881 0.7274 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1340 1.1749 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0970 0.7920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6785 2.9265 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2473 0.0178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4145 1.8220 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8519 0.2508 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1975 -0.3477 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9508 -1.0990 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1603 -1.0990 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4145 -1.7393 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8889 -2.3777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2435 -2.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9265 -1.8285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2925 -1.8285 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9755 -2.3775 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2925 -2.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9265 -2.9266 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
19 20 1 0 0 0 0
20 21 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
24 26 1 0 0 0 0
23 27 1 0 0 0 0
20 18 1 0 0 0 0
18 8 1 0 0 0 0
22 21 1 1 0 0 0
15 28 1 0 0 0 0
3 29 1 0 0 0 0
1 30 1 0 0 0 0
22 31 1 0 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 2 0 0 0 0
33 35 1 0 0 0 0
35 36 2 0 0 0 0
36 37 1 0 0 0 0
37 38 2 0 0 0 0
38 39 1 0 0 0 0
39 40 2 0 0 0 0
40 41 1 0 0 0 0
41 42 2 0 0 0 0
42 37 1 0 0 0 0
S SKP 8
ID FL5FAAGL0066
KNApSAcK_ID C00005848
NAME Kaempferol 3-(6''-(Z)-cinnamylglucoside)
CAS_RN 134779-23-0
FORMULA C30H26O12
EXACTMASS 578.1424262959999
AVERAGEMASS 578.5202400000001
SMILES C(=CC(OCC(O2)C(O)C(O)C(O)C(OC(=C3c(c5)ccc(c5)O)C(c(c(O)4)c(cc(O)c4)O3)=O)2)=O)c(c1)cccc1
M END