Mol:FL5FAAGL0045
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.4996 1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4996 1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4996 0.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4996 0.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7851 -0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7851 -0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0707 0.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0707 0.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0707 1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0707 1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7851 1.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7851 1.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6438 -0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6438 -0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3582 0.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3582 0.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3582 1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3582 1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6438 1.6283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6438 1.6283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6438 -0.6647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6438 -0.6647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0723 1.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0723 1.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8004 1.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8004 1.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5285 1.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5285 1.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5285 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5285 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8004 2.8894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8004 2.8894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0723 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0723 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2137 1.6282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2137 1.6282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2564 2.8893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2564 2.8893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1132 -0.0480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1132 -0.0480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7851 -0.8462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7851 -0.8462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9131 -1.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9131 -1.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4820 -2.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4820 -2.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3111 -1.9250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3111 -1.9250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1451 -2.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1451 -2.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5763 -1.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5763 -1.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7472 -1.6521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7472 -1.6521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1124 -1.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1124 -1.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3140 0.5528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3140 0.5528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8829 -0.1940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8829 -0.1940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7120 0.0429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7120 0.0429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5460 -0.1940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5460 -0.1940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9772 0.5528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9772 0.5528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1481 0.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1481 0.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5133 0.3382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5133 0.3382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6764 0.9677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6764 0.9677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5954 0.4483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5954 0.4483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2195 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2195 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4820 -0.7129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4820 -0.7129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.9443 -2.0178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.9443 -2.0178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7925 -2.6125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7925 -2.6125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1451 -2.8894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1451 -2.8894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9154 1.5748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9154 1.5748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3947 0.8875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3947 0.8875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6449 1.1790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6449 1.1790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8635 1.1704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8635 1.1704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4472 1.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4472 1.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2131 1.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2131 1.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5954 1.2736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5954 1.2736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1432 0.9497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1432 0.9497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8840 0.7041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8840 0.7041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9025 1.9945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9025 1.9945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9849 1.4648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9849 1.4648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 26 39 1 0 0 0 0 | + | 26 39 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 23 40 1 0 0 0 0 | + | 23 40 1 0 0 0 0 |
| − | 24 41 1 0 0 0 0 | + | 24 41 1 0 0 0 0 |
| − | 25 42 1 0 0 0 0 | + | 25 42 1 0 0 0 0 |
| − | 30 20 1 0 0 0 0 | + | 30 20 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 45 1 1 0 0 0 | + | 44 45 1 1 0 0 0 |
| − | 46 45 1 1 0 0 0 | + | 46 45 1 1 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 1 0 0 0 0 | + | 47 48 1 0 0 0 0 |
| − | 48 43 1 0 0 0 0 | + | 48 43 1 0 0 0 0 |
| − | 43 49 1 0 0 0 0 | + | 43 49 1 0 0 0 0 |
| − | 44 50 1 0 0 0 0 | + | 44 50 1 0 0 0 0 |
| − | 45 51 1 0 0 0 0 | + | 45 51 1 0 0 0 0 |
| − | 46 18 1 0 0 0 0 | + | 46 18 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 48 52 1 0 0 0 0 | + | 48 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 52 53 | + | M SAL 1 2 52 53 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 58 0.6894 -0.5569 | + | M SBV 1 58 0.6894 -0.5569 |
| − | S SKP 5 | + | S SKP 5 |
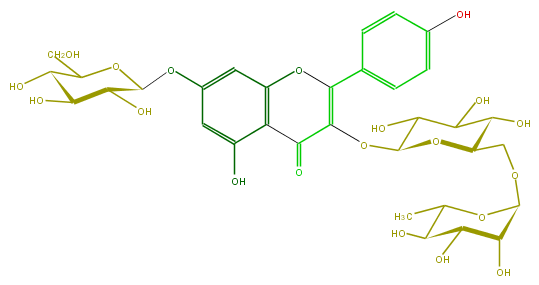
| − | ID FL5FAAGL0045 | + | ID FL5FAAGL0045 |
| − | FORMULA C33H40O20 | + | FORMULA C33H40O20 |
| − | EXACTMASS 756.21129372 | + | EXACTMASS 756.21129372 |
| − | AVERAGEMASS 756.6587 | + | AVERAGEMASS 756.6587 |
| − | SMILES C(=C(c(c6)ccc(c6)O)1)(OC(O4)C(O)C(C(C4COC(O5)C(C(C(O)C5C)O)O)O)O)C(c(c3O)c(cc(c3)OC(C(O)2)OC(CO)C(O)C2O)O1)=O | + | SMILES C(=C(c(c6)ccc(c6)O)1)(OC(O4)C(O)C(C(C4COC(O5)C(C(C(O)C5C)O)O)O)O)C(c(c3O)c(cc(c3)OC(C(O)2)OC(CO)C(O)C2O)O1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-1.4996 1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4996 0.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7851 -0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0707 0.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0707 1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7851 1.6283 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6438 -0.0216 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3582 0.3909 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3582 1.2159 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6438 1.6283 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6438 -0.6647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0723 1.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8004 1.2078 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5285 1.6282 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.5285 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8004 2.8894 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0723 2.4690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2137 1.6282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2564 2.8893 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1132 -0.0480 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7851 -0.8462 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9131 -1.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4820 -2.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3111 -1.9250 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1451 -2.1619 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.5763 -1.4151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7472 -1.6521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1124 -1.6297 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3140 0.5528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8829 -0.1940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7120 0.0429 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5460 -0.1940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9772 0.5528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1481 0.3158 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5133 0.3382 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.6764 0.9677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5954 0.4483 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2195 -0.0530 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4820 -0.7129 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.9443 -2.0178 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7925 -2.6125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.1451 -2.8894 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9154 1.5748 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3947 0.8875 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6449 1.1790 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8635 1.1704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4472 1.7128 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2131 1.4377 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5954 1.2736 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.1432 0.9497 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8840 0.7041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9025 1.9945 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9849 1.4648 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
29 30 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
32 38 1 0 0 0 0
26 39 1 0 0 0 0
38 39 1 0 0 0 0
23 40 1 0 0 0 0
24 41 1 0 0 0 0
25 42 1 0 0 0 0
30 20 1 0 0 0 0
43 44 1 1 0 0 0
44 45 1 1 0 0 0
46 45 1 1 0 0 0
46 47 1 0 0 0 0
47 48 1 0 0 0 0
48 43 1 0 0 0 0
43 49 1 0 0 0 0
44 50 1 0 0 0 0
45 51 1 0 0 0 0
46 18 1 0 0 0 0
52 53 1 0 0 0 0
48 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 52 53
M SBL 1 1 58
M SMT 1 ^CH2OH
M SBV 1 58 0.6894 -0.5569
S SKP 5
ID FL5FAAGL0045
FORMULA C33H40O20
EXACTMASS 756.21129372
AVERAGEMASS 756.6587
SMILES C(=C(c(c6)ccc(c6)O)1)(OC(O4)C(O)C(C(C4COC(O5)C(C(C(O)C5C)O)O)O)O)C(c(c3O)c(cc(c3)OC(C(O)2)OC(CO)C(O)C2O)O1)=O
M END