Mol:FL5FAAGL0036
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 53 58 0 0 0 0 0 0 0 0999 V2000 | + | 53 58 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1684 1.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1684 1.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1684 0.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1684 0.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4673 0.2827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4673 0.2827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7663 0.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7663 0.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7663 1.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7663 1.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4673 1.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4673 1.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0653 0.2827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0653 0.2827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6357 0.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6357 0.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6357 1.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6357 1.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0653 1.9016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0653 1.9016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0653 -0.3485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0653 -0.3485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3365 1.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3365 1.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0510 1.4889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0510 1.4889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7655 1.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7655 1.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7655 2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7655 2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0510 3.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0510 3.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3365 2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3365 2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4673 -0.5266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4673 -0.5266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8691 1.9015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8691 1.9015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4797 3.1388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4797 3.1388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4432 0.1960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4432 0.1960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0437 -0.9320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0437 -0.9320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1976 -0.4436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1976 -0.4436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4661 -1.3829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4661 -1.3829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1976 -2.3280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1976 -2.3280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0437 -2.8166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0437 -2.8166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7753 -1.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7753 -1.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8831 -0.1898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8831 -0.1898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3703 -2.4465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3703 -2.4465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4805 0.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4805 0.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.2094 -0.1261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.2094 -0.1261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9041 0.3833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9041 0.3833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.6501 0.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.6501 0.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9607 0.9900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9607 0.9900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3783 0.4501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3783 0.4501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6091 -0.2282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6091 -0.2282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7352 0.0644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7352 0.0644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0968 0.8887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0968 0.8887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4294 2.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4294 2.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6378 1.6342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6378 1.6342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8890 2.5132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8890 2.5132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6378 3.3974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6378 3.3974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4294 3.8546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4294 3.8546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1783 2.9756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1783 2.9756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1871 4.4717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1871 4.4717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7200 4.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7200 4.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7859 3.4538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7859 3.4538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0968 2.0757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0968 2.0757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8495 -3.4068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8495 -3.4068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7129 1.3076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7129 1.3076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1643 2.4422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1643 2.4422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1779 -3.2129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1779 -3.2129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1180 -4.4717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1180 -4.4717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 31 36 1 0 0 0 0 | + | 31 36 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 28 30 1 0 0 0 0 | + | 28 30 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 40 39 1 1 0 0 0 | + | 40 39 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | 43 44 1 1 0 0 0 | + | 43 44 1 1 0 0 0 |
| − | 44 39 1 1 0 0 0 | + | 44 39 1 1 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 42 46 1 0 0 0 0 | + | 42 46 1 0 0 0 0 |
| − | 44 47 1 0 0 0 0 | + | 44 47 1 0 0 0 0 |
| − | 39 48 1 0 0 0 0 | + | 39 48 1 0 0 0 0 |
| − | 40 19 1 0 0 0 0 | + | 40 19 1 0 0 0 0 |
| − | 26 49 1 0 0 0 0 | + | 26 49 1 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 34 50 1 0 0 0 0 | + | 34 50 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 25 52 1 0 0 0 0 | + | 25 52 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 50 51 | + | M SAL 1 2 50 51 |
| − | M SBL 1 1 56 | + | M SBL 1 1 56 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SBV 1 56 -0.7522 -0.3177 | + | M SBV 1 56 -0.7522 -0.3177 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 52 53 | + | M SAL 2 2 52 53 |
| − | M SBL 2 1 58 | + | M SBL 2 1 58 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 58 0.0197 0.8849 | + | M SBV 2 58 0.0197 0.8849 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGL0036 | + | ID FL5FAAGL0036 |
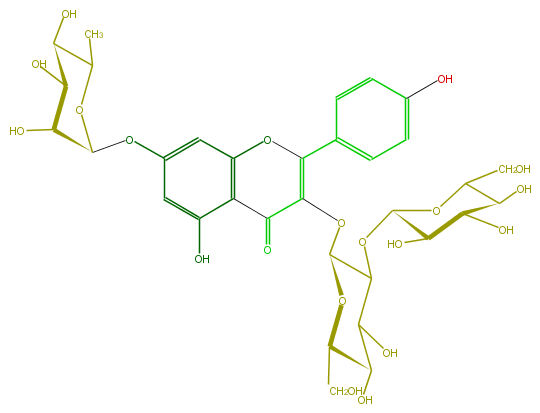
| − | FORMULA C33H40O20 | + | FORMULA C33H40O20 |
| − | EXACTMASS 756.21129372 | + | EXACTMASS 756.21129372 |
| − | AVERAGEMASS 756.6587 | + | AVERAGEMASS 756.6587 |
| − | SMILES O(C(O6)C(C(O)C(C(CO)6)O)OC(O5)C(C(O)C(C(CO)5)O)O)C(=C3c(c4)ccc(O)c4)C(=O)c(c(O3)1)c(cc(OC(C2O)OC(C)C(C2O)O)c1)O | + | SMILES O(C(O6)C(C(O)C(C(CO)6)O)OC(O5)C(C(O)C(C(CO)5)O)O)C(=C3c(c4)ccc(O)c4)C(=O)c(c(O3)1)c(cc(OC(C2O)OC(C)C(C2O)O)c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
53 58 0 0 0 0 0 0 0 0999 V2000
-2.1684 1.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1684 0.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4673 0.2827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7663 0.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7663 1.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4673 1.9016 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0653 0.2827 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6357 0.6873 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6357 1.4969 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0653 1.9016 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0653 -0.3485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3365 1.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0510 1.4889 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7655 1.9015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.7655 2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0510 3.1390 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3365 2.7265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4673 -0.5266 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8691 1.9015 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4797 3.1388 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4432 0.1960 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.0437 -0.9320 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1976 -0.4436 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4661 -1.3829 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1976 -2.3280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0437 -2.8166 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7753 -1.8771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8831 -0.1898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3703 -2.4465 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4805 0.4404 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.2094 -0.1261 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9041 0.3833 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.6501 0.5918 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9607 0.9900 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3783 0.4501 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.6091 -0.2282 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7352 0.0644 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0968 0.8887 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.4294 2.0913 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6378 1.6342 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8890 2.5132 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6378 3.3974 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4294 3.8546 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1783 2.9756 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.1871 4.4717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7200 4.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7859 3.4538 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0968 2.0757 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8495 -3.4068 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7129 1.3076 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.1643 2.4422 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1779 -3.2129 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1180 -4.4717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
8 21 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
23 21 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
31 36 1 0 0 0 0
32 37 1 0 0 0 0
28 30 1 0 0 0 0
33 38 1 0 0 0 0
40 39 1 1 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 1 0 0 0 0
43 44 1 1 0 0 0
44 39 1 1 0 0 0
43 45 1 0 0 0 0
42 46 1 0 0 0 0
44 47 1 0 0 0 0
39 48 1 0 0 0 0
40 19 1 0 0 0 0
26 49 1 0 0 0 0
50 51 1 0 0 0 0
34 50 1 0 0 0 0
52 53 1 0 0 0 0
25 52 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 50 51
M SBL 1 1 56
M SMT 1 CH2OH
M SBV 1 56 -0.7522 -0.3177
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 52 53
M SBL 2 1 58
M SMT 2 ^ CH2OH
M SBV 2 58 0.0197 0.8849
S SKP 5
ID FL5FAAGL0036
FORMULA C33H40O20
EXACTMASS 756.21129372
AVERAGEMASS 756.6587
SMILES O(C(O6)C(C(O)C(C(CO)6)O)OC(O5)C(C(O)C(C(CO)5)O)O)C(=C3c(c4)ccc(O)c4)C(=O)c(c(O3)1)c(cc(OC(C2O)OC(C)C(C2O)O)c1)O
M END