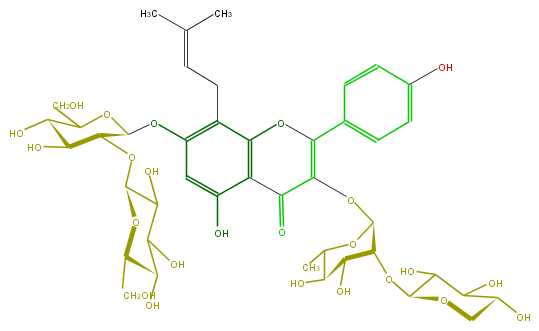
Mol:FL5FAAGI0013
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 67 73 0 0 0 0 0 0 0 0999 V2000 | + | 67 73 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8965 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8965 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8965 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8965 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1782 -0.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1782 -0.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4599 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4599 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4599 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4599 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1782 1.2898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1782 1.2898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2584 -0.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2584 -0.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9767 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9767 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9767 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9767 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2584 1.2898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2584 1.2898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2823 -1.1904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2823 -1.1904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6948 1.2895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6948 1.2895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4268 0.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4268 0.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1589 1.2895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1589 1.2895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1589 2.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1589 2.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4268 2.5576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4268 2.5576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6948 2.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6948 2.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6146 1.2895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6146 1.2895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8908 2.5575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8908 2.5575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8376 -0.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8376 -0.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1782 -1.1983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1782 -1.1983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1782 2.1189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1782 2.1189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8963 2.5334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8963 2.5334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8963 3.3626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8963 3.3626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1782 3.7771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1782 3.7771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6143 3.7771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6143 3.7771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0284 1.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0284 1.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5491 0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5491 0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8590 0.9915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8590 0.9915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1931 0.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1931 0.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6769 1.4827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6769 1.4827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2807 1.1640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2807 1.1640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6537 1.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6537 1.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2477 0.7449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2477 0.7449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1169 0.5051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1169 0.5051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7457 -2.1670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7457 -2.1670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1506 -2.6663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1506 -2.6663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9256 -2.7212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9256 -2.7212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5588 -3.1788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5588 -3.1788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1539 -2.6796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1539 -2.6796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3789 -2.6246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3789 -2.6246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1887 -2.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1887 -2.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.0115 -2.3366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.0115 -2.3366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.6537 -3.0942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.6537 -3.0942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2427 -1.6151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2427 -1.6151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2427 -2.2759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2427 -2.2759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7021 -1.8010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7021 -1.8010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3464 -1.6386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3464 -1.6386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3464 -0.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3464 -0.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8870 -1.4527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8870 -1.4527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5819 -2.5202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5819 -2.5202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6957 -2.3453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6957 -2.3453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8063 -1.9734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8063 -1.9734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6920 -2.2431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6920 -2.2431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5471 -0.5805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5471 -0.5805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2616 -0.1679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2616 -0.1679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0348 -0.9612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0348 -0.9612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2616 -1.7591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2616 -1.7591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5471 -2.1717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5471 -2.1717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7737 -1.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7737 -1.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7524 0.1856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7524 0.1856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1822 -1.9125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1822 -1.9125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7456 -2.8361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7456 -2.8361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9660 1.6965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9660 1.6965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3304 2.6976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3304 2.6976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3517 -2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3517 -2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8759 -3.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8759 -3.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 15 19 1 0 0 0 0 | + | 15 19 1 0 0 0 0 |
| − | 8 20 1 0 0 0 0 | + | 8 20 1 0 0 0 0 |
| − | 3 21 1 0 0 0 0 | + | 3 21 1 0 0 0 0 |
| − | 6 22 1 0 0 0 0 | + | 6 22 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 2 0 0 0 0 | + | 23 24 2 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 24 26 1 0 0 0 0 | + | 24 26 1 0 0 0 0 |
| − | 27 28 1 1 0 0 0 | + | 27 28 1 1 0 0 0 |
| − | 28 29 1 1 0 0 0 | + | 28 29 1 1 0 0 0 |
| − | 30 29 1 1 0 0 0 | + | 30 29 1 1 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 27 1 0 0 0 0 | + | 32 27 1 0 0 0 0 |
| − | 27 33 1 0 0 0 0 | + | 27 33 1 0 0 0 0 |
| − | 28 34 1 0 0 0 0 | + | 28 34 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 18 1 0 0 0 0 | + | 30 18 1 0 0 0 0 |
| − | 36 37 1 0 0 0 0 | + | 36 37 1 0 0 0 0 |
| − | 37 38 1 1 0 0 0 | + | 37 38 1 1 0 0 0 |
| − | 38 39 1 1 0 0 0 | + | 38 39 1 1 0 0 0 |
| − | 40 39 1 1 0 0 0 | + | 40 39 1 1 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 36 1 0 0 0 0 | + | 41 36 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 41 43 1 0 0 0 0 | + | 41 43 1 0 0 0 0 |
| − | 40 44 1 0 0 0 0 | + | 40 44 1 0 0 0 0 |
| − | 45 46 1 0 0 0 0 | + | 45 46 1 0 0 0 0 |
| − | 46 47 1 1 0 0 0 | + | 46 47 1 1 0 0 0 |
| − | 47 48 1 1 0 0 0 | + | 47 48 1 1 0 0 0 |
| − | 49 48 1 1 0 0 0 | + | 49 48 1 1 0 0 0 |
| − | 49 50 1 0 0 0 0 | + | 49 50 1 0 0 0 0 |
| − | 50 45 1 0 0 0 0 | + | 50 45 1 0 0 0 0 |
| − | 47 51 1 0 0 0 0 | + | 47 51 1 0 0 0 0 |
| − | 46 52 1 0 0 0 0 | + | 46 52 1 0 0 0 0 |
| − | 45 53 1 0 0 0 0 | + | 45 53 1 0 0 0 0 |
| − | 48 54 1 0 0 0 0 | + | 48 54 1 0 0 0 0 |
| − | 49 20 1 0 0 0 0 | + | 49 20 1 0 0 0 0 |
| − | 37 54 1 0 0 0 0 | + | 37 54 1 0 0 0 0 |
| − | 55 56 1 0 0 0 0 | + | 55 56 1 0 0 0 0 |
| − | 56 57 1 1 0 0 0 | + | 56 57 1 1 0 0 0 |
| − | 57 58 1 1 0 0 0 | + | 57 58 1 1 0 0 0 |
| − | 59 58 1 1 0 0 0 | + | 59 58 1 1 0 0 0 |
| − | 59 60 1 0 0 0 0 | + | 59 60 1 0 0 0 0 |
| − | 60 55 1 0 0 0 0 | + | 60 55 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 60 62 1 0 0 0 0 | + | 60 62 1 0 0 0 0 |
| − | 59 63 1 0 0 0 0 | + | 59 63 1 0 0 0 0 |
| − | 56 35 1 0 0 0 0 | + | 56 35 1 0 0 0 0 |
| − | 64 65 1 0 0 0 0 | + | 64 65 1 0 0 0 0 |
| − | 32 64 1 0 0 0 0 | + | 32 64 1 0 0 0 0 |
| − | 66 67 1 0 0 0 0 | + | 66 67 1 0 0 0 0 |
| − | 58 66 1 0 0 0 0 | + | 58 66 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 64 65 | + | M SAL 1 2 64 65 |
| − | M SBL 1 1 71 | + | M SBL 1 1 71 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 71 0.6853 -0.5325 | + | M SBV 1 71 0.6853 -0.5325 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 66 67 | + | M SAL 2 2 66 67 |
| − | M SBL 2 1 73 | + | M SBL 2 1 73 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 73 0.0901 0.8379 | + | M SBV 2 73 0.0901 0.8379 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGI0013 | + | ID FL5FAAGI0013 |
| − | FORMULA C43H56O24 | + | FORMULA C43H56O24 |
| − | EXACTMASS 956.31615272 | + | EXACTMASS 956.31615272 |
| − | AVERAGEMASS 956.89034 | + | AVERAGEMASS 956.89034 |
| − | SMILES c(c(O)3)c(c(c(O6)c3C(C(=C6c(c7)ccc(c7)O)OC(C(OC(C5O)OCC(C5O)O)4)OC(C(O)C(O)4)C)=O)CC=C(C)C)OC(C(OC(O2)C(C(C(O)C2CO)O)O)1)OC(CO)C(C(O)1)O | + | SMILES c(c(O)3)c(c(c(O6)c3C(C(=C6c(c7)ccc(c7)O)OC(C(OC(C5O)OCC(C5O)O)4)OC(C(O)C(O)4)C)=O)CC=C(C)C)OC(C(OC(O2)C(C(C(O)C2CO)O)O)1)OC(CO)C(C(O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
67 73 0 0 0 0 0 0 0 0999 V2000
-1.8965 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8965 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1782 -0.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4599 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4599 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1782 1.2898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2584 -0.3692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9767 0.0455 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9767 0.8750 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2584 1.2898 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2823 -1.1904 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6948 1.2895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4268 0.8669 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1589 1.2895 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1589 2.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4268 2.5576 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6948 2.1349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6146 1.2895 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8908 2.5575 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8376 -0.4580 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1782 -1.1983 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1782 2.1189 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8963 2.5334 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8963 3.3626 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1782 3.7771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6143 3.7771 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0284 1.3557 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5491 0.7230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8590 0.9915 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.1931 0.9987 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6769 1.4827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2807 1.1640 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6537 1.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2477 0.7449 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1169 0.5051 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7457 -2.1670 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1506 -2.6663 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.9256 -2.7212 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5588 -3.1788 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1539 -2.6796 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3789 -2.6246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1887 -2.0620 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.0115 -2.3366 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.6537 -3.0942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.2427 -1.6151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2427 -2.2759 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7021 -1.8010 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3464 -1.6386 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3464 -0.9778 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8870 -1.4527 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5819 -2.5202 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6957 -2.3453 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8063 -1.9734 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6920 -2.2431 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.5471 -0.5805 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2616 -0.1679 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0348 -0.9612 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2616 -1.7591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5471 -2.1717 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7737 -1.3785 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7524 0.1856 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1822 -1.9125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.7456 -2.8361 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9660 1.6965 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3304 2.6976 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3517 -2.5970 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8759 -3.7771 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
15 19 1 0 0 0 0
8 20 1 0 0 0 0
3 21 1 0 0 0 0
6 22 1 0 0 0 0
22 23 1 0 0 0 0
23 24 2 0 0 0 0
24 25 1 0 0 0 0
24 26 1 0 0 0 0
27 28 1 1 0 0 0
28 29 1 1 0 0 0
30 29 1 1 0 0 0
30 31 1 0 0 0 0
31 32 1 0 0 0 0
32 27 1 0 0 0 0
27 33 1 0 0 0 0
28 34 1 0 0 0 0
29 35 1 0 0 0 0
30 18 1 0 0 0 0
36 37 1 0 0 0 0
37 38 1 1 0 0 0
38 39 1 1 0 0 0
40 39 1 1 0 0 0
40 41 1 0 0 0 0
41 36 1 0 0 0 0
36 42 1 0 0 0 0
41 43 1 0 0 0 0
40 44 1 0 0 0 0
45 46 1 0 0 0 0
46 47 1 1 0 0 0
47 48 1 1 0 0 0
49 48 1 1 0 0 0
49 50 1 0 0 0 0
50 45 1 0 0 0 0
47 51 1 0 0 0 0
46 52 1 0 0 0 0
45 53 1 0 0 0 0
48 54 1 0 0 0 0
49 20 1 0 0 0 0
37 54 1 0 0 0 0
55 56 1 0 0 0 0
56 57 1 1 0 0 0
57 58 1 1 0 0 0
59 58 1 1 0 0 0
59 60 1 0 0 0 0
60 55 1 0 0 0 0
55 61 1 0 0 0 0
60 62 1 0 0 0 0
59 63 1 0 0 0 0
56 35 1 0 0 0 0
64 65 1 0 0 0 0
32 64 1 0 0 0 0
66 67 1 0 0 0 0
58 66 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 64 65
M SBL 1 1 71
M SMT 1 ^ CH2OH
M SBV 1 71 0.6853 -0.5325
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 66 67
M SBL 2 1 73
M SMT 2 ^ CH2OH
M SBV 2 73 0.0901 0.8379
S SKP 5
ID FL5FAAGI0013
FORMULA C43H56O24
EXACTMASS 956.31615272
AVERAGEMASS 956.89034
SMILES c(c(O)3)c(c(c(O6)c3C(C(=C6c(c7)ccc(c7)O)OC(C(OC(C5O)OCC(C5O)O)4)OC(C(O)C(O)4)C)=O)CC=C(C)C)OC(C(OC(O2)C(C(C(O)C2CO)O)O)1)OC(CO)C(C(O)1)O
M END