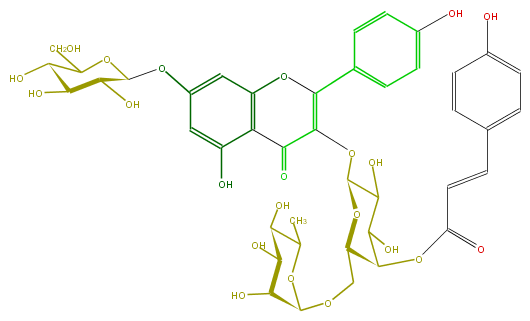
Mol:FL5FAAGA0043
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 64 70 0 0 0 0 0 0 0 0999 V2000 | + | 64 70 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.8237 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8237 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8237 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8237 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1093 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1093 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3949 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3949 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3949 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3949 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1093 1.9677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1093 1.9677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3196 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3196 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0341 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0341 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0341 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0341 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3196 1.9677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3196 1.9677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3196 -0.3254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3196 -0.3254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9336 2.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9336 2.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6618 1.7061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6618 1.7061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3899 2.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3899 2.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3899 2.9673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3899 2.9673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6618 3.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6618 3.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9336 2.9673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9336 2.9673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1093 -0.5069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1093 -0.5069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1692 3.4173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1692 3.4173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4859 2.1380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4859 2.1380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8850 0.2004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8850 0.2004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4777 -0.8281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4777 -0.8281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7671 -0.4179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7671 -0.4179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9926 -1.2067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9926 -1.2067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7671 -2.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7671 -2.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4777 -2.4106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4777 -2.4106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2523 -1.6217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2523 -1.6217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3381 -0.0071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3381 -0.0071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4216 -2.2250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4216 -2.2250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9127 -2.7746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9127 -2.7746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0167 -3.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0167 -3.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6912 -3.4173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6912 -3.4173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4772 -2.6683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4772 -2.6683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6912 -1.9150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6912 -1.9150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0167 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0167 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2306 -2.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2306 -2.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2009 -0.9933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2009 -0.9933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5069 -1.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5069 -1.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3334 -1.9070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3334 -1.9070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6343 -3.0581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6343 -3.0581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3335 -3.2450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3335 -3.2450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7100 -1.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7100 -1.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0784 -1.5683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0784 -1.5683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8261 -2.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8261 -2.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0784 -0.5848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0784 -0.5848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9837 -0.2542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9837 -0.2542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9766 0.7294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9766 0.7294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7316 1.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7316 1.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.7316 2.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.7316 2.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9766 2.4731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9766 2.4731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2215 2.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2215 2.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2215 1.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2215 1.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.9766 3.3437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.9766 3.3437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0732 2.2255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0732 2.2255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5964 1.5964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5964 1.5964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9100 1.8632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9100 1.8632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2477 1.8704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2477 1.8704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7289 2.3518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7289 2.3518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3296 2.0348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3296 2.0348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7316 1.9257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7316 1.9257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2969 1.5794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2969 1.5794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2266 1.3254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2266 1.3254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9842 2.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9842 2.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9608 2.3332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9608 2.3332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 15 1 0 0 0 0 | + | 19 15 1 0 0 0 0 |
| − | 4 3 1 0 0 0 0 | + | 4 3 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 8 1 0 0 0 0 | + | 21 8 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 26 29 1 0 0 0 0 | + | 26 29 1 0 0 0 0 |
| − | 25 30 1 0 0 0 0 | + | 25 30 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 36 31 1 1 0 0 0 | + | 36 31 1 1 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 36 39 1 0 0 0 0 | + | 36 39 1 0 0 0 0 |
| − | 31 40 1 0 0 0 0 | + | 31 40 1 0 0 0 0 |
| − | 32 41 1 0 0 0 0 | + | 32 41 1 0 0 0 0 |
| − | 41 30 1 0 0 0 0 | + | 41 30 1 0 0 0 0 |
| − | 27 42 1 0 0 0 0 | + | 27 42 1 0 0 0 0 |
| − | 29 43 1 0 0 0 0 | + | 29 43 1 0 0 0 0 |
| − | 43 44 2 0 0 0 0 | + | 43 44 2 0 0 0 0 |
| − | 43 45 1 0 0 0 0 | + | 43 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 47 1 0 0 0 0 | + | 52 47 1 0 0 0 0 |
| − | 50 53 1 0 0 0 0 | + | 50 53 1 0 0 0 0 |
| − | 54 55 1 1 0 0 0 | + | 54 55 1 1 0 0 0 |
| − | 55 56 1 1 0 0 0 | + | 55 56 1 1 0 0 0 |
| − | 57 56 1 1 0 0 0 | + | 57 56 1 1 0 0 0 |
| − | 57 58 1 0 0 0 0 | + | 57 58 1 0 0 0 0 |
| − | 58 59 1 0 0 0 0 | + | 58 59 1 0 0 0 0 |
| − | 59 54 1 0 0 0 0 | + | 59 54 1 0 0 0 0 |
| − | 54 60 1 0 0 0 0 | + | 54 60 1 0 0 0 0 |
| − | 55 61 1 0 0 0 0 | + | 55 61 1 0 0 0 0 |
| − | 56 62 1 0 0 0 0 | + | 56 62 1 0 0 0 0 |
| − | 57 20 1 0 0 0 0 | + | 57 20 1 0 0 0 0 |
| − | 63 64 1 0 0 0 0 | + | 63 64 1 0 0 0 0 |
| − | 59 63 1 0 0 0 0 | + | 59 63 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 63 64 | + | M SAL 1 2 63 64 |
| − | M SBL 1 1 70 | + | M SBL 1 1 70 |
| − | M SMT 1 ^CH2OH | + | M SMT 1 ^CH2OH |
| − | M SBV 1 70 0.6546 -0.5726 | + | M SBV 1 70 0.6546 -0.5726 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL5FAAGA0043 | + | ID FL5FAAGA0043 |
| − | FORMULA C42H46O22 | + | FORMULA C42H46O22 |
| − | EXACTMASS 902.248073156 | + | EXACTMASS 902.248073156 |
| − | AVERAGEMASS 902.80144 | + | AVERAGEMASS 902.80144 |
| − | SMILES O=C(OC(C(COC(O7)C(C(C(O)C7C)O)O)6)C(O)C(O)C(O6)OC(C(=O)2)=C(c(c5)ccc(O)c5)Oc(c3)c(c(O)cc3OC(O4)C(C(O)C(C4CO)O)O)2)C=Cc(c1)ccc(c1)O | + | SMILES O=C(OC(C(COC(O7)C(C(C(O)C7C)O)O)6)C(O)C(O)C(O6)OC(C(=O)2)=C(c(c5)ccc(O)c5)Oc(c3)c(c(O)cc3OC(O4)C(C(O)C(C4CO)O)O)2)C=Cc(c1)ccc(c1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
64 70 0 0 0 0 0 0 0 0999 V2000
-1.8237 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8237 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1093 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3949 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3949 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1093 1.9677 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3196 0.3179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0341 0.7302 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0341 1.5552 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3196 1.9677 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.3196 -0.3254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9336 2.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6618 1.7061 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3899 2.1265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3899 2.9673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6618 3.3876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9336 2.9673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1093 -0.5069 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1692 3.4173 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4859 2.1380 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8850 0.2004 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4777 -0.8281 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7671 -0.4179 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9926 -1.2067 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7671 -2.0003 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4777 -2.4106 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2523 -1.6217 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3381 -0.0071 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4216 -2.2250 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.9127 -2.7746 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0167 -3.0278 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6912 -3.4173 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4772 -2.6683 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6912 -1.9150 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0167 -1.5254 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2306 -2.2743 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2009 -0.9933 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5069 -1.3272 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3334 -1.9070 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6343 -3.0581 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3335 -3.2450 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7100 -1.9919 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0784 -1.5683 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8261 -2.0000 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.0784 -0.5848 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9837 -0.2542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9766 0.7294 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7316 1.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.7316 2.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9766 2.4731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2215 2.0371 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.2215 1.1653 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.9766 3.3437 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0732 2.2255 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5964 1.5964 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9100 1.8632 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2477 1.8704 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.7289 2.3518 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.3296 2.0348 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7316 1.9257 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.2969 1.5794 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.2266 1.3254 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9842 2.6074 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9608 2.3332 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 15 1 0 0 0 0
4 3 1 0 0 0 0
1 20 1 0 0 0 0
21 8 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
26 29 1 0 0 0 0
25 30 1 0 0 0 0
23 21 1 0 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 36 1 1 0 0 0
36 31 1 1 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
36 39 1 0 0 0 0
31 40 1 0 0 0 0
32 41 1 0 0 0 0
41 30 1 0 0 0 0
27 42 1 0 0 0 0
29 43 1 0 0 0 0
43 44 2 0 0 0 0
43 45 1 0 0 0 0
45 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 47 1 0 0 0 0
50 53 1 0 0 0 0
54 55 1 1 0 0 0
55 56 1 1 0 0 0
57 56 1 1 0 0 0
57 58 1 0 0 0 0
58 59 1 0 0 0 0
59 54 1 0 0 0 0
54 60 1 0 0 0 0
55 61 1 0 0 0 0
56 62 1 0 0 0 0
57 20 1 0 0 0 0
63 64 1 0 0 0 0
59 63 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 63 64
M SBL 1 1 70
M SMT 1 ^CH2OH
M SBV 1 70 0.6546 -0.5726
S SKP 5
ID FL5FAAGA0043
FORMULA C42H46O22
EXACTMASS 902.248073156
AVERAGEMASS 902.80144
SMILES O=C(OC(C(COC(O7)C(C(C(O)C7C)O)O)6)C(O)C(O)C(O6)OC(C(=O)2)=C(c(c5)ccc(O)c5)Oc(c3)c(c(O)cc3OC(O4)C(C(O)C(C4CO)O)O)2)C=Cc(c1)ccc(c1)O
M END