Mol:FL5FAAGA0010
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 1.7045 -4.8813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7045 -4.8813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3371 -4.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3371 -4.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5569 -4.1662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5569 -4.1662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1439 -3.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1439 -3.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5113 -3.7856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5113 -3.7856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2916 -4.3893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2916 -4.3893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3636 -3.0705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3636 -3.0705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.9507 -2.5784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9507 -2.5784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3181 -2.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3181 -2.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.0984 -3.2936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.0984 -3.2936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8567 -2.9834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8567 -2.9834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9054 -2.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9054 -2.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1293 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1293 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7085 -1.0813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7085 -1.0813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.0637 -1.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.0637 -1.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1602 -1.8102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1602 -1.8102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2606 -2.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2606 -2.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1891 -4.0546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1891 -4.0546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4849 -5.4847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4849 -5.4847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3570 -0.6936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3570 -0.6936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3359 -1.8314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3359 -1.8314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7865 -0.4809 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.7865 -0.4809 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.3989 -1.1523 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.3989 -1.1523 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.1443 -0.9393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1443 -0.9393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8943 -1.1523 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 3.8943 -1.1523 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 4.2820 -0.4809 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 4.2820 -0.4809 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 3.5365 -0.6939 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 3.5365 -0.6939 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 2.5317 0.2194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5317 0.2194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8435 -0.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8435 -0.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.2820 0.2674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.2820 0.2674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3098 -8.3140 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | 2.3098 -8.3140 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | 2.9288 -7.7860 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.9288 -7.7860 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.4821 -6.7966 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.4821 -6.7966 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 2.3500 -6.0924 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | 2.3500 -6.0924 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | 1.9276 -6.6956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.9276 -6.6956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3780 -7.2758 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | 2.3780 -7.2758 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | 1.4305 -8.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4305 -8.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0734 -8.2625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0734 -8.2625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1115 -6.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1115 -6.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7204 -1.4822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7204 -1.4822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.8636 -2.4719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.8636 -2.4719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4306 -7.7002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4306 -7.7002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1449 -7.2876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1449 -7.2876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 1 19 1 0 0 0 0 | + | 1 19 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 22 1 0 0 0 0 | + | 27 22 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 27 29 1 0 0 0 0 | + | 27 29 1 0 0 0 0 |
| − | 26 30 1 0 0 0 0 | + | 26 30 1 0 0 0 0 |
| − | 23 21 1 0 0 0 0 | + | 23 21 1 0 0 0 0 |
| − | 8 21 1 0 0 0 0 | + | 8 21 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 34 19 1 0 0 0 0 | + | 34 19 1 0 0 0 0 |
| − | 25 40 1 0 0 0 0 | + | 25 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 -3.365 0.2689 | + | M SVB 2 46 -3.365 0.2689 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 4.7204 -1.4822 | + | M SVB 1 44 4.7204 -1.4822 |
| − | S SKP 8 | + | S SKP 8 |
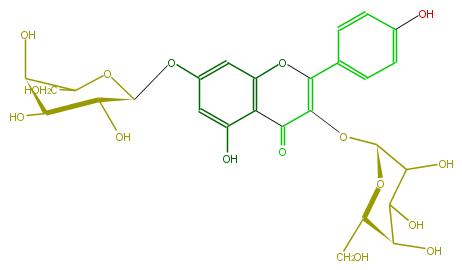
| − | ID FL5FAAGA0010 | + | ID FL5FAAGA0010 |
| − | KNApSAcK_ID C00005177 | + | KNApSAcK_ID C00005177 |
| − | NAME Kaempferol 3,7-digalactoside | + | NAME Kaempferol 3,7-digalactoside |
| − | CAS_RN 54557-66-3 | + | CAS_RN 54557-66-3 |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES O([C@@H]([C@@H](O)5)OC(CO)[C@H](O)[C@@H]5O)c(c4)cc(c(c41)C(=O)C(O[C@@H](C(O)3)O[C@@H]([C@@H](C(O)3)O)CO)=C(c(c2)ccc(c2)O)O1)O | + | SMILES O([C@@H]([C@@H](O)5)OC(CO)[C@H](O)[C@@H]5O)c(c4)cc(c(c41)C(=O)C(O[C@@H](C(O)3)O[C@@H]([C@@H](C(O)3)O)CO)=C(c(c2)ccc(c2)O)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
1.7045 -4.8813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3371 -4.7698 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5569 -4.1662 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1439 -3.6741 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5113 -3.7856 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2916 -4.3893 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3636 -3.0705 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.9507 -2.5784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3181 -2.6899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.0984 -3.2936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8567 -2.9834 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.9054 -2.1980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1293 -1.5828 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7085 -1.0813 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.0637 -1.1949 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1602 -1.8102 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.2606 -2.3117 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1891 -4.0546 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.4849 -5.4847 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3570 -0.6936 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3359 -1.8314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7865 -0.4809 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.3989 -1.1523 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.1443 -0.9393 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8943 -1.1523 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
4.2820 -0.4809 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
3.5365 -0.6939 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
2.5317 0.2194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.8435 -0.1623 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.2820 0.2674 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3098 -8.3140 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
2.9288 -7.7860 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.4821 -6.7966 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
2.3500 -6.0924 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
1.9276 -6.6956 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3780 -7.2758 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
1.4305 -8.4690 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.0734 -8.2625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.1115 -6.2537 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.7204 -1.4822 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.8636 -2.4719 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4306 -7.7002 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1449 -7.2876 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
1 19 1 0 0 0 0
15 20 1 0 0 0 0
22 23 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 22 1 0 0 0 0
22 28 1 0 0 0 0
27 29 1 0 0 0 0
26 30 1 0 0 0 0
23 21 1 0 0 0 0
8 21 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 39 1 0 0 0 0
34 19 1 0 0 0 0
25 40 1 0 0 0 0
40 41 1 0 0 0 0
36 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 -3.365 0.2689
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 4.7204 -1.4822
S SKP 8
ID FL5FAAGA0010
KNApSAcK_ID C00005177
NAME Kaempferol 3,7-digalactoside
CAS_RN 54557-66-3
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES O([C@@H]([C@@H](O)5)OC(CO)[C@H](O)[C@@H]5O)c(c4)cc(c(c41)C(=O)C(O[C@@H](C(O)3)O[C@@H]([C@@H](C(O)3)O)CO)=C(c(c2)ccc(c2)O)O1)O
M END