Mol:FL5FA9GS0004
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 32 35 0 0 0 0 0 0 0 0999 V2000 | + | 32 35 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.1008 0.1513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1008 0.1513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1008 -0.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1008 -0.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6571 -0.8123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6571 -0.8123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2134 -0.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2134 -0.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2134 0.1513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2134 0.1513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6571 0.4725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6571 0.4725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7697 -0.8123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7697 -0.8123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3260 -0.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3260 -0.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3260 0.1513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3260 0.1513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7697 0.4725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7697 0.4725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.7697 -1.3131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.7697 -1.3131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8821 0.4723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8821 0.4723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4491 0.1450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4491 0.1450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0161 0.4723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0161 0.4723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0161 1.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0161 1.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4491 1.4544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4491 1.4544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8821 1.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8821 1.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4553 0.4723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4553 0.4723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3023 0.4607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3023 0.4607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7867 -0.2200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7867 -0.2200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0442 0.0688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0442 0.0688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3277 0.0765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3277 0.0765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8483 0.5973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8483 0.5973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4979 0.2544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4979 0.2544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0161 0.0485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0161 0.0485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5905 -0.2591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5905 -0.2591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6187 -0.6454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6187 -0.6454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6571 -1.4544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6571 -1.4544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.7193 -0.7318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.7193 -0.7318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4338 -1.1443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4338 -1.1443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8885 0.6507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8885 0.6507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4719 1.2341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4719 1.2341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 1 18 1 0 0 0 0 | + | 1 18 1 0 0 0 0 |
| − | 19 20 1 1 0 0 0 | + | 19 20 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 22 21 1 1 0 0 0 | + | 22 21 1 1 0 0 0 |
| − | 22 23 1 0 0 0 0 | + | 22 23 1 0 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 19 25 1 0 0 0 0 | + | 19 25 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 18 1 0 0 0 0 | + | 22 18 1 0 0 0 0 |
| − | 3 28 1 0 0 0 0 | + | 3 28 1 0 0 0 0 |
| − | 8 29 1 0 0 0 0 | + | 8 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 24 31 1 0 0 0 0 | + | 24 31 1 0 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 29 30 | + | M SAL 1 2 29 30 |
| − | M SBL 1 1 32 | + | M SBL 1 1 32 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 32 -5.7924 4.5306 | + | M SBV 1 32 -5.7924 4.5306 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 31 32 | + | M SAL 2 2 31 32 |
| − | M SBL 2 1 34 | + | M SBL 2 1 34 |
| − | M SMT 2 ^CH2OH | + | M SMT 2 ^CH2OH |
| − | M SBV 2 34 -6.5762 5.1676 | + | M SBV 2 34 -6.5762 5.1676 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL5FA9GS0004 | + | ID FL5FA9GS0004 |
| − | KNApSAcK_ID C00005125 | + | KNApSAcK_ID C00005125 |
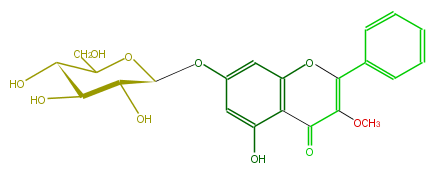
| − | NAME Galangin 3-methyl ether 7-glucoside | + | NAME Galangin 3-methyl ether 7-glucoside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C22H22O10 | + | FORMULA C22H22O10 |
| − | EXACTMASS 446.121296924 | + | EXACTMASS 446.121296924 |
| − | AVERAGEMASS 446.40408 | + | AVERAGEMASS 446.40408 |
| − | SMILES C(C1O)(C(C(OC1Oc(c2)cc(O3)c(C(=O)C(=C(c(c4)cccc4)3)OC)c(O)2)CO)O)O | + | SMILES C(C1O)(C(C(OC1Oc(c2)cc(O3)c(C(=O)C(=C(c(c4)cccc4)3)OC)c(O)2)CO)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
32 35 0 0 0 0 0 0 0 0999 V2000
0.1008 0.1513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1008 -0.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6571 -0.8123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2134 -0.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2134 0.1513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6571 0.4725 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7697 -0.8123 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3260 -0.4911 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3260 0.1513 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.7697 0.4725 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.7697 -1.3131 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8821 0.4723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4491 0.1450 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0161 0.4723 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0161 1.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4491 1.4544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8821 1.1270 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4553 0.4723 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3023 0.4607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7867 -0.2200 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0442 0.0688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3277 0.0765 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8483 0.5973 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.4979 0.2544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0161 0.0485 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.5905 -0.2591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6187 -0.6454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6571 -1.4544 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.7193 -0.7318 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.4338 -1.1443 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8885 0.6507 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4719 1.2341 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
1 18 1 0 0 0 0
19 20 1 1 0 0 0
20 21 1 1 0 0 0
22 21 1 1 0 0 0
22 23 1 0 0 0 0
23 24 1 0 0 0 0
24 19 1 0 0 0 0
19 25 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 18 1 0 0 0 0
3 28 1 0 0 0 0
8 29 1 0 0 0 0
29 30 1 0 0 0 0
24 31 1 0 0 0 0
31 32 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 29 30
M SBL 1 1 32
M SMT 1 OCH3
M SBV 1 32 -5.7924 4.5306
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 31 32
M SBL 2 1 34
M SMT 2 ^CH2OH
M SBV 2 34 -6.5762 5.1676
S SKP 8
ID FL5FA9GS0004
KNApSAcK_ID C00005125
NAME Galangin 3-methyl ether 7-glucoside
CAS_RN -
FORMULA C22H22O10
EXACTMASS 446.121296924
AVERAGEMASS 446.40408
SMILES C(C1O)(C(C(OC1Oc(c2)cc(O3)c(C(=O)C(=C(c(c4)cccc4)3)OC)c(O)2)CO)O)O
M END