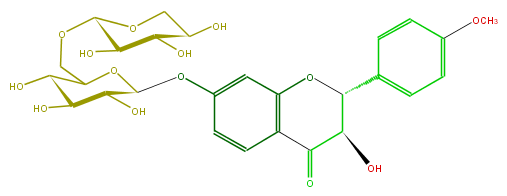
Mol:FL4D1AGS0002
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | -1.2450 -0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2450 -0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5306 -1.1903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5306 -1.1903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1838 -0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1838 -0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1838 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1838 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5306 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5306 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2450 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2450 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8983 -1.1903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8983 -1.1903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6127 -0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6127 -0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6127 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6127 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8983 0.4597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8983 0.4597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.8983 -1.9088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.8983 -1.9088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4078 0.4579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4078 0.4579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1329 0.0392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1329 0.0392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8581 0.4579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8581 0.4579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8581 1.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8581 1.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1329 1.7138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1329 1.7138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.4078 1.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.4078 1.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9762 0.4694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9762 0.4694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2549 -1.5687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2549 -1.5687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9011 0.4692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9011 0.4692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3920 -0.2027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3920 -0.2027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6589 0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6589 0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9514 0.0899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9514 0.0899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4655 0.6041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4655 0.6041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1068 0.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1068 0.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.5632 0.2466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.5632 0.2466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0398 -0.2428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0398 -0.2428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0105 -0.2942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0105 -0.2942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9079 1.7629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9079 1.7629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3569 1.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3569 1.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5635 1.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5635 1.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7979 1.3524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7979 1.3524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3542 1.9088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3542 1.9088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0484 1.5425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0484 1.5425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6280 1.4036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6280 1.4036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0049 0.9929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0049 0.9929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9839 0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9839 0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6568 0.6688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6568 0.6688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1962 1.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1962 1.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5832 1.7138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5832 1.7138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.5632 1.1481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.5632 1.1481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 6 0 0 0 | + | 9 12 1 6 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 6 18 1 0 0 0 0 | + | 6 18 1 0 0 0 0 |
| − | 8 19 1 1 0 0 0 | + | 8 19 1 1 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 18 1 0 0 0 0 | + | 23 18 1 0 0 0 0 |
| − | 29 30 1 1 0 0 0 | + | 29 30 1 1 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 32 31 1 1 0 0 0 | + | 32 31 1 1 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 29 35 1 0 0 0 0 | + | 29 35 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 38 25 1 0 0 0 0 | + | 38 25 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 15 40 1 0 0 0 0 | + | 15 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 45 -0.7251 -0.4186 | + | M SBV 1 45 -0.7251 -0.4186 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL4D1AGS0002 | + | ID FL4D1AGS0002 |
| − | FORMULA C27H32O14 | + | FORMULA C27H32O14 |
| − | EXACTMASS 580.179205732 | + | EXACTMASS 580.179205732 |
| − | AVERAGEMASS 580.53458 | + | AVERAGEMASS 580.53458 |
| − | SMILES C(C(C(O)2)OC(Oc(c3)cc(O4)c(C(C(C4c(c5)ccc(c5)OC)O)=O)c3)C(C2O)O)OC(O1)C(O)C(C(O)C1)O | + | SMILES C(C(C(O)2)OC(Oc(c3)cc(O4)c(C(C(C4c(c5)ccc(c5)OC)O)=O)c3)C(C2O)O)OC(O1)C(O)C(C(O)C1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
-1.2450 -0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5306 -1.1903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1838 -0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.1838 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5306 0.4597 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2450 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8983 -1.1903 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6127 -0.7777 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6127 0.0471 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.8983 0.4597 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.8983 -1.9088 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.4078 0.4579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1329 0.0392 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8581 0.4579 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8581 1.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1329 1.7138 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.4078 1.2952 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9762 0.4694 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.2549 -1.5687 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9011 0.4692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3920 -0.2027 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6589 0.0823 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9514 0.0899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4655 0.6041 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1068 0.2656 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.5632 0.2466 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0398 -0.2428 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0105 -0.2942 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.9079 1.7629 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.3569 1.0356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5635 1.3442 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7979 1.3524 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3542 1.9088 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.0484 1.5425 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6280 1.4036 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.0049 0.9929 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.9839 0.9922 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.6568 0.6688 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1962 1.6009 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.5832 1.7138 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.5632 1.1481 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 4 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 6 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
6 18 1 0 0 0 0
8 19 1 1 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 18 1 0 0 0 0
29 30 1 1 0 0 0
30 31 1 1 0 0 0
32 31 1 1 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 29 1 0 0 0 0
29 35 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
35 38 1 0 0 0 0
38 25 1 0 0 0 0
32 39 1 0 0 0 0
40 41 1 0 0 0 0
15 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 OCH3
M SBV 1 45 -0.7251 -0.4186
S SKP 5
ID FL4D1AGS0002
FORMULA C27H32O14
EXACTMASS 580.179205732
AVERAGEMASS 580.53458
SMILES C(C(C(O)2)OC(Oc(c3)cc(O4)c(C(C(C4c(c5)ccc(c5)OC)O)=O)c3)C(C2O)O)OC(O1)C(O)C(C(O)C1)O
M END