Mol:FL3FFAGS0011
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 43 47 0 0 0 0 0 0 0 0999 V2000 | + | 43 47 0 0 0 0 0 0 0 0999 V2000 |
| − | 0.3171 0.9109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3171 0.9109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.3171 0.3488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.3171 0.3488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7682 0.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7682 0.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2192 0.3488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2192 0.3488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.2192 0.8696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.2192 0.8696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7682 1.1300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7682 1.1300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6703 0.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6703 0.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1214 0.3488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1214 0.3488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1214 0.8696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1214 0.8696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6703 1.1300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6703 1.1300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8420 -0.2796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8420 -0.2796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5723 1.1299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5723 1.1299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0320 0.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0320 0.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4917 1.1299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4917 1.1299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.4917 1.6608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.4917 1.6608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0320 1.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0320 1.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.5723 1.6608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.5723 1.6608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7682 -0.4314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7682 -0.4314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1441 1.1772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1441 1.1772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.9514 1.9261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.9514 1.9261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9144 1.0155 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -2.9144 1.0155 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -2.3987 0.3349 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -2.3987 0.3349 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.6562 0.6236 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -1.6562 0.6236 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.9398 0.6313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.9398 0.6313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -1.4604 1.1521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4604 1.1521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2189 0.8797 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -2.2189 0.8797 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -3.4909 0.6827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4909 0.6827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1106 -0.0402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1106 -0.0402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9688 0.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9688 0.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7071 -2.7756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7071 -2.7756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6512 -1.7919 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 | + | -1.6512 -1.7919 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0 |
| − | -0.7983 -1.8343 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.7983 -1.8343 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.5423 -1.0799 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.5423 -1.0799 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.0877 -0.5265 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 | + | -0.0877 -0.5265 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0 |
| − | -0.8213 -0.5902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8213 -0.5902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1002 -1.3464 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 | + | -1.1002 -1.3464 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0 |
| − | -1.2002 -2.3133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2002 -2.3133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2782 -1.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2782 -1.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7867 1.6494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7867 1.6494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8280 1.4867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8280 1.4867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8999 2.3085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8999 2.3085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8690 -0.9544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8690 -0.9544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7895 -1.3448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7895 -1.3448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 9 12 1 0 0 0 0 | + | 9 12 1 0 0 0 0 |
| − | 12 13 2 0 0 0 0 | + | 12 13 2 0 0 0 0 |
| − | 13 14 1 0 0 0 0 | + | 13 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 12 1 0 0 0 0 | + | 17 12 1 0 0 0 0 |
| − | 3 18 1 0 0 0 0 | + | 3 18 1 0 0 0 0 |
| − | 19 1 1 0 0 0 0 | + | 19 1 1 0 0 0 0 |
| − | 15 20 1 0 0 0 0 | + | 15 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 36 1 0 0 0 0 | + | 35 36 1 0 0 0 0 |
| − | 36 31 1 0 0 0 0 | + | 36 31 1 0 0 0 0 |
| − | 32 37 1 0 0 0 0 | + | 32 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 34 29 1 0 0 0 0 | + | 34 29 1 0 0 0 0 |
| − | 24 19 1 0 0 0 0 | + | 24 19 1 0 0 0 0 |
| − | 6 39 1 0 0 0 0 | + | 6 39 1 0 0 0 0 |
| − | 26 40 1 0 0 0 0 | + | 26 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 36 42 1 0 0 0 0 | + | 36 42 1 0 0 0 0 |
| − | 42 43 1 0 0 0 0 | + | 42 43 1 0 0 0 0 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 42 43 | + | M SAL 2 2 42 43 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 CH2OH | + | M SMT 2 CH2OH |
| − | M SVB 2 46 -2.6603 -0.115 | + | M SVB 2 46 -2.6603 -0.115 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 CH2OH | + | M SMT 1 CH2OH |
| − | M SVB 1 44 -2.5144 1.6872 | + | M SVB 1 44 -2.5144 1.6872 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FFAGS0011 | + | ID FL3FFAGS0011 |
| − | KNApSAcK_ID C00004497 | + | KNApSAcK_ID C00004497 |
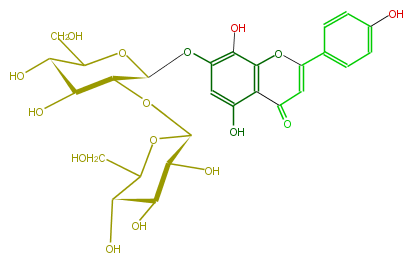
| − | NAME 8-Hydroxyapigenin 7-sophoroside | + | NAME 8-Hydroxyapigenin 7-sophoroside |
| − | CAS_RN - | + | CAS_RN - |
| − | FORMULA C27H30O16 | + | FORMULA C27H30O16 |
| − | EXACTMASS 610.153384912 | + | EXACTMASS 610.153384912 |
| − | AVERAGEMASS 610.5175 | + | AVERAGEMASS 610.5175 |
| − | SMILES O[C@@H]([C@@H](O)1)[C@H](O[C@@H]([C@H]2O)[C@H](Oc(c3)c(c(O4)c(C(C=C4c(c5)ccc(O)c5)=O)c(O)3)O)OC([C@@H]2O)CO)OC([C@@H]1O)CO | + | SMILES O[C@@H]([C@@H](O)1)[C@H](O[C@@H]([C@H]2O)[C@H](Oc(c3)c(c(O4)c(C(C=C4c(c5)ccc(O)c5)=O)c(O)3)O)OC([C@@H]2O)CO)OC([C@@H]1O)CO |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
43 47 0 0 0 0 0 0 0 0999 V2000
0.3171 0.9109 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.3171 0.3488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7682 0.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2192 0.3488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.2192 0.8696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7682 1.1300 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6703 0.0883 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1214 0.3488 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1214 0.8696 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6703 1.1300 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8420 -0.2796 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.5723 1.1299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0320 0.8645 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4917 1.1299 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.4917 1.6608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0320 1.9262 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.5723 1.6608 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7682 -0.4314 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1441 1.1772 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.9514 1.9261 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9144 1.0155 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-2.3987 0.3349 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.6562 0.6236 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.9398 0.6313 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-1.4604 1.1521 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2189 0.8797 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-3.4909 0.6827 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1106 -0.0402 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.9688 0.1561 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.7071 -2.7756 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.6512 -1.7919 0.0000 C 0 0 2 0 0 0 0 0 0 0 0 0
-0.7983 -1.8343 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.5423 -1.0799 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.0877 -0.5265 0.0000 C 0 0 1 0 0 0 0 0 0 0 0 0
-0.8213 -0.5902 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1002 -1.3464 0.0000 C 0 0 3 0 0 0 0 0 0 0 0 0
-1.2002 -2.3133 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2782 -1.2134 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7867 1.6494 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8280 1.4867 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8999 2.3085 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.8690 -0.9544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7895 -1.3448 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
9 12 1 0 0 0 0
12 13 2 0 0 0 0
13 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 12 1 0 0 0 0
3 18 1 0 0 0 0
19 1 1 0 0 0 0
15 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 36 1 0 0 0 0
36 31 1 0 0 0 0
32 37 1 0 0 0 0
33 38 1 0 0 0 0
30 31 1 0 0 0 0
34 29 1 0 0 0 0
24 19 1 0 0 0 0
6 39 1 0 0 0 0
26 40 1 0 0 0 0
40 41 1 0 0 0 0
36 42 1 0 0 0 0
42 43 1 0 0 0 0
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 42 43
M SBL 2 1 46
M SMT 2 CH2OH
M SVB 2 46 -2.6603 -0.115
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 44
M SMT 1 CH2OH
M SVB 1 44 -2.5144 1.6872
S SKP 8
ID FL3FFAGS0011
KNApSAcK_ID C00004497
NAME 8-Hydroxyapigenin 7-sophoroside
CAS_RN -
FORMULA C27H30O16
EXACTMASS 610.153384912
AVERAGEMASS 610.5175
SMILES O[C@@H]([C@@H](O)1)[C@H](O[C@@H]([C@H]2O)[C@H](Oc(c3)c(c(O4)c(C(C=C4c(c5)ccc(O)c5)=O)c(O)3)O)OC([C@@H]2O)CO)OC([C@@H]1O)CO
M END