Mol:FL3FEAGS0042
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 41 45 0 0 0 0 0 0 0 0999 V2000 | + | 41 45 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.7418 1.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7418 1.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4425 1.6357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4425 1.6357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.4425 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.4425 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.7418 2.8495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.7418 2.8495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0409 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0409 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.0409 1.6357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.0409 1.6357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9871 2.7593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9871 2.7593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3261 1.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3261 1.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6117 1.6355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6117 1.6355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8972 1.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8972 1.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.8972 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.8972 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6117 -0.0144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6117 -0.0144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.3261 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.3261 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1722 1.6415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1722 1.6415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4470 1.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4470 1.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.4470 0.3857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.4470 0.3857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1722 -0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1722 -0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.6117 -0.5574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.6117 -0.5574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0482 -0.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0482 -0.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.1722 -0.6468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.1722 -0.6468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8914 -1.0652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8914 -1.0652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0910 -1.5005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0910 -1.5005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4597 -0.8435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4597 -0.8435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5817 -0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5817 -0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3821 -0.1646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3821 -0.1646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0135 -0.8215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0135 -0.8215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.5789 -1.0897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.5789 -1.0897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8450 -1.7565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8450 -1.7565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4921 -1.6658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4921 -1.6658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0254 -0.9695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0254 -0.9695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9297 -1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9297 -1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2020 -1.6298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2020 -1.6298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8554 -2.4724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8554 -2.4724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.9510 -2.3607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.9510 -2.3607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.6787 -1.8122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.6787 -1.8122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3407 -0.6542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3407 -0.6542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9696 -0.3532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9696 -0.3532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3780 -2.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3780 -2.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9871 -1.6654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9871 -1.6654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2555 1.5955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2555 1.5955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5335 1.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5335 1.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 6 8 1 0 0 0 0 | + | 6 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 11 2 0 0 0 0 | + | 10 11 2 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 13 1 0 0 0 0 | + | 12 13 1 0 0 0 0 |
| − | 13 8 2 0 0 0 0 | + | 13 8 2 0 0 0 0 |
| − | 10 14 1 0 0 0 0 | + | 10 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 11 1 0 0 0 0 | + | 17 11 1 0 0 0 0 |
| − | 12 18 2 0 0 0 0 | + | 12 18 2 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 19 24 1 0 0 0 0 | + | 19 24 1 0 0 0 0 |
| − | 23 27 1 0 0 0 0 | + | 23 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 21 29 1 0 0 0 0 | + | 21 29 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 29 34 1 0 0 0 0 | + | 29 34 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 33 38 1 0 0 0 0 | + | 33 38 1 0 0 0 0 |
| − | 32 39 1 0 0 0 0 | + | 32 39 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 15 40 1 0 0 0 0 | + | 15 40 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 40 41 | + | M SAL 1 2 40 41 |
| − | M SBL 1 1 45 | + | M SBL 1 1 45 |
| − | M SMT 1 ^OCH3 | + | M SMT 1 ^OCH3 |
| − | M SBV 1 45 0.7025 -0.3726 | + | M SBV 1 45 0.7025 -0.3726 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FEAGS0042 | + | ID FL3FEAGS0042 |
| − | FORMULA C27H30O14 | + | FORMULA C27H30O14 |
| − | EXACTMASS 578.163555668 | + | EXACTMASS 578.163555668 |
| − | AVERAGEMASS 578.5187000000001 | + | AVERAGEMASS 578.5187000000001 |
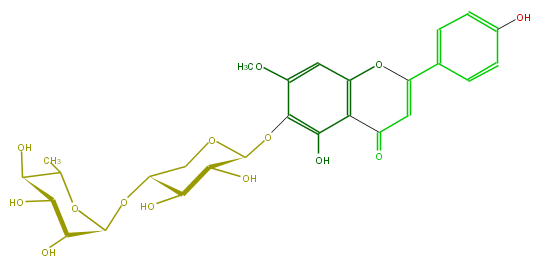
| − | SMILES C(C(O)1)(C(O)C(OC1OC(C5)C(O)C(O)C(O5)Oc(c(OC)4)c(O)c(c(c4)2)C(=O)C=C(c(c3)ccc(c3)O)O2)C)O | + | SMILES C(C(O)1)(C(O)C(OC1OC(C5)C(O)C(O)C(O5)Oc(c(OC)4)c(O)c(c(c4)2)C(=O)C=C(c(c3)ccc(c3)O)O2)C)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
41 45 0 0 0 0 0 0 0 0999 V2000
4.7418 1.2310 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4425 1.6357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.4425 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.7418 2.8495 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0409 2.4449 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.0409 1.6357 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9871 2.7593 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.3261 1.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6117 1.6355 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.8972 1.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.8972 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6117 -0.0144 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.3261 0.3980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1722 1.6415 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4470 1.2230 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.4470 0.3857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.1722 -0.0330 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.6117 -0.5574 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0482 -0.1095 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.1722 -0.6468 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8914 -1.0652 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0910 -1.5005 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4597 -0.8435 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5817 -0.5999 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3821 -0.1646 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0135 -0.8215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.5789 -1.0897 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8450 -1.7565 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.4921 -1.6658 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.0254 -0.9695 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9297 -1.0814 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2020 -1.6298 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8554 -2.4724 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.9510 -2.3607 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.6787 -1.8122 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3407 -0.6542 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9696 -0.3532 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.3780 -2.8495 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-5.9871 -1.6654 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2555 1.5955 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.5335 1.1399 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
3 7 1 0 0 0 0
6 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 1 0 0 0 0
10 11 2 0 0 0 0
11 12 1 0 0 0 0
12 13 1 0 0 0 0
13 8 2 0 0 0 0
10 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 11 1 0 0 0 0
12 18 2 0 0 0 0
16 19 1 0 0 0 0
17 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
19 24 1 0 0 0 0
23 27 1 0 0 0 0
22 28 1 0 0 0 0
21 29 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
29 34 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
33 38 1 0 0 0 0
32 39 1 0 0 0 0
40 41 1 0 0 0 0
15 40 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 40 41
M SBL 1 1 45
M SMT 1 ^OCH3
M SBV 1 45 0.7025 -0.3726
S SKP 5
ID FL3FEAGS0042
FORMULA C27H30O14
EXACTMASS 578.163555668
AVERAGEMASS 578.5187000000001
SMILES C(C(O)1)(C(O)C(OC1OC(C5)C(O)C(O)C(O5)Oc(c(OC)4)c(O)c(c(c4)2)C(=O)C=C(c(c3)ccc(c3)O)O2)C)O
M END