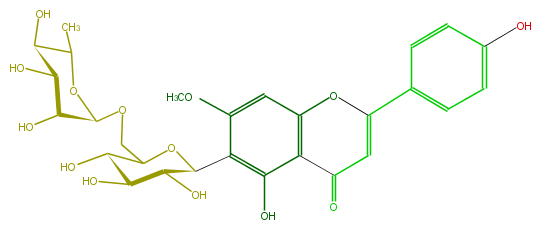
Mol:FL3FCACS0018
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.8400 -0.0203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8400 -0.0203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8400 -0.8453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8400 -0.8453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1255 -1.2578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1255 -1.2578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5890 -0.8453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5890 -0.8453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.5890 -0.0203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.5890 -0.0203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1255 0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1255 0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3036 -1.2578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3036 -1.2578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0181 -0.8453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0181 -0.8453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.0181 -0.0203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.0181 -0.0203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3036 0.3923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3036 0.3923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.3036 -1.9012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.3036 -1.9012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1255 -2.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1255 -2.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8914 0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8914 0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6443 0.0899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6443 0.0899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3973 0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3973 0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.3973 1.3940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.3973 1.3940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.6443 1.8288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.6443 1.8288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8914 1.3940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8914 1.3940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.1498 1.8286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.1498 1.8286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.3750 -0.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.3750 -0.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8982 -1.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8982 -1.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2117 -1.1806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2117 -1.1806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5492 -1.1735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5492 -1.1735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0306 -0.6920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0306 -0.6920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6312 -1.0090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6312 -1.0090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0909 -1.0789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0909 -1.0789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6524 -1.3717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6524 -1.3717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5488 -1.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5488 -1.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0822 -0.6181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0822 -0.6181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3657 -0.0080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3657 -0.0080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5985 -0.1432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5985 -0.1432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0557 0.4874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0557 0.4874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.1125 1.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.1125 1.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.8795 1.4041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.8795 1.4041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.4222 0.7733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.4222 0.7733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7669 2.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7669 2.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2317 1.8046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2317 1.8046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1498 0.9647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1498 0.9647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.9714 -0.2446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.9714 -0.2446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0509 0.0934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0509 0.0934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5656 0.3607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5656 0.3607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2077 1.4731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2077 1.4731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 25 29 1 0 0 0 0 | + | 25 29 1 0 0 0 0 |
| − | 31 30 1 1 0 0 0 | + | 31 30 1 1 0 0 0 |
| − | 31 32 1 0 0 0 0 | + | 31 32 1 0 0 0 0 |
| − | 32 33 1 0 0 0 0 | + | 32 33 1 0 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 30 1 1 0 0 0 | + | 35 30 1 1 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 33 37 1 0 0 0 0 | + | 33 37 1 0 0 0 0 |
| − | 35 38 1 0 0 0 0 | + | 35 38 1 0 0 0 0 |
| − | 30 39 1 0 0 0 0 | + | 30 39 1 0 0 0 0 |
| − | 29 40 1 0 0 0 0 | + | 29 40 1 0 0 0 0 |
| − | 40 31 1 0 0 0 0 | + | 40 31 1 0 0 0 0 |
| − | 23 2 1 0 0 0 0 | + | 23 2 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 1 41 1 0 0 0 0 | + | 1 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 41 42 | + | M SAL 1 2 41 42 |
| − | M SBL 1 1 46 | + | M SBL 1 1 46 |
| − | M SMT 1 ^ OCH3 | + | M SMT 1 ^ OCH3 |
| − | M SBV 1 46 0.7256 -0.3809 | + | M SBV 1 46 0.7256 -0.3809 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FCACS0018 | + | ID FL3FCACS0018 |
| − | FORMULA C28H32O14 | + | FORMULA C28H32O14 |
| − | EXACTMASS 592.179205732 | + | EXACTMASS 592.179205732 |
| − | AVERAGEMASS 592.54528 | + | AVERAGEMASS 592.54528 |
| − | SMILES c(c5)c(ccc5O)C(O1)=CC(c(c(O)2)c(cc(OC)c2C(O3)C(O)C(O)C(C3COC(O4)C(O)C(C(O)C4C)O)O)1)=O | + | SMILES c(c5)c(ccc5O)C(O1)=CC(c(c(O)2)c(cc(OC)c2C(O3)C(O)C(O)C(C3COC(O4)C(O)C(C(O)C4C)O)O)1)=O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-0.8400 -0.0203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.8400 -0.8453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1255 -1.2578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5890 -0.8453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.5890 -0.0203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1255 0.3923 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3036 -1.2578 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0181 -0.8453 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.0181 -0.0203 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.3036 0.3923 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.3036 -1.9012 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.1255 -2.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8914 0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6443 0.0899 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3973 0.5246 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.3973 1.3940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.6443 1.8288 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8914 1.3940 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.1498 1.8286 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.3750 -0.8182 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8982 -1.4476 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2117 -1.1806 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5492 -1.1735 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0306 -0.6920 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.6312 -1.0090 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0909 -1.0789 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.6524 -1.3717 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5488 -1.6567 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0822 -0.6181 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3657 -0.0080 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5985 -0.1432 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0557 0.4874 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.1125 1.2687 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.8795 1.4041 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.4222 0.7733 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.7669 2.0826 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2317 1.8046 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1498 0.9647 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.9714 -0.2446 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0509 0.0934 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5656 0.3607 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.2077 1.4731 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
25 29 1 0 0 0 0
31 30 1 1 0 0 0
31 32 1 0 0 0 0
32 33 1 0 0 0 0
33 34 1 0 0 0 0
34 35 1 1 0 0 0
35 30 1 1 0 0 0
34 36 1 0 0 0 0
33 37 1 0 0 0 0
35 38 1 0 0 0 0
30 39 1 0 0 0 0
29 40 1 0 0 0 0
40 31 1 0 0 0 0
23 2 1 0 0 0 0
41 42 1 0 0 0 0
1 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 41 42
M SBL 1 1 46
M SMT 1 ^ OCH3
M SBV 1 46 0.7256 -0.3809
S SKP 5
ID FL3FCACS0018
FORMULA C28H32O14
EXACTMASS 592.179205732
AVERAGEMASS 592.54528
SMILES c(c5)c(ccc5O)C(O1)=CC(c(c(O)2)c(cc(OC)c2C(O3)C(O)C(O)C(C3COC(O4)C(O)C(C(O)C4C)O)O)1)=O
M END