Mol:FL3FADGS0026
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 55 60 0 0 0 0 0 0 0 0999 V2000 | + | 55 60 0 0 0 0 0 0 0 0999 V2000 |
| − | 4.5385 -0.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5385 -0.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.5385 -1.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.5385 -1.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2530 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2530 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9674 -1.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9674 -1.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.9674 -0.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.9674 -0.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2530 0.1784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2530 0.1784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8240 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8240 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1096 -1.0591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1096 -1.0591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3951 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3951 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.3951 -2.2966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.3951 -2.2966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1096 -2.7091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1096 -2.7091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.8240 -2.2966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.8240 -2.2966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6806 -1.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6806 -1.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9661 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9661 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.9661 -2.2966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.9661 -2.2966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6806 -2.7091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6806 -2.7091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.1096 -3.4273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.1096 -3.4273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.2517 -1.0591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.2517 -1.0591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.6806 -3.3950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.6806 -3.3950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.6730 0.1675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.6730 0.1675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.5419 -1.3908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.5419 -1.3908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.2675 -0.9718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.2675 -0.9718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3332 -0.9211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3332 -0.9211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9207 -1.6356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9207 -1.6356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1226 -1.4265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1226 -1.4265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3294 -1.6533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3294 -1.6533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7418 -0.9387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7418 -0.9387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5400 -1.1477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5400 -1.1477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.6969 -0.5621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.6969 -0.5621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1766 -0.2852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1766 -0.2852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8163 -1.4042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8163 -1.4042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3692 -1.3766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3692 -1.3766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5089 -2.0662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5089 -2.0662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9783 -1.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9783 -1.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.4497 -2.5143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.4497 -2.5143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.7975 -1.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.7975 -1.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3050 -2.4931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3050 -2.4931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0850 -2.2786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0850 -2.2786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.3479 -1.4966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.3479 -1.4966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1565 -1.3332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1565 -1.3332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.7023 -1.9519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.7023 -1.9519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.4395 -2.7339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.4395 -2.7339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.6309 -2.8973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.6309 -2.8973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.2675 -1.8377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.2675 -1.8377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1766 0.5398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1766 0.5398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4621 0.9523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4621 0.9523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8911 0.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8911 0.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8911 1.7773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8911 1.7773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6055 2.1898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6055 2.1898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.6055 3.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.6055 3.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3200 3.4273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3200 3.4273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0345 3.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0345 3.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.0345 2.1898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.0345 2.1898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.3200 1.7773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.3200 1.7773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.7490 3.4273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.7490 3.4273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 2 0 0 0 0 | + | 1 2 2 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 2 0 0 0 0 | + | 5 6 2 0 0 0 0 |
| − | 6 1 1 0 0 0 0 | + | 6 1 1 0 0 0 0 |
| − | 2 7 1 0 0 0 0 | + | 2 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 11 1 0 0 0 0 | + | 10 11 1 0 0 0 0 |
| − | 11 12 1 0 0 0 0 | + | 11 12 1 0 0 0 0 |
| − | 12 7 2 0 0 0 0 | + | 12 7 2 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 10 1 0 0 0 0 | + | 16 10 1 0 0 0 0 |
| − | 14 18 1 0 0 0 0 | + | 14 18 1 0 0 0 0 |
| − | 11 17 2 0 0 0 0 | + | 11 17 2 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 5 20 1 0 0 0 0 | + | 5 20 1 0 0 0 0 |
| − | 4 21 1 0 0 0 0 | + | 4 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 26 18 1 0 0 0 0 | + | 26 18 1 0 0 0 0 |
| − | 32 34 1 0 0 0 0 | + | 32 34 1 0 0 0 0 |
| − | 34 35 2 0 0 0 0 | + | 34 35 2 0 0 0 0 |
| − | 34 36 1 0 0 0 0 | + | 34 36 1 0 0 0 0 |
| − | 36 37 2 0 0 0 0 | + | 36 37 2 0 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 2 0 0 0 0 | + | 38 39 2 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 2 0 0 0 0 | + | 40 41 2 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 38 1 0 0 0 0 | + | 43 38 1 0 0 0 0 |
| − | 41 44 1 0 0 0 0 | + | 41 44 1 0 0 0 0 |
| − | 30 45 1 0 0 0 0 | + | 30 45 1 0 0 0 0 |
| − | 45 46 2 0 0 0 0 | + | 45 46 2 0 0 0 0 |
| − | 45 47 1 0 0 0 0 | + | 45 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 51 1 0 0 0 0 | + | 50 51 1 0 0 0 0 |
| − | 51 52 2 0 0 0 0 | + | 51 52 2 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 53 54 2 0 0 0 0 | + | 53 54 2 0 0 0 0 |
| − | 54 49 1 0 0 0 0 | + | 54 49 1 0 0 0 0 |
| − | 52 55 1 0 0 0 0 | + | 52 55 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FADGS0026 | + | ID FL3FADGS0026 |
| − | KNApSAcK_ID C00013679 | + | KNApSAcK_ID C00013679 |
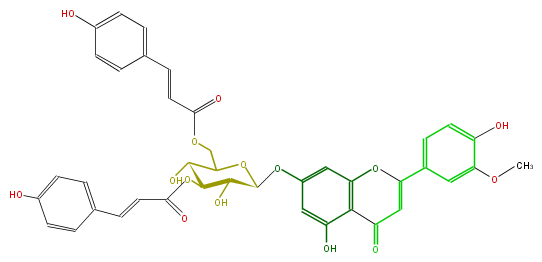
| − | NAME Chrysoeriol 7-(3'',6''-di-(E)-p-coumaroylglucoside);7-[[3,6-Bis-O-[(2E)-3-(4-hydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-5-hydroxy-2-(4-hydroxy-3-methoxyphenyl)-4H-1-benzopyran-4-one | + | NAME Chrysoeriol 7-(3'',6''-di-(E)-p-coumaroylglucoside);7-[[3,6-Bis-O-[(2E)-3-(4-hydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-5-hydroxy-2-(4-hydroxy-3-methoxyphenyl)-4H-1-benzopyran-4-one |
| − | CAS_RN 625409-67-8 | + | CAS_RN 625409-67-8 |
| − | FORMULA C40H34O15 | + | FORMULA C40H34O15 |
| − | EXACTMASS 754.1897704180001 | + | EXACTMASS 754.1897704180001 |
| − | AVERAGEMASS 754.68896 | + | AVERAGEMASS 754.68896 |
| − | SMILES c(c1)c(ccc(C=CC(=O)OCC(O2)C(O)C(OC(C=Cc(c6)ccc(c6)O)=O)C(O)C2Oc(c5)cc(O)c(c53)C(C=C(c(c4)ccc(O)c4OC)O3)=O)1)O | + | SMILES c(c1)c(ccc(C=CC(=O)OCC(O2)C(O)C(OC(C=Cc(c6)ccc(c6)O)=O)C(O)C2Oc(c5)cc(O)c(c53)C(C=C(c(c4)ccc(O)c4OC)O3)=O)1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
55 60 0 0 0 0 0 0 0 0999 V2000
4.5385 -0.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.5385 -1.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2530 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9674 -1.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.9674 -0.2341 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2530 0.1784 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8240 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1096 -1.0591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.3951 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.3951 -2.2966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1096 -2.7091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.8240 -2.2966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6806 -1.0591 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9661 -1.4716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.9661 -2.2966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.6806 -2.7091 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.1096 -3.4273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.2517 -1.0591 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.6806 -3.3950 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.6730 0.1675 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.5419 -1.3908 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.2675 -0.9718 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3332 -0.9211 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9207 -1.6356 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1226 -1.4265 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3294 -1.6533 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7418 -0.9387 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5400 -1.1477 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.6969 -0.5621 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1766 -0.2852 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8163 -1.4042 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3692 -1.3766 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5089 -2.0662 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9783 -1.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.4497 -2.5143 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.7975 -1.9857 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3050 -2.4931 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0850 -2.2786 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.3479 -1.4966 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.1565 -1.3332 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.7023 -1.9519 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-6.4395 -2.7339 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.6309 -2.8973 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-7.2675 -1.8377 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1766 0.5398 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4621 0.9523 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8911 0.9523 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8911 1.7773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6055 2.1898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.6055 3.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3200 3.4273 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0345 3.0148 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.0345 2.1898 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.3200 1.7773 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.7490 3.4273 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 2 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 2 0 0 0 0
6 1 1 0 0 0 0
2 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 11 1 0 0 0 0
11 12 1 0 0 0 0
12 7 2 0 0 0 0
9 13 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 10 1 0 0 0 0
14 18 1 0 0 0 0
11 17 2 0 0 0 0
16 19 1 0 0 0 0
5 20 1 0 0 0 0
4 21 1 0 0 0 0
21 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
26 18 1 0 0 0 0
32 34 1 0 0 0 0
34 35 2 0 0 0 0
34 36 1 0 0 0 0
36 37 2 0 0 0 0
37 38 1 0 0 0 0
38 39 2 0 0 0 0
39 40 1 0 0 0 0
40 41 2 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
43 38 1 0 0 0 0
41 44 1 0 0 0 0
30 45 1 0 0 0 0
45 46 2 0 0 0 0
45 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 51 1 0 0 0 0
51 52 2 0 0 0 0
52 53 1 0 0 0 0
53 54 2 0 0 0 0
54 49 1 0 0 0 0
52 55 1 0 0 0 0
S SKP 8
ID FL3FADGS0026
KNApSAcK_ID C00013679
NAME Chrysoeriol 7-(3'',6''-di-(E)-p-coumaroylglucoside);7-[[3,6-Bis-O-[(2E)-3-(4-hydroxyphenyl)-1-oxo-2-propenyl]-beta-D-glucopyranosyl]oxy]-5-hydroxy-2-(4-hydroxy-3-methoxyphenyl)-4H-1-benzopyran-4-one
CAS_RN 625409-67-8
FORMULA C40H34O15
EXACTMASS 754.1897704180001
AVERAGEMASS 754.68896
SMILES c(c1)c(ccc(C=CC(=O)OCC(O2)C(O)C(OC(C=Cc(c6)ccc(c6)O)=O)C(O)C2Oc(c5)cc(O)c(c53)C(C=C(c(c4)ccc(O)c4OC)O3)=O)1)O
M END