Mol:FL3FADCS0023
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 44 48 0 0 0 0 0 0 0 0999 V2000 | + | 44 48 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.0085 -0.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0085 -0.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7230 -0.4026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7230 -0.4026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4375 -0.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4375 -0.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4375 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4375 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7230 -2.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7230 -2.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0085 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0085 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1519 -0.4026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1519 -0.4026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8664 -0.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8664 -0.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8664 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8664 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1519 -2.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1519 -2.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5121 -0.4423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5121 -0.4423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1519 -2.7921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1519 -2.7921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7230 -2.6246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7230 -2.6246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7732 -0.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7732 -0.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4877 -0.7762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4877 -0.7762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2022 -0.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2022 -0.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2022 0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2022 0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4877 0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4877 0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7732 0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7732 0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8174 0.8165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8174 0.8165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8564 -0.7414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8564 -0.7414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.5121 -0.3628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.5121 -0.3628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3019 2.4267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3019 2.4267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9086 1.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9086 1.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5294 1.1343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5294 1.1343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5766 0.3104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5766 0.3104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.9698 0.8697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.9698 0.8697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.3490 1.6028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.3490 1.6028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.8119 1.8845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.8119 1.8845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4939 2.6066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4939 2.6066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8794 2.7921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8794 2.7921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9086 2.4582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9086 2.4582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.1914 1.1611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.1914 1.1611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8574 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8574 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.1962 0.8544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.1962 0.8544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4979 1.6226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4979 1.6226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3660 2.4372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3660 2.4372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.0273 1.9434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.0273 1.9434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7257 1.1752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7257 1.1752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.2889 0.9503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.2889 0.9503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5076 0.4412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5076 0.4412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.3207 -0.0625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.3207 -0.0625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4007 0.3784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4007 0.3784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.1578 1.5276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.1578 1.5276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 1 0 0 0 0 | + | 2 3 1 0 0 0 0 |
| − | 3 4 2 0 0 0 0 | + | 3 4 2 0 0 0 0 |
| − | 4 5 1 0 0 0 0 | + | 4 5 1 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 3 7 1 0 0 0 0 | + | 3 7 1 0 0 0 0 |
| − | 7 8 2 0 0 0 0 | + | 7 8 2 0 0 0 0 |
| − | 8 9 1 0 0 0 0 | + | 8 9 1 0 0 0 0 |
| − | 9 10 2 0 0 0 0 | + | 9 10 2 0 0 0 0 |
| − | 10 4 1 0 0 0 0 | + | 10 4 1 0 0 0 0 |
| − | 8 11 1 0 0 0 0 | + | 8 11 1 0 0 0 0 |
| − | 10 12 1 0 0 0 0 | + | 10 12 1 0 0 0 0 |
| − | 5 13 2 0 0 0 0 | + | 5 13 2 0 0 0 0 |
| − | 1 14 1 0 0 0 0 | + | 1 14 1 0 0 0 0 |
| − | 14 15 2 0 0 0 0 | + | 14 15 2 0 0 0 0 |
| − | 15 16 1 0 0 0 0 | + | 15 16 1 0 0 0 0 |
| − | 16 17 2 0 0 0 0 | + | 16 17 2 0 0 0 0 |
| − | 17 18 1 0 0 0 0 | + | 17 18 1 0 0 0 0 |
| − | 18 19 2 0 0 0 0 | + | 18 19 2 0 0 0 0 |
| − | 19 14 1 0 0 0 0 | + | 19 14 1 0 0 0 0 |
| − | 17 20 1 0 0 0 0 | + | 17 20 1 0 0 0 0 |
| − | 16 21 1 0 0 0 0 | + | 16 21 1 0 0 0 0 |
| − | 21 22 1 0 0 0 0 | + | 21 22 1 0 0 0 0 |
| − | 23 24 1 1 0 0 0 | + | 23 24 1 1 0 0 0 |
| − | 24 25 1 1 0 0 0 | + | 24 25 1 1 0 0 0 |
| − | 26 25 1 1 0 0 0 | + | 26 25 1 1 0 0 0 |
| − | 26 27 1 0 0 0 0 | + | 26 27 1 0 0 0 0 |
| − | 27 28 1 0 0 0 0 | + | 27 28 1 0 0 0 0 |
| − | 28 23 1 0 0 0 0 | + | 28 23 1 0 0 0 0 |
| − | 28 29 1 0 0 0 0 | + | 28 29 1 0 0 0 0 |
| − | 29 30 1 0 0 0 0 | + | 29 30 1 0 0 0 0 |
| − | 23 31 1 0 0 0 0 | + | 23 31 1 0 0 0 0 |
| − | 24 32 1 0 0 0 0 | + | 24 32 1 0 0 0 0 |
| − | 25 33 1 0 0 0 0 | + | 25 33 1 0 0 0 0 |
| − | 7 26 1 0 0 0 0 | + | 7 26 1 0 0 0 0 |
| − | 34 35 1 1 0 0 0 | + | 34 35 1 1 0 0 0 |
| − | 35 36 1 1 0 0 0 | + | 35 36 1 1 0 0 0 |
| − | 37 36 1 1 0 0 0 | + | 37 36 1 1 0 0 0 |
| − | 37 38 1 0 0 0 0 | + | 37 38 1 0 0 0 0 |
| − | 38 39 1 0 0 0 0 | + | 38 39 1 0 0 0 0 |
| − | 39 34 1 0 0 0 0 | + | 39 34 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 34 42 1 0 0 0 0 | + | 34 42 1 0 0 0 0 |
| − | 35 43 1 0 0 0 0 | + | 35 43 1 0 0 0 0 |
| − | 36 44 1 0 0 0 0 | + | 36 44 1 0 0 0 0 |
| − | 37 30 1 0 0 0 0 | + | 37 30 1 0 0 0 0 |
| − | S SKP 8 | + | S SKP 8 |
| − | ID FL3FADCS0023 | + | ID FL3FADCS0023 |
| − | KNApSAcK_ID C00014068 | + | KNApSAcK_ID C00014068 |
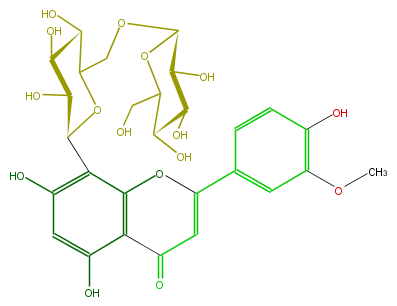
| − | NAME Scoparin 6''-O-glucoside | + | NAME Scoparin 6''-O-glucoside |
| − | CAS_RN 605652-87-7 | + | CAS_RN 605652-87-7 |
| − | FORMULA C28H32O16 | + | FORMULA C28H32O16 |
| − | EXACTMASS 624.1690349759999 | + | EXACTMASS 624.1690349759999 |
| − | AVERAGEMASS 624.54408 | + | AVERAGEMASS 624.54408 |
| − | SMILES OC(C1c(c34)c(cc(c3C(=O)C=C(c(c5)ccc(c5OC)O)O4)O)O)C(C(O)C(COC(O2)C(O)C(C(O)C(CO)2)O)O1)O | + | SMILES OC(C1c(c34)c(cc(c3C(=O)C=C(c(c5)ccc(c5OC)O)O4)O)O)C(C(O)C(COC(O2)C(O)C(C(O)C(CO)2)O)O1)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
44 48 0 0 0 0 0 0 0 0999 V2000
-0.0085 -0.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7230 -0.4026 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4375 -0.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4375 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7230 -2.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0085 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1519 -0.4026 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8664 -0.8151 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.8664 -1.6401 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1519 -2.0526 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5121 -0.4423 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1519 -2.7921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7230 -2.6246 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.7732 -0.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4877 -0.7762 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2022 -0.3637 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2022 0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4877 0.8738 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7732 0.4613 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8174 0.8165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.8564 -0.7414 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.5121 -0.3628 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.3019 2.4267 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.9086 1.8673 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5294 1.1343 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5766 0.3104 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.9698 0.8697 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.3490 1.6028 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.8119 1.8845 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4939 2.6066 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8794 2.7921 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9086 2.4582 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.1914 1.1611 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8574 0.3605 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.1962 0.8544 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4979 1.6226 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.3660 2.4372 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.0273 1.9434 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7257 1.1752 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.2889 0.9503 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5076 0.4412 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.3207 -0.0625 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4007 0.3784 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.1578 1.5276 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 1 0 0 0 0
3 4 2 0 0 0 0
4 5 1 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
3 7 1 0 0 0 0
7 8 2 0 0 0 0
8 9 1 0 0 0 0
9 10 2 0 0 0 0
10 4 1 0 0 0 0
8 11 1 0 0 0 0
10 12 1 0 0 0 0
5 13 2 0 0 0 0
1 14 1 0 0 0 0
14 15 2 0 0 0 0
15 16 1 0 0 0 0
16 17 2 0 0 0 0
17 18 1 0 0 0 0
18 19 2 0 0 0 0
19 14 1 0 0 0 0
17 20 1 0 0 0 0
16 21 1 0 0 0 0
21 22 1 0 0 0 0
23 24 1 1 0 0 0
24 25 1 1 0 0 0
26 25 1 1 0 0 0
26 27 1 0 0 0 0
27 28 1 0 0 0 0
28 23 1 0 0 0 0
28 29 1 0 0 0 0
29 30 1 0 0 0 0
23 31 1 0 0 0 0
24 32 1 0 0 0 0
25 33 1 0 0 0 0
7 26 1 0 0 0 0
34 35 1 1 0 0 0
35 36 1 1 0 0 0
37 36 1 1 0 0 0
37 38 1 0 0 0 0
38 39 1 0 0 0 0
39 34 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
34 42 1 0 0 0 0
35 43 1 0 0 0 0
36 44 1 0 0 0 0
37 30 1 0 0 0 0
S SKP 8
ID FL3FADCS0023
KNApSAcK_ID C00014068
NAME Scoparin 6''-O-glucoside
CAS_RN 605652-87-7
FORMULA C28H32O16
EXACTMASS 624.1690349759999
AVERAGEMASS 624.54408
SMILES OC(C1c(c34)c(cc(c3C(=O)C=C(c(c5)ccc(c5OC)O)O4)O)O)C(C(O)C(COC(O2)C(O)C(C(O)C(CO)2)O)O1)O
M END