Mol:FL3FADCS0016
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 57 62 0 0 0 0 0 0 0 0999 V2000 | + | 57 62 0 0 0 0 0 0 0 0999 V2000 |
| − | -0.0279 2.2427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0279 2.2427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0279 1.4176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0279 1.4176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6866 1.0052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6866 1.0052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4011 1.4176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4011 1.4176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.4011 2.2427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.4011 2.2427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6866 2.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6866 2.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1156 1.0052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1156 1.0052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8302 1.4176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8302 1.4176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.8302 2.2427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.8302 2.2427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1156 2.6553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1156 2.6553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.1156 0.3618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.1156 0.3618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.6866 0.1804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.6866 0.1804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7033 2.7876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7033 2.7876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4563 2.3528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4563 2.3528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2093 2.7876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2093 2.7876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2093 3.6570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2093 3.6570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.4563 4.0916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.4563 4.0916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7033 3.6570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7033 3.6570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.0634 4.1502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.0634 4.1502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7630 2.6671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7630 2.6671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9126 1.6134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9126 1.6134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.2503 0.8716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.2503 0.8716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5880 1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5880 1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6874 1.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6874 1.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3232 1.7458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3232 1.7458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0383 1.4015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0383 1.4015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.5700 1.2338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.5700 1.2338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.0104 0.8716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.0104 0.8716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.8636 0.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.8636 0.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4880 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4880 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1985 -0.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1985 -0.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.9731 -0.8604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.9731 -0.8604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1985 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1985 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4880 -2.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4880 -2.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7134 -1.2754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7134 -1.2754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.4880 0.3263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.4880 0.3263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.2369 -1.7520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.2369 -1.7520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6229 -2.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6229 -2.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1111 -2.4402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1111 -2.4402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4764 -3.0764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4764 -3.0764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0974 -3.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0974 -3.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.6809 -3.0980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.6809 -3.0980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2636 -3.4344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2636 -3.4344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8464 -3.0980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8464 -3.0980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.0974 -4.1502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.0974 -4.1502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8464 -2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8464 -2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5452 -1.8876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5452 -1.8876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2441 -2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2441 -2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2441 -3.0980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2441 -3.0980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5452 -3.5015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5452 -3.5015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.9415 -1.8884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.9415 -1.8884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 6.3216 2.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 6.3216 2.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 7.4340 1.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 7.4340 1.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.5649 1.9280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.5649 1.9280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7489 2.9714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7489 2.9714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -6.1618 -2.9709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -6.1618 -2.9709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -7.4340 -2.7946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -7.4340 -2.7946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 3 12 1 0 0 0 0 | + | 3 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 9 13 1 0 0 0 0 | + | 9 13 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 1 20 1 0 0 0 0 | + | 1 20 1 0 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 22 23 1 1 0 0 0 | + | 22 23 1 1 0 0 0 |
| − | 24 23 1 1 0 0 0 | + | 24 23 1 1 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 26 1 0 0 0 0 | + | 25 26 1 0 0 0 0 |
| − | 26 21 1 0 0 0 0 | + | 26 21 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 23 29 1 0 0 0 0 | + | 23 29 1 0 0 0 0 |
| − | 24 2 1 0 0 0 0 | + | 24 2 1 0 0 0 0 |
| − | 30 31 1 0 0 0 0 | + | 30 31 1 0 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 32 33 1 1 0 0 0 | + | 32 33 1 1 0 0 0 |
| − | 34 33 1 1 0 0 0 | + | 34 33 1 1 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 35 37 1 0 0 0 0 | + | 35 37 1 0 0 0 0 |
| − | 34 38 1 0 0 0 0 | + | 34 38 1 0 0 0 0 |
| − | 33 39 1 0 0 0 0 | + | 33 39 1 0 0 0 0 |
| − | 31 29 1 0 0 0 0 | + | 31 29 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 40 41 1 0 0 0 0 | + | 40 41 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 42 43 2 0 0 0 0 | + | 42 43 2 0 0 0 0 |
| − | 43 44 1 0 0 0 0 | + | 43 44 1 0 0 0 0 |
| − | 41 45 2 0 0 0 0 | + | 41 45 2 0 0 0 0 |
| − | 44 46 2 0 0 0 0 | + | 44 46 2 0 0 0 0 |
| − | 46 47 1 0 0 0 0 | + | 46 47 1 0 0 0 0 |
| − | 47 48 2 0 0 0 0 | + | 47 48 2 0 0 0 0 |
| − | 48 49 1 0 0 0 0 | + | 48 49 1 0 0 0 0 |
| − | 49 50 2 0 0 0 0 | + | 49 50 2 0 0 0 0 |
| − | 50 44 1 0 0 0 0 | + | 50 44 1 0 0 0 0 |
| − | 48 51 1 0 0 0 0 | + | 48 51 1 0 0 0 0 |
| − | 52 53 1 0 0 0 0 | + | 52 53 1 0 0 0 0 |
| − | 15 52 1 0 0 0 0 | + | 15 52 1 0 0 0 0 |
| − | 54 55 1 0 0 0 0 | + | 54 55 1 0 0 0 0 |
| − | 26 54 1 0 0 0 0 | + | 26 54 1 0 0 0 0 |
| − | 56 57 1 0 0 0 0 | + | 56 57 1 0 0 0 0 |
| − | 49 56 1 0 0 0 0 | + | 49 56 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 52 53 | + | M SAL 1 2 52 53 |
| − | M SBL 1 1 58 | + | M SBL 1 1 58 |
| − | M SMT 1 OCH3 | + | M SMT 1 OCH3 |
| − | M SBV 1 58 -1.1123 0.6422 | + | M SBV 1 58 -1.1123 0.6422 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 54 55 | + | M SAL 2 2 54 55 |
| − | M SBL 2 1 60 | + | M SBL 2 1 60 |
| − | M SMT 2 ^ CH2OH | + | M SMT 2 ^ CH2OH |
| − | M SBV 2 60 0.5265 -0.5265 | + | M SBV 2 60 0.5265 -0.5265 |
| − | M STY 1 3 SUP | + | M STY 1 3 SUP |
| − | M SLB 1 3 3 | + | M SLB 1 3 3 |
| − | M SAL 3 2 56 57 | + | M SAL 3 2 56 57 |
| − | M SBL 3 1 62 | + | M SBL 3 1 62 |
| − | M SMT 3 ^ OCH3 | + | M SMT 3 ^ OCH3 |
| − | M SBV 3 62 0.9177 -0.1272 | + | M SBV 3 62 0.9177 -0.1272 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FADCS0016 | + | ID FL3FADCS0016 |
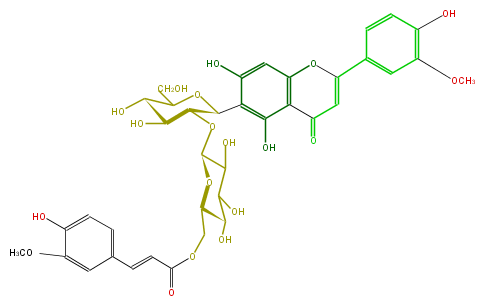
| − | FORMULA C38H40O19 | + | FORMULA C38H40O19 |
| − | EXACTMASS 800.216379098 | + | EXACTMASS 800.216379098 |
| − | AVERAGEMASS 800.7128 | + | AVERAGEMASS 800.7128 |
| − | SMILES C(c(c6)ccc(c(OC)6)O)=CC(=O)OCC(C(O)1)OC(OC(C2c(c5O)c(cc(c45)OC(=CC4=O)c(c3)ccc(O)c3OC)O)C(O)C(C(O2)CO)O)C(O)C1O | + | SMILES C(c(c6)ccc(c(OC)6)O)=CC(=O)OCC(C(O)1)OC(OC(C2c(c5O)c(cc(c45)OC(=CC4=O)c(c3)ccc(O)c3OC)O)C(O)C(C(O2)CO)O)C(O)C1O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
57 62 0 0 0 0 0 0 0 0999 V2000
-0.0279 2.2427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0279 1.4176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6866 1.0052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4011 1.4176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.4011 2.2427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.6866 2.6553 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1156 1.0052 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8302 1.4176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.8302 2.2427 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.1156 2.6553 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
2.1156 0.3618 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
0.6866 0.1804 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
3.7033 2.7876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4563 2.3528 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2093 2.7876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
5.2093 3.6570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
4.4563 4.0916 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7033 3.6570 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
6.0634 4.1502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.7630 2.6671 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9126 1.6134 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.2503 0.8716 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.5880 1.2690 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6874 1.1631 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.3232 1.7458 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0383 1.4015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.5700 1.2338 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.0104 0.8716 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.8636 0.7980 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.4880 -0.4817 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1985 -0.0714 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.9731 -0.8604 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1985 -1.6541 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4880 -2.0644 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7134 -1.2754 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.4880 0.3263 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.2369 -1.7520 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.6229 -2.5678 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.1111 -2.4402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4764 -3.0764 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.0974 -3.4349 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.6809 -3.0980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2636 -3.4344 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.8464 -3.0980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.0974 -4.1502 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8464 -2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5452 -1.8876 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2441 -2.2910 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2441 -3.0980 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.5452 -3.5015 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.9415 -1.8884 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
6.3216 2.1454 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
7.4340 1.5033 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.5649 1.9280 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7489 2.9714 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-6.1618 -2.9709 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-7.4340 -2.7946 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
3 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
9 13 1 0 0 0 0
16 19 1 0 0 0 0
1 20 1 0 0 0 0
21 22 1 1 0 0 0
22 23 1 1 0 0 0
24 23 1 1 0 0 0
24 25 1 0 0 0 0
25 26 1 0 0 0 0
26 21 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
23 29 1 0 0 0 0
24 2 1 0 0 0 0
30 31 1 0 0 0 0
31 32 1 1 0 0 0
32 33 1 1 0 0 0
34 33 1 1 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
35 37 1 0 0 0 0
34 38 1 0 0 0 0
33 39 1 0 0 0 0
31 29 1 0 0 0 0
39 40 1 0 0 0 0
40 41 1 0 0 0 0
41 42 1 0 0 0 0
42 43 2 0 0 0 0
43 44 1 0 0 0 0
41 45 2 0 0 0 0
44 46 2 0 0 0 0
46 47 1 0 0 0 0
47 48 2 0 0 0 0
48 49 1 0 0 0 0
49 50 2 0 0 0 0
50 44 1 0 0 0 0
48 51 1 0 0 0 0
52 53 1 0 0 0 0
15 52 1 0 0 0 0
54 55 1 0 0 0 0
26 54 1 0 0 0 0
56 57 1 0 0 0 0
49 56 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 52 53
M SBL 1 1 58
M SMT 1 OCH3
M SBV 1 58 -1.1123 0.6422
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 54 55
M SBL 2 1 60
M SMT 2 ^ CH2OH
M SBV 2 60 0.5265 -0.5265
M STY 1 3 SUP
M SLB 1 3 3
M SAL 3 2 56 57
M SBL 3 1 62
M SMT 3 ^ OCH3
M SBV 3 62 0.9177 -0.1272
S SKP 5
ID FL3FADCS0016
FORMULA C38H40O19
EXACTMASS 800.216379098
AVERAGEMASS 800.7128
SMILES C(c(c6)ccc(c(OC)6)O)=CC(=O)OCC(C(O)1)OC(OC(C2c(c5O)c(cc(c45)OC(=CC4=O)c(c3)ccc(O)c3OC)O)C(O)C(C(O2)CO)O)C(O)C1O
M END