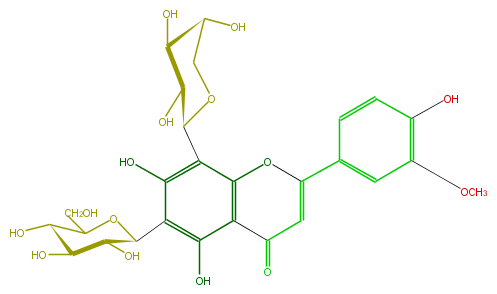
Mol:FL3FADCS0006
From Metabolomics.JP
(Difference between revisions)
| Line 1: | Line 1: | ||
| − | + | ||
| − | + | ||
| − | Copyright: ARM project http://www.metabolome.jp/ | + | Copyright: ARM project http://www.metabolome.jp/ |
| − | 42 46 0 0 0 0 0 0 0 0999 V2000 | + | 42 46 0 0 0 0 0 0 0 0999 V2000 |
| − | -2.1481 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1481 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1481 -1.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1481 -1.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4336 -1.9692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4336 -1.9692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7191 -1.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7191 -1.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.7191 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.7191 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4336 -0.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4336 -0.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0047 -1.9692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0047 -1.9692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7098 -1.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7098 -1.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 0.7098 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 0.7098 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0047 -0.3193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0047 -0.3193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.0047 -2.6125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.0047 -2.6125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.8623 -0.3194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.8623 -0.3194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5035 -0.2930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5035 -0.2930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2563 -0.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2563 -0.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0092 -0.2930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0092 -0.2930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.0092 0.5764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.0092 0.5764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 2.2563 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 2.2563 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1.5035 0.5764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 1.5035 0.5764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 3.7617 1.0109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 3.7617 1.0109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.3305 2.6779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.3305 2.6779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1083 2.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1083 2.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7784 1.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7784 1.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.7695 0.4215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.7695 0.4215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.1745 1.0165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.1745 1.0165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.5662 1.7587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.5662 1.7587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -0.6871 2.5055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -0.6871 2.5055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1083 2.7939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1083 2.7939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.1917 0.5243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.1917 0.5243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -1.4336 -2.7939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -1.4336 -2.7939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.5739 -1.6269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.5739 -1.6269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.0972 -2.2562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.0972 -2.2562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.4107 -1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.4107 -1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.7483 -1.9820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.7483 -1.9820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.2297 -1.5007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.2297 -1.5007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -3.8302 -1.8176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -3.8302 -1.8176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.1795 -1.7892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.1795 -1.7892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.7059 -2.2562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.7059 -2.2562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -2.9180 -2.2737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -2.9180 -2.2737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -4.2733 -1.3745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | -4.2733 -1.3745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | -5.2336 -0.9267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | -5.2336 -0.9267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 4.1214 -0.9351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 | + | 4.1214 -0.9351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 5.2336 -1.5774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 | + | 5.2336 -1.5774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0 |
| − | 1 2 1 0 0 0 0 | + | 1 2 1 0 0 0 0 |
| − | 2 3 2 0 0 0 0 | + | 2 3 2 0 0 0 0 |
| − | 3 4 1 0 0 0 0 | + | 3 4 1 0 0 0 0 |
| − | 4 5 2 0 0 0 0 | + | 4 5 2 0 0 0 0 |
| − | 5 6 1 0 0 0 0 | + | 5 6 1 0 0 0 0 |
| − | 6 1 2 0 0 0 0 | + | 6 1 2 0 0 0 0 |
| − | 4 7 1 0 0 0 0 | + | 4 7 1 0 0 0 0 |
| − | 7 8 1 0 0 0 0 | + | 7 8 1 0 0 0 0 |
| − | 8 9 2 0 0 0 0 | + | 8 9 2 0 0 0 0 |
| − | 9 10 1 0 0 0 0 | + | 9 10 1 0 0 0 0 |
| − | 10 5 1 0 0 0 0 | + | 10 5 1 0 0 0 0 |
| − | 7 11 2 0 0 0 0 | + | 7 11 2 0 0 0 0 |
| − | 1 12 1 0 0 0 0 | + | 1 12 1 0 0 0 0 |
| − | 13 14 2 0 0 0 0 | + | 13 14 2 0 0 0 0 |
| − | 14 15 1 0 0 0 0 | + | 14 15 1 0 0 0 0 |
| − | 15 16 2 0 0 0 0 | + | 15 16 2 0 0 0 0 |
| − | 16 17 1 0 0 0 0 | + | 16 17 1 0 0 0 0 |
| − | 17 18 2 0 0 0 0 | + | 17 18 2 0 0 0 0 |
| − | 18 13 1 0 0 0 0 | + | 18 13 1 0 0 0 0 |
| − | 13 9 1 0 0 0 0 | + | 13 9 1 0 0 0 0 |
| − | 16 19 1 0 0 0 0 | + | 16 19 1 0 0 0 0 |
| − | 20 21 1 1 0 0 0 | + | 20 21 1 1 0 0 0 |
| − | 21 22 1 1 0 0 0 | + | 21 22 1 1 0 0 0 |
| − | 23 22 1 1 0 0 0 | + | 23 22 1 1 0 0 0 |
| − | 23 24 1 0 0 0 0 | + | 23 24 1 0 0 0 0 |
| − | 24 25 1 0 0 0 0 | + | 24 25 1 0 0 0 0 |
| − | 25 20 1 0 0 0 0 | + | 25 20 1 0 0 0 0 |
| − | 20 26 1 0 0 0 0 | + | 20 26 1 0 0 0 0 |
| − | 21 27 1 0 0 0 0 | + | 21 27 1 0 0 0 0 |
| − | 22 28 1 0 0 0 0 | + | 22 28 1 0 0 0 0 |
| − | 6 23 1 0 0 0 0 | + | 6 23 1 0 0 0 0 |
| − | 3 29 1 0 0 0 0 | + | 3 29 1 0 0 0 0 |
| − | 30 31 1 1 0 0 0 | + | 30 31 1 1 0 0 0 |
| − | 31 32 1 1 0 0 0 | + | 31 32 1 1 0 0 0 |
| − | 33 32 1 1 0 0 0 | + | 33 32 1 1 0 0 0 |
| − | 33 34 1 0 0 0 0 | + | 33 34 1 0 0 0 0 |
| − | 34 35 1 0 0 0 0 | + | 34 35 1 0 0 0 0 |
| − | 35 30 1 0 0 0 0 | + | 35 30 1 0 0 0 0 |
| − | 30 36 1 0 0 0 0 | + | 30 36 1 0 0 0 0 |
| − | 31 37 1 0 0 0 0 | + | 31 37 1 0 0 0 0 |
| − | 32 38 1 0 0 0 0 | + | 32 38 1 0 0 0 0 |
| − | 33 2 1 0 0 0 0 | + | 33 2 1 0 0 0 0 |
| − | 39 40 1 0 0 0 0 | + | 39 40 1 0 0 0 0 |
| − | 35 39 1 0 0 0 0 | + | 35 39 1 0 0 0 0 |
| − | 41 42 1 0 0 0 0 | + | 41 42 1 0 0 0 0 |
| − | 15 41 1 0 0 0 0 | + | 15 41 1 0 0 0 0 |
| − | M STY 1 1 SUP | + | M STY 1 1 SUP |
| − | M SLB 1 1 1 | + | M SLB 1 1 1 |
| − | M SAL 1 2 39 40 | + | M SAL 1 2 39 40 |
| − | M SBL 1 1 44 | + | M SBL 1 1 44 |
| − | M SMT 1 ^ CH2OH | + | M SMT 1 ^ CH2OH |
| − | M SBV 1 44 0.4431 -0.4431 | + | M SBV 1 44 0.4431 -0.4431 |
| − | M STY 1 2 SUP | + | M STY 1 2 SUP |
| − | M SLB 1 2 2 | + | M SLB 1 2 2 |
| − | M SAL 2 2 41 42 | + | M SAL 2 2 41 42 |
| − | M SBL 2 1 46 | + | M SBL 2 1 46 |
| − | M SMT 2 OCH3 | + | M SMT 2 OCH3 |
| − | M SBV 2 46 -1.1122 0.6422 | + | M SBV 2 46 -1.1122 0.6422 |
| − | S SKP 5 | + | S SKP 5 |
| − | ID FL3FADCS0006 | + | ID FL3FADCS0006 |
| − | FORMULA C27H30O15 | + | FORMULA C27H30O15 |
| − | EXACTMASS 594.15847029 | + | EXACTMASS 594.15847029 |
| − | AVERAGEMASS 594.5181 | + | AVERAGEMASS 594.5181 |
| − | SMILES C(C(C1O)OC(c(c4O)c(c(c(c4C(O5)C(O)C(O)C(O)C5)2)C(=O)C=C(c(c3)cc(OC)c(O)c3)O2)O)C(C1O)O)O | + | SMILES C(C(C1O)OC(c(c4O)c(c(c(c4C(O5)C(O)C(O)C(O)C5)2)C(=O)C=C(c(c3)cc(OC)c(O)c3)O2)O)C(C1O)O)O |
M END | M END | ||
| − | |||
Latest revision as of 09:00, 14 March 2009
Copyright: ARM project http://www.metabolome.jp/
42 46 0 0 0 0 0 0 0 0999 V2000
-2.1481 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1481 -1.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4336 -1.9692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7191 -1.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.7191 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.4336 -0.3193 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0047 -1.9692 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7098 -1.5567 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
0.7098 -0.7318 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.0047 -0.3193 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-0.0047 -2.6125 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.8623 -0.3194 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
1.5035 -0.2930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2563 -0.7276 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0092 -0.2930 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.0092 0.5764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
2.2563 1.0111 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1.5035 0.5764 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
3.7617 1.0109 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.3305 2.6779 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.1083 2.0886 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7784 1.2402 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.7695 0.4215 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-1.1745 1.0165 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.5662 1.7587 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-0.6871 2.5055 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1083 2.7939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.1917 0.5243 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-1.4336 -2.7939 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.5739 -1.6269 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-4.0972 -2.2562 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.4107 -1.9892 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-2.7483 -1.9820 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-3.2297 -1.5007 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-3.8302 -1.8176 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.1795 -1.7892 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.7059 -2.2562 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-2.9180 -2.2737 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
-4.2733 -1.3745 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
-5.2336 -0.9267 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
4.1214 -0.9351 0.0000 O 0 0 0 0 0 0 0 0 0 0 0 0
5.2336 -1.5774 0.0000 C 0 0 0 0 0 0 0 0 0 0 0 0
1 2 1 0 0 0 0
2 3 2 0 0 0 0
3 4 1 0 0 0 0
4 5 2 0 0 0 0
5 6 1 0 0 0 0
6 1 2 0 0 0 0
4 7 1 0 0 0 0
7 8 1 0 0 0 0
8 9 2 0 0 0 0
9 10 1 0 0 0 0
10 5 1 0 0 0 0
7 11 2 0 0 0 0
1 12 1 0 0 0 0
13 14 2 0 0 0 0
14 15 1 0 0 0 0
15 16 2 0 0 0 0
16 17 1 0 0 0 0
17 18 2 0 0 0 0
18 13 1 0 0 0 0
13 9 1 0 0 0 0
16 19 1 0 0 0 0
20 21 1 1 0 0 0
21 22 1 1 0 0 0
23 22 1 1 0 0 0
23 24 1 0 0 0 0
24 25 1 0 0 0 0
25 20 1 0 0 0 0
20 26 1 0 0 0 0
21 27 1 0 0 0 0
22 28 1 0 0 0 0
6 23 1 0 0 0 0
3 29 1 0 0 0 0
30 31 1 1 0 0 0
31 32 1 1 0 0 0
33 32 1 1 0 0 0
33 34 1 0 0 0 0
34 35 1 0 0 0 0
35 30 1 0 0 0 0
30 36 1 0 0 0 0
31 37 1 0 0 0 0
32 38 1 0 0 0 0
33 2 1 0 0 0 0
39 40 1 0 0 0 0
35 39 1 0 0 0 0
41 42 1 0 0 0 0
15 41 1 0 0 0 0
M STY 1 1 SUP
M SLB 1 1 1
M SAL 1 2 39 40
M SBL 1 1 44
M SMT 1 ^ CH2OH
M SBV 1 44 0.4431 -0.4431
M STY 1 2 SUP
M SLB 1 2 2
M SAL 2 2 41 42
M SBL 2 1 46
M SMT 2 OCH3
M SBV 2 46 -1.1122 0.6422
S SKP 5
ID FL3FADCS0006
FORMULA C27H30O15
EXACTMASS 594.15847029
AVERAGEMASS 594.5181
SMILES C(C(C1O)OC(c(c4O)c(c(c(c4C(O5)C(O)C(O)C(O)C5)2)C(=O)C=C(c(c3)cc(OC)c(O)c3)O2)O)C(C1O)O)O
M END